用遗传算法(GA)优化支持向量机(SVM)解决分类与回归预测问题(MATLAB实现)
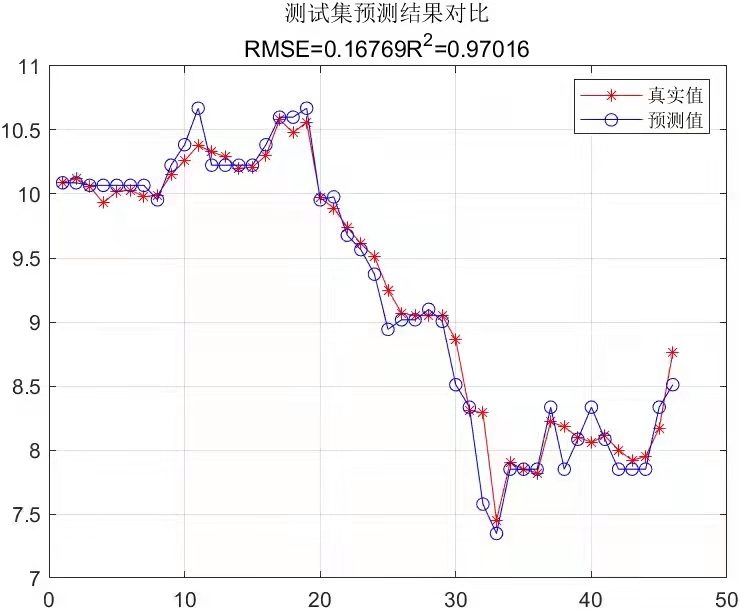
通过上面的代码示例,大家应该能很清晰地看到如何用遗传算法优化 SVM 的c和g参数来解决分类问题啦。如果是回归问题,只需要把svmtrain和svmpredict函数换成回归版本的函数,同时调整适应度函数,比如可以用均方误差(MSE)等回归评估指标作为适应度。只要按照这个框架,把数据换成你自己的,就能轻松上手解决实际的分类或回归预测问题。希望大家都能在自己的项目里用上这个方法,提升模型性能。怎么样
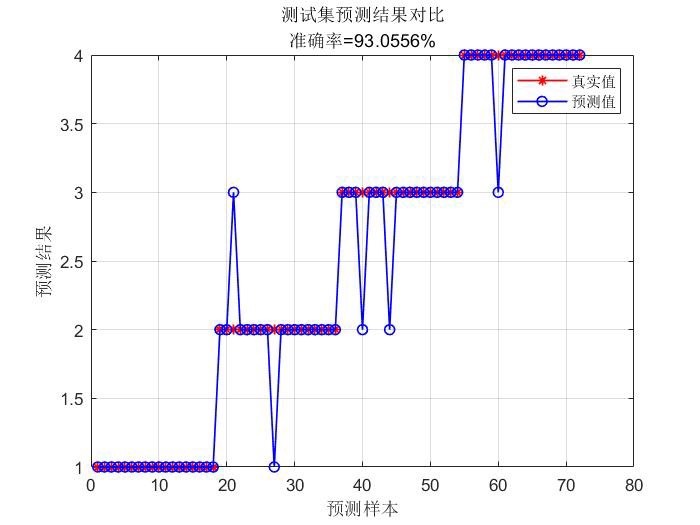
遗传算法ga优化支持向量机SVM回归或分类程序(MATLAB),优化svm两个参数c,g,解决分类或回归预测问题。 有例子,易上手,只要换数据就行,保证正常运行,
在机器学习领域,支持向量机(SVM)是一种非常强大的分类和回归算法。然而,SVM 的性能很大程度上依赖于其两个关键参数:惩罚参数 c 和核函数参数 g。如何选择最优的 c 和 g 值成为了提升 SVM 性能的关键。这时候,遗传算法(GA)就派上用场啦。今天咱就来唠唠怎么用 GA 优化 SVM 来解决分类或回归预测问题,而且有超简单易上手的例子,换个数据就能跑起来。
遗传算法优化 SVM 的原理
遗传算法是模拟生物在自然环境中的遗传和进化过程而形成的一种自适应全局优化概率搜索算法。它通过模拟自然选择和遗传机制,从一组初始解(种群)开始,逐步进化出更优的解。在优化 SVM 的 c 和 g 参数时,我们把每一组可能的 c 和 g 组合看成一个个体,整个种群就是这些个体的集合。然后通过遗传算法的选择、交叉、变异等操作,让种群朝着适应度更高(也就是 SVM 性能更好)的方向进化。
MATLAB 代码实现
数据准备
假设我们有一组简单的二维数据用于分类任务,先来看数据生成部分代码:
% 生成训练数据
data = [1 1; 1 2; 2 1; 2 2; 3 3; 3 4; 4 3; 4 4];
labels = [1; 1; 1; 1; -1; -1; -1; -1];这里我们创建了一个简单的 8 个样本的数据集,前 4 个样本标签为 1,后 4 个样本标签为 -1。实际应用中,你可以把这里的数据替换成你自己的真实数据。
遗传算法参数设置
% 遗传算法参数设置
NIND = 40; % 种群个体数目
MAXGEN = 20; % 最大遗传代数
PRECI = 20; % 变量的二进制位数
GGAP = 0.9; % 代沟
trace = zeros(2, MAXGEN); % 寻优结果的初始值
FieldD = [PRECI; log10([0.01 100]); PRECI; log10([0.01 100]); 1; 0; 1]; % 区域描述器
Chrom = crtbp(NIND, PRECI * 2); % 初始种群解释一下,NIND 定义了我们初始种群里有多少个个体,这里设置为 40。MAXGEN 是最大遗传代数,意味着算法最多迭代 20 次。PRECI 是把 c 和 g 参数用二进制编码时的位数。GGAP 代沟决定了每一代有多少比例的个体能遗传到下一代。FieldD 是用来描述参数范围的,这里 c 和 g 的范围都设置在 0.01 到 100 之间。Chrom 则生成了初始种群。
适应度函数
function fitness = svm_fitness(individual)
global data labels
c = 10 ^ (bs2rv(individual(1:20), FieldD(1:2)));
g = 10 ^ (bs2rv(individual(21:40), FieldD(3:4)));
model = svmtrain(labels, data, ['-c ', num2str(c),' -g ', num2str(g)]);
[predict_label, accuracy, ~] = svmpredict(labels, data, model);
fitness = accuracy(1);
end这个函数是遗传算法里非常关键的适应度函数。它接收一个个体(也就是一组 c 和 g 的二进制编码),先把二进制编码转回实际的 c 和 g 值。然后用 svmtrain 函数训练 SVM 模型,再用 svmpredict 对训练数据进行预测,把预测准确率作为适应度返回。这里要注意,因为是简单示例,直接在训练集上评估准确率了,实际应用建议用交叉验证等更合理的方式评估。
遗传算法主循环
gen = 0;
while gen < MAXGEN
FitnV = ranking(-ObjV); % 分配适应度值
SelCh = select('sus', Chrom, FitnV, GGAP); % 选择
SelCh = recombin('xovsp', SelCh, 0.7); % 重组
SelCh = mut(SelCh); % 变异
ObjVSel = objfun(SelCh); % 计算子代的目标函数值
[Chrom, ObjV] = reins(Chrom, SelCh, 1, 1, ObjV, ObjVSel); % 重插入子代到父代,得到新种群
[bestfitness, bestindex] = max(ObjV);
trace(1, gen + 1) = bestfitness;
trace(2, gen + 1) = sum(ObjV) / length(ObjV);
gen = gen + 1;
end这里就是遗传算法的主循环啦。每一代都先计算适应度,然后进行选择、交叉(重组)、变异操作,生成子代。再把子代和父代合并,根据适应度选择留下哪些个体形成新的种群。同时记录每一代的最优适应度和平均适应度,方便后续查看算法收敛情况。
最终结果与预测
bestindividual = Chrom(bestindex, :);
bestc = 10 ^ (bs2rv(bestindividual(1:20), FieldD(1:2)));
bestg = 10 ^ (bs2rv(bestindividual(21:40), FieldD(3:4)));
bestmodel = svmtrain(labels, data, ['-c ', num2str(bestc),' -g ', num2str(g)]);
[final_predict_label, final_accuracy, ~] = svmpredict(labels, data, bestmodel);
disp(['最优 c 值: ', num2str(bestc)]);
disp(['最优 g 值: ', num2str(bestg)]);
disp(['最终预测准确率: ', num2str(final_accuracy(1))]);最后找到最优个体,得到最优的 c 和 g 值,用这组最优参数训练最终的 SVM 模型,并对数据进行预测,输出最优参数和最终预测准确率。
总结
通过上面的代码示例,大家应该能很清晰地看到如何用遗传算法优化 SVM 的 c 和 g 参数来解决分类问题啦。如果是回归问题,只需要把 svmtrain 和 svmpredict 函数换成回归版本的函数,同时调整适应度函数,比如可以用均方误差(MSE)等回归评估指标作为适应度。只要按照这个框架,把数据换成你自己的,就能轻松上手解决实际的分类或回归预测问题。希望大家都能在自己的项目里用上这个方法,提升模型性能。
怎么样,是不是感觉遗传算法优化 SVM 也没那么难呀?赶紧动手试试吧!
更多推荐
 已为社区贡献17条内容
已为社区贡献17条内容








所有评论(0)