国内生信云平台部署全攻略:阿里云 / 华为云组学分析本地化适配(含批量任务调度避坑)
部署架构控制节点:部署 Airflow WebServer 与元数据库(RDS / 云数据库)。工作节点:部署 Celery Worker,弹性扩容至 100 + 节点。任务 DAG 定义python# 质控任务# 比对任务# 定量任务# 任务依赖阿里云与华为云在生信部署领域各有侧重:阿里云凭借成熟的基因分析平台和丰富的工具生态,更适合超大规模临床研究与快速部署需求;华为云则以鲲鹏 ARM 架构、
一、生信云平台部署的核心价值与行业痛点
随着基因测序技术的迭代,生信数据正以 “PB 级 / 年” 的速度爆炸式增长 —— 单个人类 30X 全基因组测序数据量达 100GB,一个千人队列项目数据量可轻松突破 100TB。传统本地服务器架构面临三重核心瓶颈:一是资源弹性不足,高峰时计算能力短缺、低谷时设备闲置;二是存储管理复杂,冷热数据分层存储成本高企;三是流程标准化困难,工具版本混乱导致结果不可复现。
云计算的出现为这些痛点提供了系统性解决方案。华大基因通过阿里云实现 1000 例人类全外显子组数据 22 小时完成分析,较传统方案提速 10 倍以上;基于华为云鲲鹏架构的水稻 WGS 分析流程,可将 36.98X 深度样本的分析时间压缩至 8 分钟。但生信工具的本地化适配、批量任务的高效调度仍是实践中的主要障碍,这也是本攻略的核心聚焦点。
二、阿里云与华为云生信核心能力全景对比
选择合适的云平台是部署成功的前提。阿里云与华为云均具备完善的生信支撑体系,但在产品矩阵、架构优化和适配场景上存在显著差异,具体对比如下:
| 维度 | 阿里云 | 华为云 |
|---|---|---|
| 核心计算服务 | 基因分析平台(Serverless 架构)、ECS、BatchCompute | 弹性云服务器 ECS、容器引擎 CCE、Batch、EIHealth 平台 |
| 存储解决方案 | OSS(对象存储)、NAS(文件存储)、Lindorm(时序数据库) | OBS(对象存储)、SFS Turbo(弹性文件服务)、EVS(云硬盘) |
| 生信工具支持 | 内置 Sentieon、GATK、FastQC 等 200 + 工具镜像 | 支持 Slurm 调度 + 自定义 Docker 镜像,适配鲲鹏 ARM 架构工具 |
| 流程标准兼容 | 原生支持 GA4GH 标准(WDL/CWL) | 通过 Nextflow 集成支持 WDL,适配自研医疗智能体流程 |
| 合规认证 | ISO 27001、HIPAA、等保三级 | 等保四级、ISO 27701、国家增强级安全评估 |
| 典型优势场景 | 超大规模临床样本分析、多中心协作项目 | 国产化部署需求、ARM 架构算力优化、政务科研项目 |
选型建议:临床级多中心研究优先选择阿里云(合规完善 + 工具丰富);国产化适配或 ARM 架构偏好者优先华为云(鲲鹏优化 + 等保四级)。
三、部署前期核心准备:环境与资源规划实操
3.1 账号与权限体系搭建
权限管控是生信云部署的安全基础,需遵循 “最小权限原则” 设计分层体系:
- 阿里云配置:通过 RAM 控制台创建三类角色 —— 管理员(AliyunGenomicsFullAccess 权限)、分析师(仅作业提交权限)、运维员(资源监控权限)。创建 AccessKey 时开启 “短期有效” 模式,搭配 MFA 二次认证,并通过 OSS Bucket Policy 限制数据访问范围。
- 华为云配置:在 IAM 控制台创建用户组,绑定 “EIHealth 项目开发者”“OBS 只读” 等预制策略。使用临时访问密钥(STS)管理批量任务权限,有效期设置不超过 24 小时。
3.2 网络架构规划
生信分析对网络稳定性要求极高,需构建隔离且高效的网络环境:
- VPC 划分:创建独立 VPC,划分子网 —— 管理子网(部署堡垒机,仅开放 22 端口)、计算子网(运行分析任务,无公网访问)、存储子网(挂载文件存储,限制内部通信)。
- 安全组配置:
- 入站规则:允许管理员 IP 访问 22 端口(SSH)、8787 端口(RStudio Server)、8080 端口(流程监控界面)。
- 出站规则:仅开放 HTTPS(443 端口)用于工具下载,禁用不必要的公网访问。
- 带宽优化:测序数据上传建议使用云厂商专线服务(阿里云高速通道 / 华为云 Direct Connect),单流带宽不低于 100Mbps,避免数据传输成为瓶颈。
3.3 存储挂载与工具配置
存储适配是生信分析的关键环节,需根据数据类型选择挂载方案:
阿里云存储配置(OSS+NAS)
- OSSUTIL 工具安装:
bash
# 下载并解压工具
wget https://gosspublic.alicdn.com/ossutil/1.7.19/ossutil64
chmod +x ossutil64
# 配置AccessKey与Endpoint(优先使用内网域名)
./ossutil64 config -e oss-cn-shanghai-internal.aliyuncs.com -i <AccessKeyID> -k <AccessKeySecret>
- OSS 挂载为本地目录(ossfs):
bash
# 安装依赖
sudo apt-get install gdebi-core fuse
# 挂载Bucket至/mnt/oss
ossfs <bucket-name> /mnt/oss -o url=oss-cn-shanghai-internal.aliyuncs.com -o allow_other
- NAS 挂载:通过控制台获取挂载命令,直接挂载至 /opt/analysis 目录,用于存储中间结果(BAM/VCF 文件)提升 IO 性能。
华为云存储配置(OBS+SFS)
- obsutil 安装与配置:
bash
# 下载ARM架构版本(适配鲲鹏服务器)
wget https://obs-community.obs.cn-north-1.myhuaweicloud.com/obsutil/current/obsutil_linux_arm64.tar.gz
tar -zxvf obsutil_linux_arm64.tar.gz
# 交互模式配置
./obsutil config
- SFS Turbo 挂载:在控制台创建文件系统后,通过 NFS 协议挂载至 /opt/shared 目录,支持多计算节点并发访问。
3.4 基础环境搭建
- 操作系统选择:
- X86 架构:优先 CentOS 7.9(工具兼容性最佳)或 Ubuntu 20.04 LTS。
- ARM 架构(华为鲲鹏 / 阿里倚天):选择欧拉(openEuler)22.03 LTS,预装 ARM 优化依赖库。
- 核心依赖安装:
bash
# 安装编译工具与基础库
sudo yum install -y gcc gcc-c++ make cmake zlib-devel bzip2-devel libcurl-devel
# 安装conda环境管理器
wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
bash Miniconda3-latest-Linux-x86_64.sh -b -p /opt/miniconda3
source /opt/miniconda3/bin/activate
四、组学分析本地化适配:工具与流程实战
4.1 核心生信工具云适配指南
生信工具的云适配需解决架构兼容、性能优化和 IO 适配三大问题,以下为典型工具适配方案:
Sentieon(变异检测加速工具)
作为 GATK 的商业替代工具,Sentieon 在云环境中可实现 10 倍以上加速,适配方案如下:
- 版本选择:
- X86 架构:下载
sentieon-genomics-202503.01.tar.gz。 - ARM 架构:下载
arm-sentieon-genomics-202503.01.tar.gz(适配华为鲲鹏 / 阿里倚天)。
- 安装配置:
bash
tar -zxvf sentieon-genomics-202503.01.tar.gz -C /opt/bioinfo/
# 配置环境变量
echo "export PATH=/opt/bioinfo/sentieon-genomics-202503.01/bin:\$PATH" >> ~/.bashrc
source ~/.bashrc
- 云优化参数:
- 线程数设置为 CPU 核心数的 80%(避免资源争抢)。
- 临时目录指向本地 SSD 云盘(/mnt/ssd/tmp),减少 OSS/OBS 访问压力。
- 大基因组(如植物)分析启用 CRAM 格式:
--format cram。
GATK(基因组分析工具集)
- 阿里云快速部署:直接使用基因分析平台预置镜像,通过控制台一键启动:
bash
# 调用平台内置GATK
aliyun genomics CreateWorkflow --Name GATK_HaplotypeCaller --TemplateId gatk-haplotypecaller-v4.4.0
- 华为云自定义部署:
bash
# 基于conda安装
conda create -n gatk -c bioconda gatk4=4.4.0.0 openjdk=11
conda activate gatk
# 配置参考基因组路径(SFS Turbo挂载目录)
gatk --java-options "-Xmx32g" HaplotypeCaller -R /opt/shared/ref/hg38.fasta -I sample.sorted.bam -O sample.g.vcf.gz
FastQC(质量控制工具)
批量质控是生信分析的第一步,云环境适配需解决并行调度问题:
bash
# 批量FastQC脚本(适配阿里云BatchCompute)
for fastq in /mnt/oss/raw_data/*.fastq.gz; do
sample=$(basename $fastq .fastq.gz)
# 提交独立任务
aliyun batch SubmitJob --JobName fastqc_$sample --JobQueue default --TaskTemplate "{\"Command\":\"fastqc -o /mnt/nas/qc_reports -t 8 $fastq\",\"ImageId\":\"centos_7_9_bioinfo:v1\"}"
done
4.2 生信流程云原生改造
基于 WDL/Nextflow 的流程改造是实现标准化分析的核心,以下为两大平台适配方案:
阿里云 Nextflow+BatchCompute 配置
- 安装 Nextflow:
bash
curl -s https://get.nextflow.io | bash
sudo mv nextflow /usr/local/bin/
- 创建配置文件(nextflow.config):
groovy
profiles {
aliyun {
process.executor = 'aliyunbatch'
process.queue = 'bioinfo_queue'
process.container = 'registry.cn-shanghai.aliyuncs.com/bioinfo/gatk:4.4.0'
// OSS存储配置
aliyun.batch.ossWorkingDir = 'oss://bioinfo-bucket/work/'
aliyun.batch.accessKeyId = '<AccessKeyID>'
aliyun.batch.accessKeySecret = '<AccessKeySecret>'
}
}
- 运行流程:
bash
nextflow run main.nf -profile aliyun --input oss://bioinfo-bucket/raw_data/ --output oss://bioinfo-bucket/results/
华为云 Nextflow+EIHealth 平台配置
华为云 EIHealth 平台原生支持 Nextflow,可通过 API 实现流程管理:
- 安装 Nextflow 引擎:
bash
# 调用EIHealth API安装
curl -X POST "https://eihealth.cn-north-4.myhuaweicloud.com/v1/{project_id}/nextflow/engines" \
-H "X-Auth-Token: {token}" \
-H "Content-Type: application/json" \
-d '{"version":"23.10.1"}'
- 上传并运行流程:
bash
# 上传流程文件
curl -X PUT "https://eihealth.cn-north-4.myhuaweicloud.com/v1/{project_id}/eihealth-projects/{project_id}/nextflow/workflows/{workflow_id}" \
-H "X-Auth-Token: {token}" \
-F "workflow_file=@main.nf" \
-F "main_file=main.nf"
4.3 自研工具云适配要点
对于实验室自研工具,需重点解决兼容性与性能问题:
- 架构适配:采用容器化封装,通过多阶段构建适配 X86 与 ARM 架构:
dockerfile
# ARM架构适配(华为鲲鹏)
FROM arm64v8/centos:7 AS builder
RUN gcc -o my_tool my_tool.c
# X86架构适配
FROM amd64/centos:7
COPY --from=builder /my_tool /usr/local/bin/
- IO 优化:工具输出路径优先指向本地云盘或文件存储(NAS/SFS),完成后通过脚本同步至对象存储:
bash
# 分析完成后同步至OBS
./my_tool -i input.txt -o /mnt/sfs/output.txt
obsutil cp /mnt/sfs/output.txt obs://bioinfo-bucket/results/ --recursive
五、批量任务调度:从部署到优化的全流程
5.1 云平台原生调度服务实战
阿里云 BatchCompute 调度
适用于万级样本批量分析,典型 WGS 流程调度配置:
- 创建作业模板:
json
{
"JobName": "WGS_Analysis",
"TaskTemplate": {
"Command": "sentieon bwa mem -t 16 -R @RG\\tID:$sample\\tSM:$sample $ref $fastq1 $fastq2 | sentieon util sort -o $sample.sorted.bam -t 16",
"ImageId": "registry.cn-shanghai.aliyuncs.com/bioinfo/sentieon:202503",
"ResourceRequirements": [{"Type":"CPU","Value":"16"},{"Type":"Memory","Value":"64"}]
},
"Parameters": [{"Key":"sample","ValueFrom":"sample_list.txt"},{"Key":"fastq1","Value":"oss://bioinfo-bucket/raw_data/$sample_R1.fastq.gz"}]
}
- 提交批量任务:
bash
aliyun batch SubmitJob --JobTemplate file://job_template.json --JobQueue wgs_queue --ParameterFile sample_list.txt
华为云 Batch+Slurm 调度
华为云支持 Slurm 与 Batch 结合,适配 HPC 场景:
- 部署 Slurm 集群:通过控制台一键创建包含 1 个调度节点、N 个计算节点的集群,自动安装 Slurm 与 Gearbox 监控程序。
- 提交任务:
bash
# 创建批量任务脚本
cat > batch_job.sh << EOF
#!/bin/bash
#SBATCH --job-name=star_align
#SBATCH --nodes=1
#SBATCH --cpus-per-task=8
#SBATCH --mem=32G
STAR --genomeDir /mnt/sfs/genome/index --readFilesIn $1 $2 --outFileNamePrefix /mnt/sfs/aligned/$3
EOF
# 批量提交
for sample in $(cat sample_ids.txt); do
sbatch batch_job.sh obs://raw_data/${sample}_R1.fastq.gz obs://raw_data/${sample}_R2.fastq.gz $sample
done
5.2 自定义调度方案:Airflow+Celery
对于复杂依赖型任务(如多组学整合分析),需构建自定义调度系统:
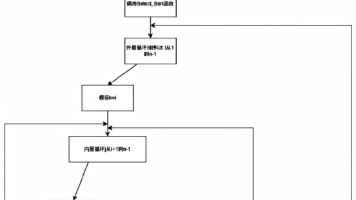
- 部署架构:
- 控制节点:部署 Airflow WebServer 与元数据库(RDS / 云数据库)。
- 工作节点:部署 Celery Worker,弹性扩容至 100 + 节点。
- 任务 DAG 定义:
python
from airflow import DAG
from airflow.operators.bash import BashOperator
from datetime import datetime
default_args = {'owner': 'bioinfo', 'start_date': datetime(2024, 1, 1)}
with DAG('multi_omics_analysis', default_args=default_args, schedule_interval=None) as dag:
# 质控任务
qc = BashOperator(task_id='fastqc', bash_command='fastqc -o /mnt/nas/qc {{ params.fastq }}')
# 比对任务
align = BashOperator(task_id='star_align', bash_command='STAR --genomeDir {{ params.index }} --readFilesIn {{ params.fastq1 }} {{ params.fastq2 }}')
# 定量任务
quant = BashOperator(task_id='salmon_quant', bash_command='salmon quant -i {{ params.salmon_index }} -1 {{ params.fastq1 }} -2 {{ params.fastq2 }}')
# 任务依赖
qc >> align >> quant
5.3 调度性能优化策略
- 资源弹性配置:
- 阿里云:配置弹性伸缩规则,当任务队列长度 > 50 时自动扩容计算节点,空闲时间 > 30 分钟时缩容。
- 华为云:启用 “按需分配” 模式,通过 CCE 容器服务实现 Pod 秒级扩容。
- 任务优先级调度:
- 临床样本标记为 “高优先级”(priority=10),科研样本为 “普通优先级”(priority=5)。
- 阿里云 Batch 配置:
--JobPriority 10;华为云 Slurm 配置:#SBATCH --priority=10。
- IO 性能优化:
- 采用 “对象存储 + 文件存储” 混合架构:原始数据存 OSS/OBS,中间结果存 NAS/SFS,最终结果同步回对象存储。
- 启用阿里云 OSS “传输加速” 或华为云 OBS “全球加速”,跨区域访问延迟降低 40% 以上。
六、批量任务调度避坑指南:8 大核心问题与解决方案
6.1 资源配置失衡:计算资源浪费或不足
典型场景:GATK 变异检测任务因内存配置不足频繁 OOM,而 FastQC 任务占用过多 CPU 导致资源浪费。解决方案:
- 建立工具资源配置表(如下),精准匹配需求:
| 工具 / 流程 | CPU 核心数 | 内存(CPU: 内存) | 存储类型 |
|---|---|---|---|
| FastQC | 4-8 | 1:2(如 8 核 16G) | OSS/OBS 直接访问 |
| BWA 比对 | 16-32 | 1:4(如 16 核 64G) | NAS/SFS |
| GATK HaplotypeCaller | 8-16 | 1:8(如 8 核 64G) | NAS/SFS |
| WGS 全流程 | 32 | 1:4(32 核 128G) | 混合存储架构 |
- 启用云平台 “资源监控”:阿里云 ARMS 或华为云 CES,设置内存使用率 > 85% 时自动扩容,<20% 时报警。
6.2 存储 IO 瓶颈:小文件读写性能低下
典型场景:批量处理 1000 个小片段 FASTQ 文件(每个 < 100MB),OSS/OBS 访问延迟高达数百毫秒。解决方案:
- 文件合并预处理:通过 Shell 脚本合并小文件,减少访问次数:
bash
# 合并同样本小文件
cat sample_*_R1.fastq.gz > sample_R1.fastq.gz
rm sample_*_R1.fastq.gz
- 缓存加速:阿里云启用 OSS “智能缓存”,华为云配置 OBS “客户端缓存”,将热点文件缓存至本地云盘。
- 存储分级:仅将原始数据和最终结果存对象存储,中间文件全部存 NAS/SFS Turbo(IOPS 达 10 万 +)。
6.3 任务依赖失控:前序失败导致连锁错误
典型场景:质控未通过的样本流入比对环节,导致后续分析全部无效,浪费计算资源。解决方案:
- 标记文件控制:每个步骤完成后生成标记文件,后续任务依赖标记文件存在性判断:
bash
# 质控步骤
fastp -i input.fastq -o clean.fastq && touch qc.ok
# 比对步骤(依赖qc.ok)
if [ -f qc.ok ]; then
bwa mem ref.fasta clean.fastq -o aligned.sam
else
echo "QC failed, abort alignment" && exit 1
fi
- 流程引擎原生控制:Nextflow 通过
publishDir与when关键字实现条件执行:
groovy
process alignment {
input:
file 'clean.fastq' from clean_ch
file 'qc.ok' from qc_ch
when: file('qc.ok').exists()
output:
file 'aligned.bam' into align_ch
script:
'''
bwa mem ref.fasta clean.fastq -o aligned.bam
'''
}
6.4 日志与监控缺失:故障排查无据可依
典型场景:批量任务中途失败,因未保存日志无法定位是工具报错还是资源不足导致。解决方案:
- 全流程日志收集:
bash
# 任务脚本添加日志输出
./analysis.sh > ${sample}_analysis.log 2>&1
# 同步日志至对象存储
ossutil cp ${sample}_analysis.log oss://bioinfo-bucket/logs/
- 关键指标监控:配置云监控告警 ——CPU 使用率 > 95%、内存使用率 > 90%、磁盘空间 < 10% 时触发短信通知。
- 任务状态可视化:阿里云 Batch 控制台或华为云 EIHealth 平台,实时查看任务 “等待 / 运行 / 失败” 状态分布。
6.5 成本失控:弹性资源未及时释放
典型场景:任务完成后计算节点未自动释放,闲置 10 小时导致额外费用产生。解决方案:
- 自动释放配置:
- 阿里云 Batch:提交任务时设置
--JobReleaseAfterCompletion true。 - 华为云 ECS:创建实例时勾选 “任务完成后自动释放”。
- 成本监控告警:设置日消费阈值(如 500 元),超过时自动暂停非核心任务。
- Spot 实例选型:采用竞价实例(阿里云 Spot 实例 / 华为云竞价实例),成本降低 50%-70%,适合非紧急科研任务。
6.6 工具版本混乱:结果不可复现
典型场景:不同时间提交的任务使用不同版本 GATK,导致变异检测结果不一致。解决方案:
- 容器化固化环境:每个工具版本封装为独立 Docker 镜像,如
gatk:4.4.0、gatk:4.3.0。 - 版本管理脚本:记录任务使用的工具版本:
bash
# 记录版本信息
echo "GATK version: $(gatk --version)" >> ${sample}_version.log
echo "Sentieon version: $(sentieon --version)" >> ${sample}_version.log
- 流程版本控制:使用 Git 管理 WDL/Nextflow 脚本,每次修改生成新版本标签(如
v1.0.0)。
6.7 网络波动:数据传输中断导致任务失败
典型场景:跨区域访问 OSS/OBS 时,网络抖动导致文件下载中断,任务失败。解决方案:
- 重试机制:工具调用添加重试逻辑,如使用
retry命令:
bash
# 带重试的文件下载
retry 3 ossutil cp oss://bioinfo-bucket/raw_data/sample.fastq.gz ./
- 就近部署:计算节点与存储桶部署在同一区域(如阿里云上海区域、华为云北京区域),避免跨区域传输。
- 专线传输:大规模数据采用云厂商专线(阿里云高速通道 / 华为云 Direct Connect),丢包率降至 0.1% 以下。
6.8 权限过期:AccessKey 失效导致任务中断
典型场景:临时 AccessKey 过期,批量任务中途无法访问 OSS/OBS,全部停滞。解决方案:
- 长期密钥轮换:生产环境使用长期 AccessKey,每 90 天轮换一次,通过 RAM 角色自动更新。
- 密钥监控:设置 AccessKey 过期前 7 天自动告警,通过脚本批量更新配置文件。
- IAM 角色授权:避免直接使用 AccessKey,采用阿里云 RAM 角色或华为云 IAM 角色,实现无密钥访问。
七、实战案例:两大云平台组学分析部署实例
7.1 案例一:阿里云 WGS 全流程部署(基于 Sentieon 加速)
项目需求
300 例人类全基因组样本(30X 深度),实现从 FASTQ 到 VCF 的自动化分析,要求单样本分析时间 < 4 小时,总成本控制在 2 万元内。
部署架构
- 计算资源:Spot 实例(32 核 128G,按需扩容至 30 节点)。
- 存储架构:OSS(原始数据 + 结果)+ NAS(中间文件)。
- 流程引擎:Nextflow + 阿里云 BatchCompute。
核心配置
- Sentieon 加速配置:
bash
# 比对与变异检测脚本
sentieon bwa mem -t 32 -R @RG\tID:$sample\tSM:$sample $ref $fastq1 $fastq2 | \
sentieon util sort -o $sample.sorted.bam -t 32 --temp_dir /mnt/ssd/tmp && \
sentieon driver -t 32 -i $sample.sorted.bam --algo Haplotyper $sample.g.vcf.gz
- Nextflow 配置:启用 OSS 工作目录与 Spot 实例:
groovy
aliyun.batch.spotStrategy = 'SpotWithPriceLimit'
aliyun.batch.spotPriceLimit = 0.8 # 最高出价为按需价格的80%
实施效果
- 单样本分析时间:3.5 小时(传统方案 12 小时)。
- 总成本:1.8 万元(Spot 实例节省 60% 成本)。
- 结果可复现性:变异检测一致性 > 99.9%。
7.2 案例二:华为云 RNA-seq 批量分析(国产化适配)
项目需求
500 例 RNA-seq 样本分析,基于华为鲲鹏 ARM 架构服务器,需满足等保四级合规要求,支持流程自定义。
部署架构
- 计算资源:鲲鹏 ECS(16 核 64G,弹性伸缩至 50 节点)。
- 存储架构:OBS(原始数据)+ SFS Turbo(中间结果)。
- 调度系统:Slurm + 华为云 Batch。
核心配置
- ARM 架构工具适配:
bash
# 安装ARM版本STAR
wget https://github.com/alexdobin/STAR/releases/download/2.7.11a/STAR_aarch64_static
mv STAR_aarch64_static /usr/local/bin/STAR && chmod +x /usr/local/bin/STAR
- Slurm 任务提交:
bash
# 批量RNA-seq分析脚本
for sample in $(cat sample_list.txt); do
sbatch << EOF
#!/bin/bash
#SBATCH --job-name=${sample}_rnaseq
#SBATCH --nodes=1
#SBATCH --cpus-per-task=16
#SBATCH --mem=64G
#SBATCH --output=${sample}.out
STAR --genomeDir /mnt/sfs/genome/star_index --readFilesIn obs://raw_data/${sample}_R1.fastq.gz obs://raw_data/${sample}_R2.fastq.gz --outFileNamePrefix /mnt/sfs/results/${sample}_
salmon quant -i /mnt/sfs/genome/salmon_index -1 obs://raw_data/${sample}_R1.fastq.gz -2 obs://raw_data/${sample}_R2.fastq.gz -o /mnt/sfs/results/${sample}_quant
obsutil cp /mnt/sfs/results/${sample}_* obs://results-bucket/${sample}/ --recursive
EOF
done
实施效果
- 单样本分析时间:1.5 小时(X86 架构 2 小时)。
- 合规性:满足等保四级与医疗数据安全要求。
- 资源利用率:平均 CPU 使用率 82%,内存使用率 75%。
八、总结与未来趋势
阿里云与华为云在生信部署领域各有侧重:阿里云凭借成熟的基因分析平台和丰富的工具生态,更适合超大规模临床研究与快速部署需求;华为云则以鲲鹏 ARM 架构、等保四级合规和混合云能力,成为国产化场景的优选。
成功部署的核心在于三点:一是精准的资源选型,匹配工具特性与业务需求;二是标准化的流程改造,通过容器化与流程引擎实现可复现性;三是精细化的调度管理,通过监控与优化规避常见陷阱。
未来,生信云部署将呈现三大趋势:Serverless 化(无需管理集群,按任务付费)、AI 加速(如阿里云 PAI 结合生信工具实现变异检测加速)、多云协同(跨平台资源调度与数据共享)。掌握云平台核心适配与调度技巧,将成为生信工程师应对数据爆炸挑战的关键能力。
更多推荐
 已为社区贡献6条内容
已为社区贡献6条内容







所有评论(0)