【生信MOOC】生物序列比对工具
1、需了解的背景知识2、替换计分矩阵核酸替换计分矩阵蛋白质替换计分矩阵3、序列比对方法(1)打点法(2)两两序列比对算法4、在线序列比对工具EMBL 全局双序列比对工具Biotools 的双序列比对工具
【生信MOOC】生物序列比对工具
文章的文字/图片/代码部分/全部来源网络或学术论文,文章会持续修缮更新,仅供大家学习使用。
目录
1、需了解的背景知识
(1)Fasta文件格式和一些其他形式的生物数据文件格式,参考以下博客:
(2)可以稍微看一下之前整理的一些序列比对算法和知识点,参考以下博客,本篇blog主要是介绍序列比对工具:
【生信】生物序列比对_梦里是碗妹的博客-CSDN博客_生物序列比对
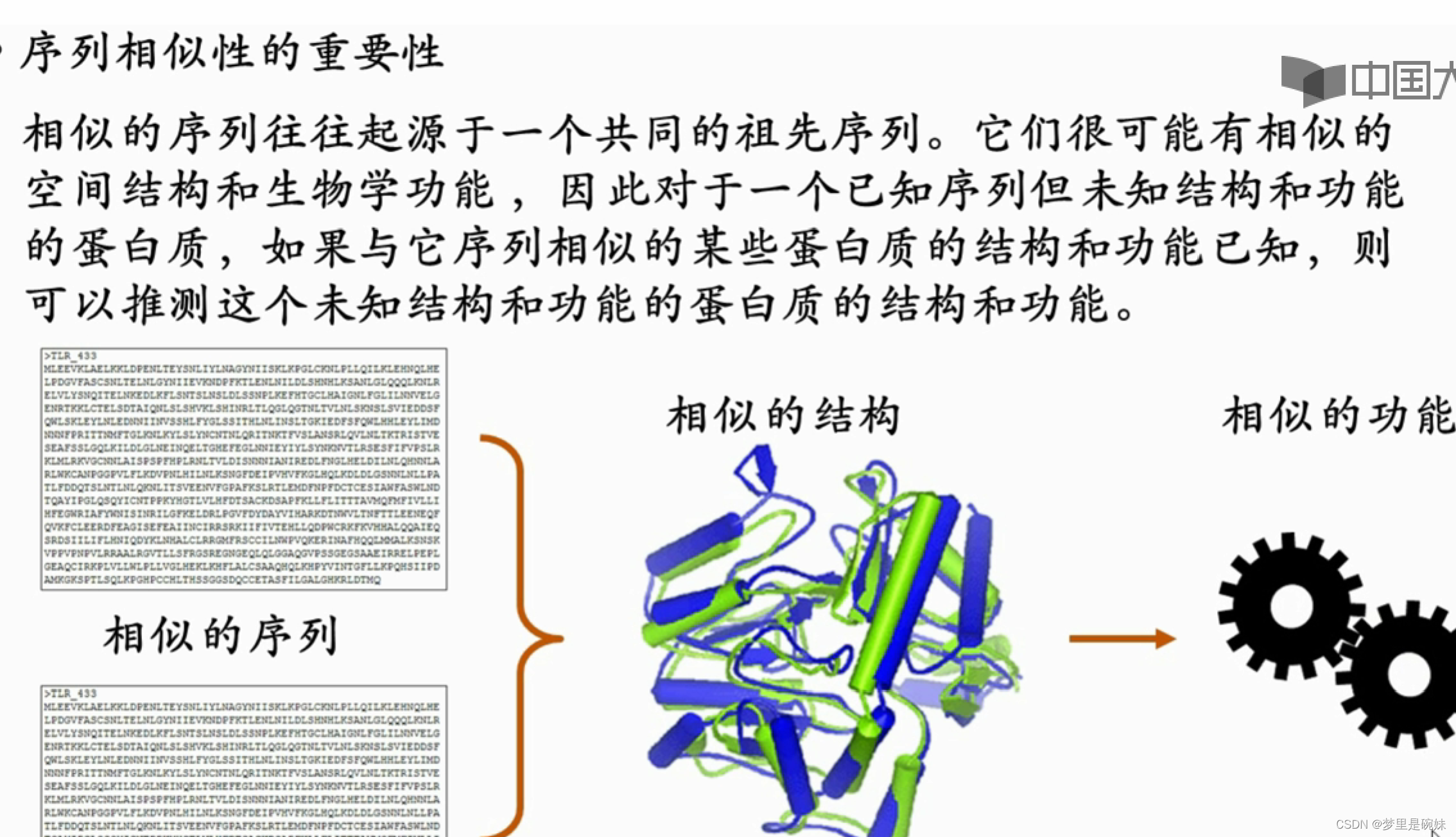
(3)序列相似性与一致性
一致度(identity):如果两个序列长度相同,那么它们的一致度可以暂时定义为它们对应位置上相同的残基数目占总长度的百分比。一个残基就是指一个字母(氨基酸或碱基)。
下例中,上下相同的残基位置有 2 个,序列长度为 4。它们的一致度就是 2 除以 4,50%。

相似度(similarity):如果两个序列长度相同,那么它们的相似度可以暂时定义为他们对应位置上相似的残基与相同的残基的数目和占总长度的百分比。 残基两两之间的相似量化关系由替换计分矩阵代替。
2、替换计分矩阵
替换计分矩阵:替换记分矩阵是反映残基之间相互替换率的矩阵。它描述了残基两两相似的量化关系。矩阵中行和列分别是 20 种氨基酸,且两两之 间有一个分值。根据这个分值就可以知道谁和谁相似,谁和谁不相似。替换记分矩阵有很多种。DNA 序列有 DNA 序列的替换记分矩阵,蛋白质序列有蛋白质序列的替换记分矩阵, 两者不可混用。
核酸替换计分矩阵
常用的DNA替换计分矩阵:有三种
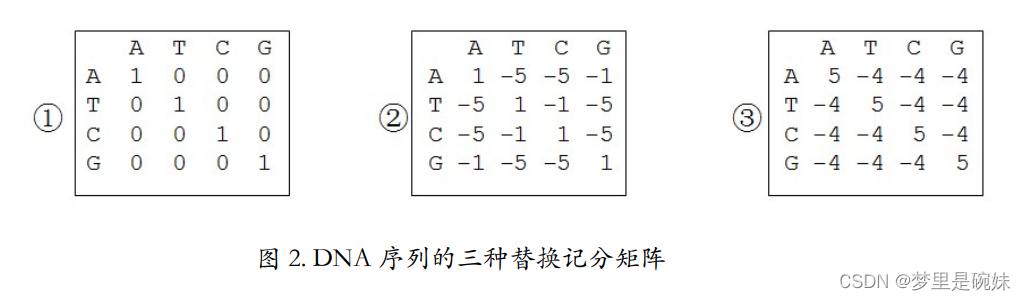
(1)等价矩阵:直接定义相同核苷酸之间的匹配得分为 1,不同核苷酸间的替换得分为 0。在实际的序列比较中很少使用,一般只用于理论计算。
(2)转换-颠换矩阵:核酸的碱基按照环结构特征被划分为两类,一类是嘌呤(含有两个环),一类是嘧啶(含有一个环)。
嘌呤之间、嘧啶之间的替换成为转换,嘌呤和嘧啶之间的替换称为颠换。大自然更倾向于接受嘌呤 和嘌呤之间的替换,以及嘧啶和嘧啶之间的替换,而嘌呤和嘧啶之间的替换会导致不好的事情发生。
转换-颠换矩阵中, 转换的得分比颠换要高为-1 分,而颠换的得分为-5 分。
(3) BLAST 矩阵:经过大量实际比对发现(经验所得),如果令被比对的两个核苷酸相同时 得分为+5 分,不相同为-4 分,这时比对效果最好。这个矩阵广泛地被 DNA 序列比较所采 用。

蛋白质替换计分矩阵
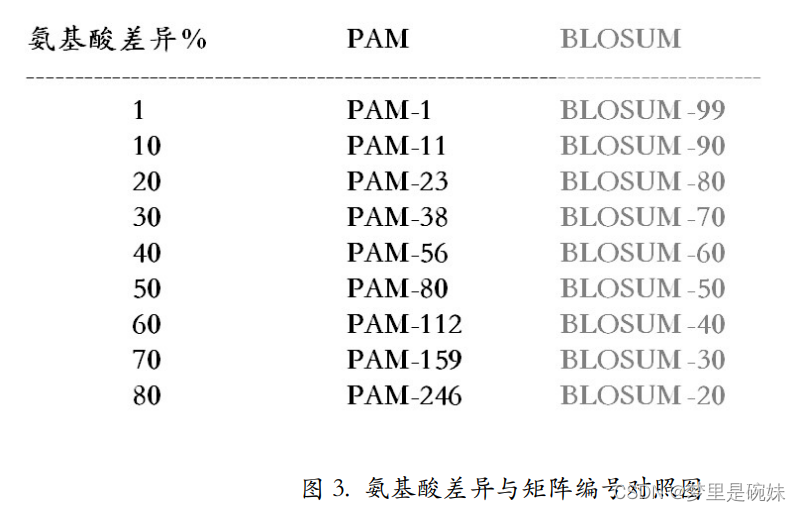
蛋白质最矩阵有五种: 等价矩阵、PAM 矩阵、 BLOSUM 矩阵、遗传密码矩阵、疏水矩阵。


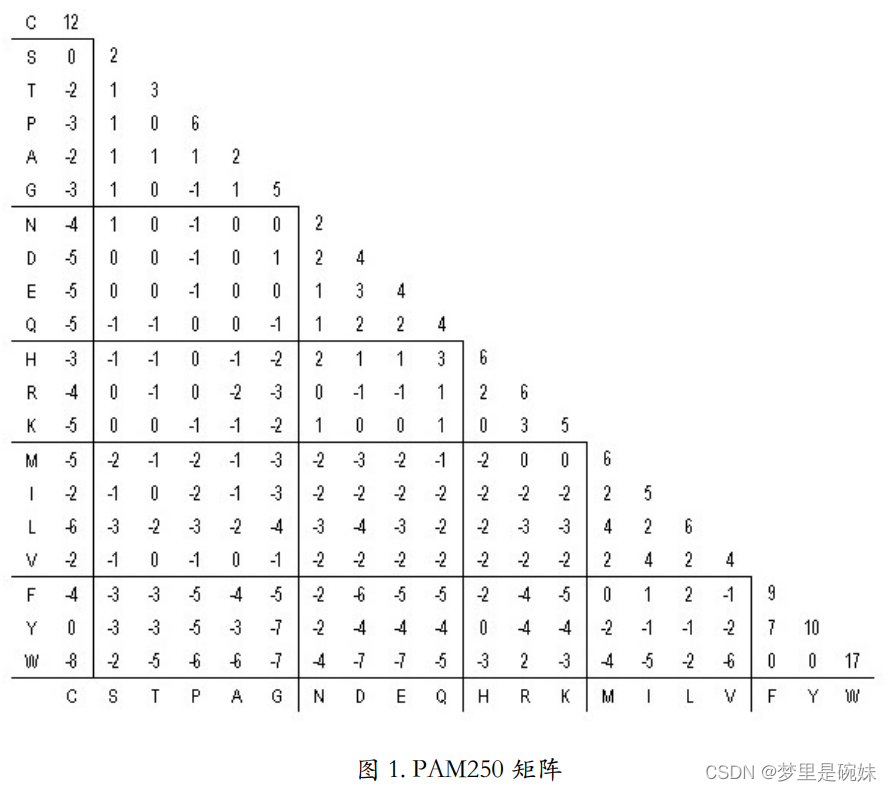
PAM 矩阵:
基础的 PAM-1 矩阵反应的是进化产生的每一百个氨基酸平均发生一个突变的量值,由统计方法得到。 PAM-1 自乘 n 次,可以得到 PAM-n ,表示发生了更多次突变。
需要根据要比较的序列 之间的亲缘关系远近,来选择适合的 PAM 矩阵。
(a)如果序列亲缘关系远,也就是说序列间会 有很多突变,那就选 PAM 后面跟一个大数字的矩阵。
(b)如果亲缘关系近,也就是突变比较少, 序列间大多数地方都是一样的,那就选 PAM 后面跟一个小数字的矩阵。
PAM 矩阵对角线上的数值为匹配氨基酸的得分。其他位置上≥0 的得分代 表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。
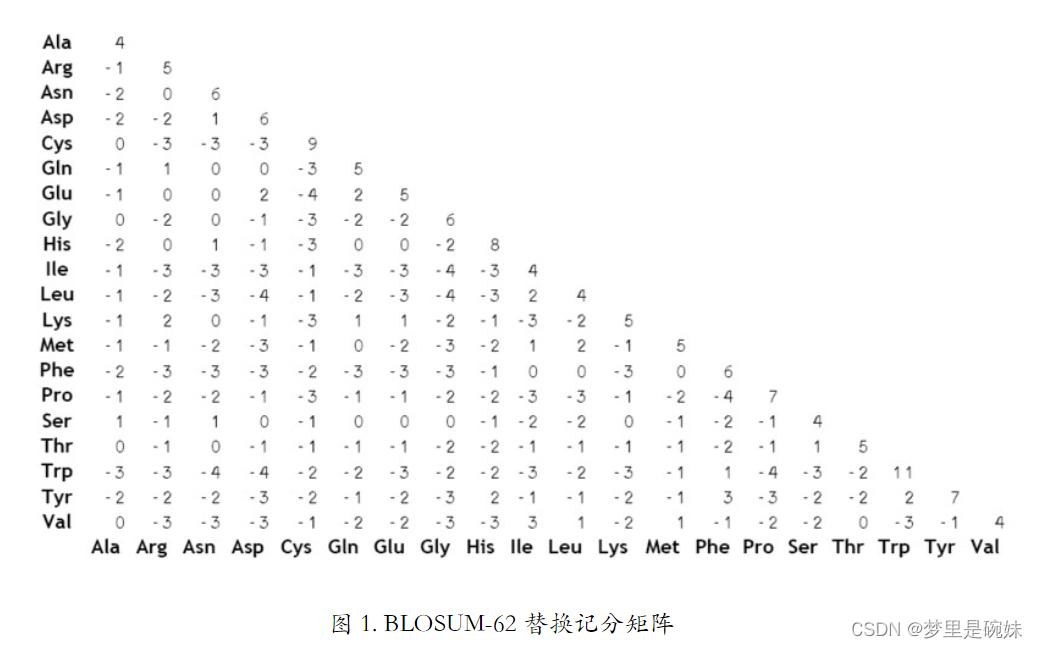
BLOSUM 矩阵:
BLOSUM 矩阵都是通过对大量符合特定要求的序列计算而来的。BLOSUM 矩阵的相似性是根据真实数据产生的,而 PAM 矩阵是通过矩阵自乘外推而来的。
常用的蛋白质替换计分矩阵: BLOSUM-62 替换记分矩阵。
在BLOSUM80 中的 80,代表这个矩阵是由一致度≥80% 的序列计算而来的。同理,BLOSUM62 是指这个矩阵是由一致度≥62%的序列计算而来的。
亲缘关系远的序列,BLOSUM 后面跟一个小数字的矩阵,相似度低。
亲缘关系近的序列,BLOSUM 后面跟一个大数字的矩阵,相似度高。
如何选择PAM和BLOUSUM的参数:
亲缘关系较近的序列之间的比较,用 PAM 数小的矩阵或 BLOSUM 数大的矩阵。
亲缘关系较远的序列之间的比较,用 PAM 数大的矩阵或 BLOSUM 数小的矩阵。
对于关系较远的序列之间 的比较,由于 PAM250 是通过矩阵自乘推算而来的,所以其准确度受到一定限制。相比之下BLOSUM 矩阵更具优势。
对于关系较近的序列之间的比较,用 PAM 或 BLOSUM 矩阵做出 的比对结果,差别不大。
如果关于要比较的序列你不知道亲缘关系远近,那么就闭着眼睛用BLOSUM62。

遗传密码矩阵:
它是通过计算一个氨基酸转换成另一个氨基酸所需的密码子变化的数目而得到的。 矩阵的值对应为据此付出的代价。
如果变化一个碱基就可以使一个氨基酸的密码子转换为另 一个氨基酸的密码子,则这两个氨基酸的替换代价为 1;
如果需要 2 个碱基的改变,则替换 代价为 2;
如从蛋氨酸(Met)到酪氨酸(Tyr)三个密码子都要变,则代价为 3。
适用范围:遗传密码矩阵常用于进化距离的计算,优点是计算结果可以直接用于绘制进化树,但是它在 蛋白质序列比对,尤其是相似程度很低的蛋白质序列比对中,很少被使用。

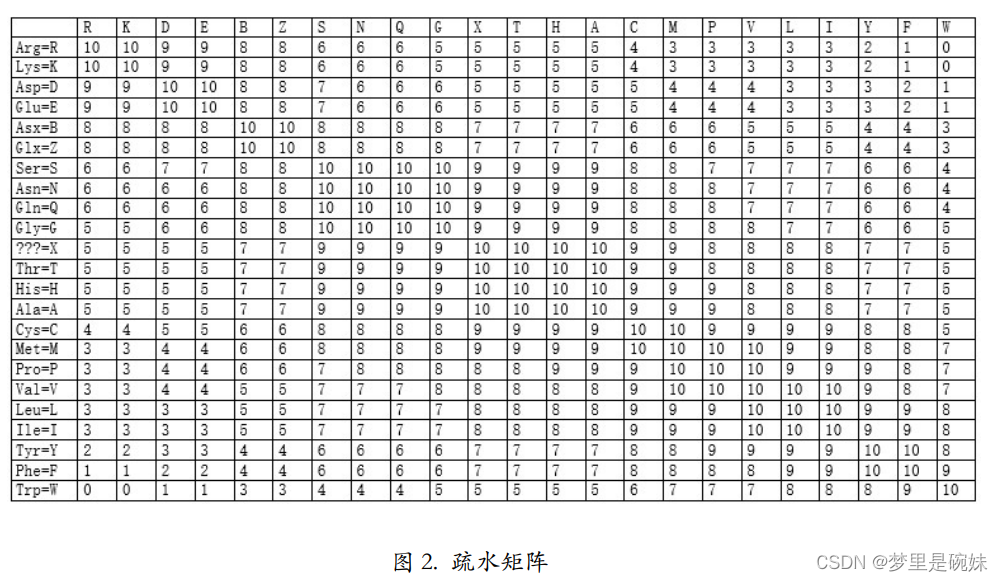
疏水矩阵:
它是根据氨基酸残基替换前后疏水性的变化而得到的矩阵。 若一次氨基酸替换导致疏水特性不发生太大的变化,则这种替换得分高,否则替换得分低。
疏水矩阵物理意义明确,有一定的理化性质依据,适用于偏重蛋白质功能方面的序列比对。 在这个矩阵里,氨基酸按照亲疏水性排列。前边是亲水的,后面是疏水的。
3、序列比对方法
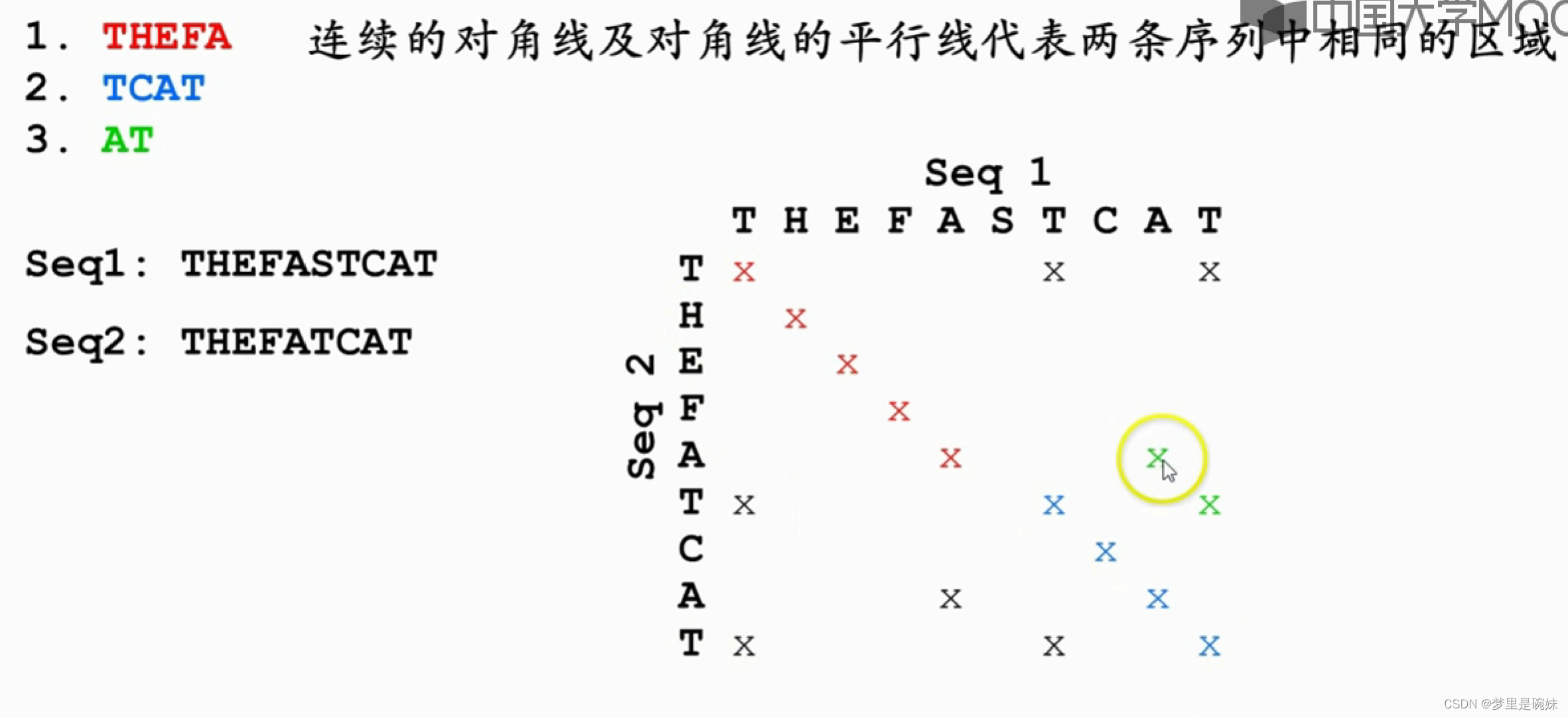
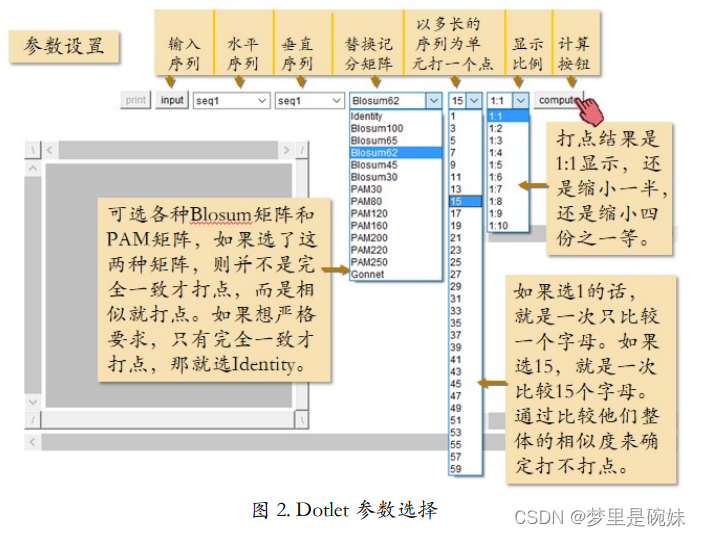
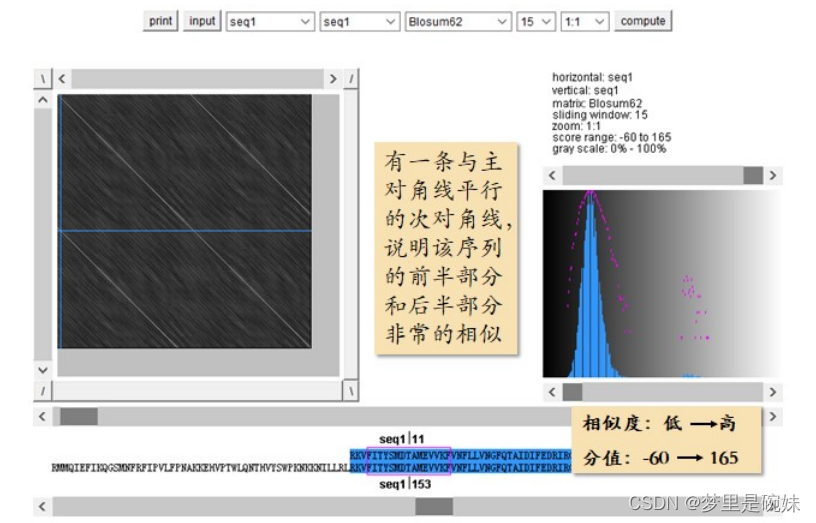
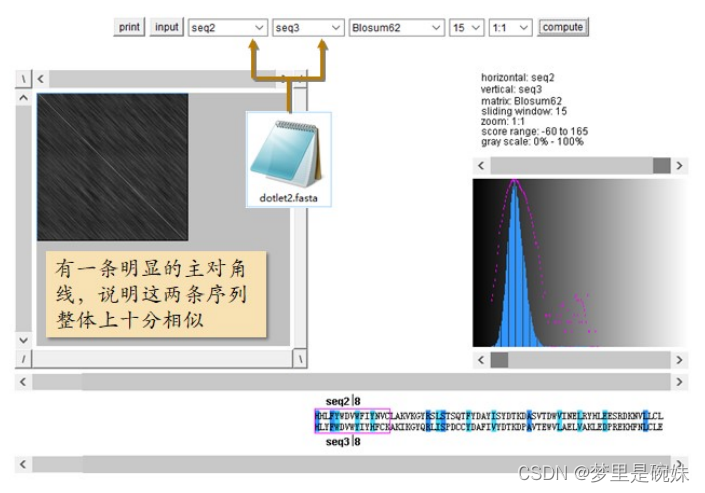
(1)打点法
非常简单无需多说,相同打点,不同空白,查看两个序列的相似情况。


打点法使用的工具:Dotlet
Dotlet 基于 Java 开发,所以页面打开后会蹦出 JAVA 对话框。(http://myhits.isb-sib.ch/cgi-bin/dotlet)。


dotter下载链接:https://sonnhammer.sbc.su.se/download/software/dotter/old_releases/windotter.zip
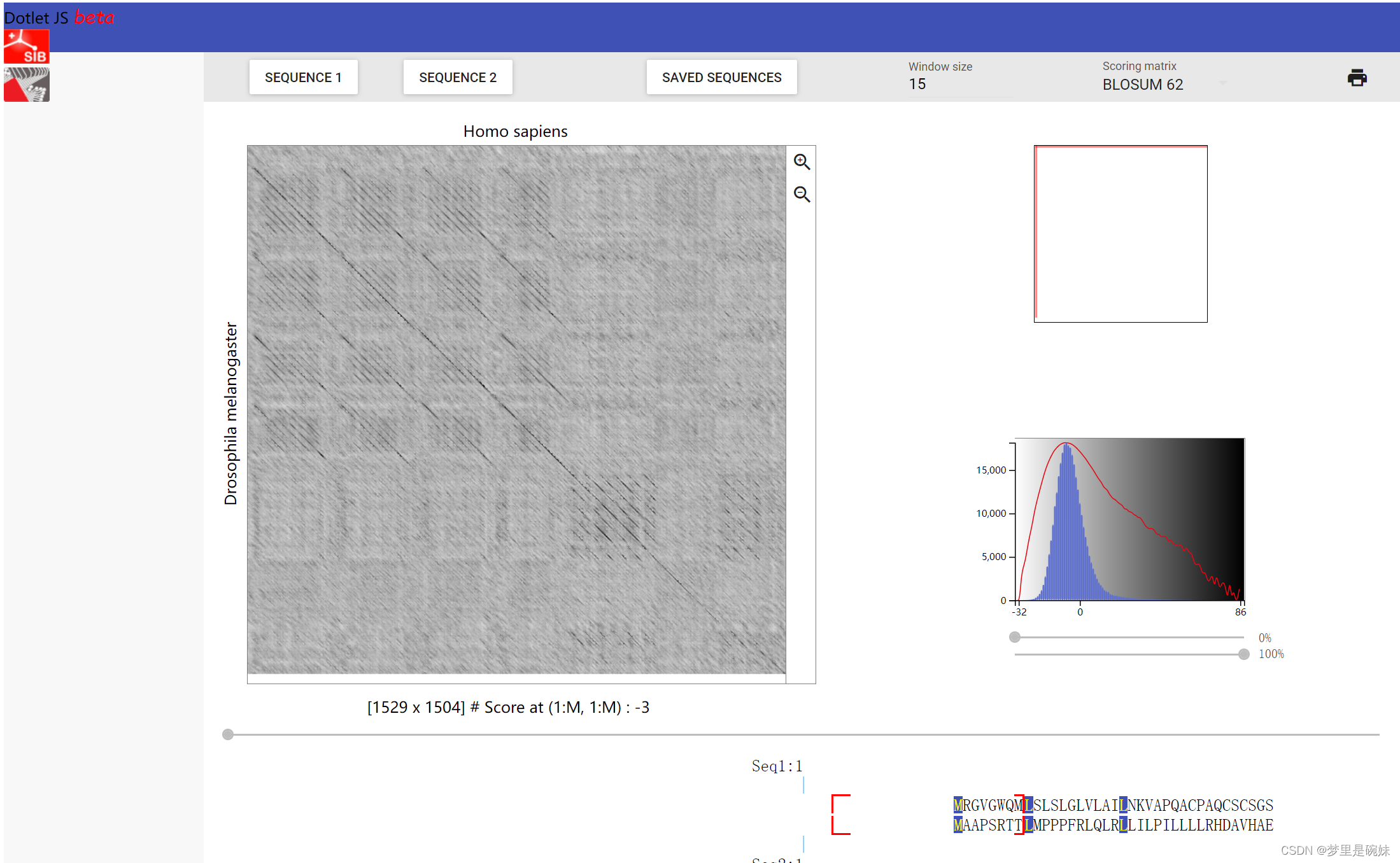
(2)两两序列比对算法
算法原理和计算过程详细参考下面的blog
【生信】生物序列比对_梦里是碗妹的博客-CSDN博客_生物序列比对
【果壳笔记】生物信息学——王秀杰老师部分_梦里是碗妹的博客-CSDN博客
全局比对算法: Needleman-Wunsch 算法。
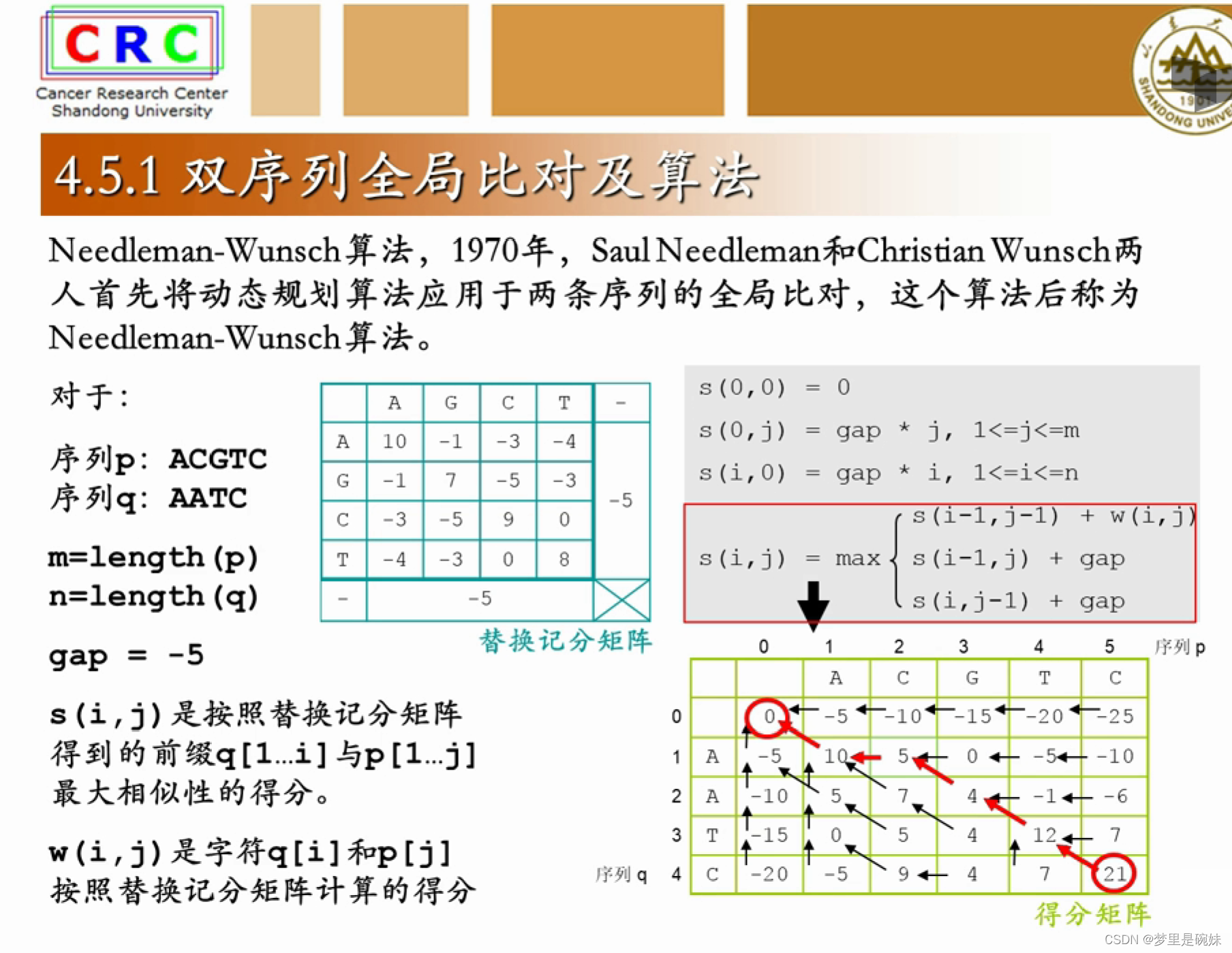
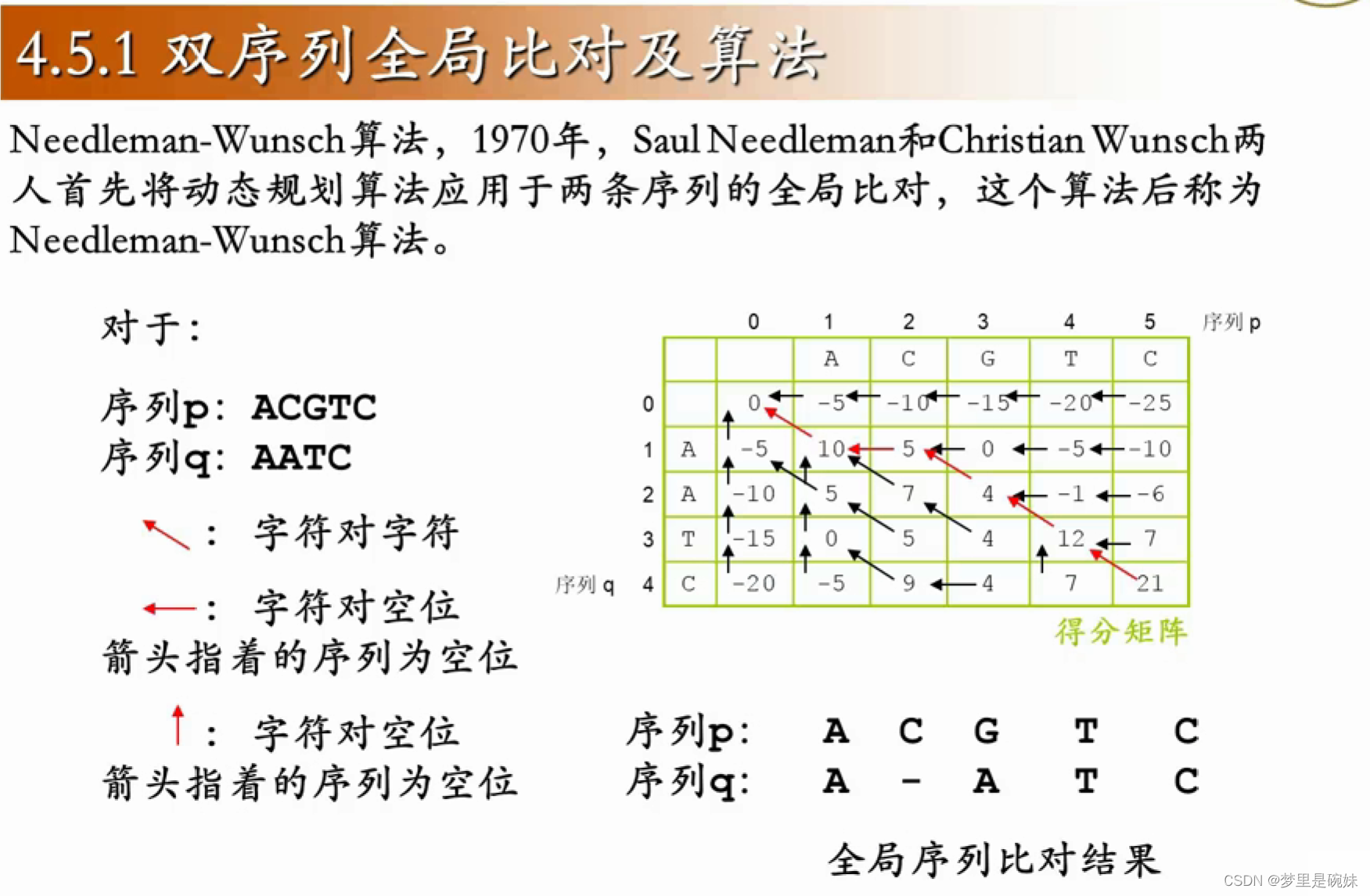

局部比较算法:Smith-Waterman算法
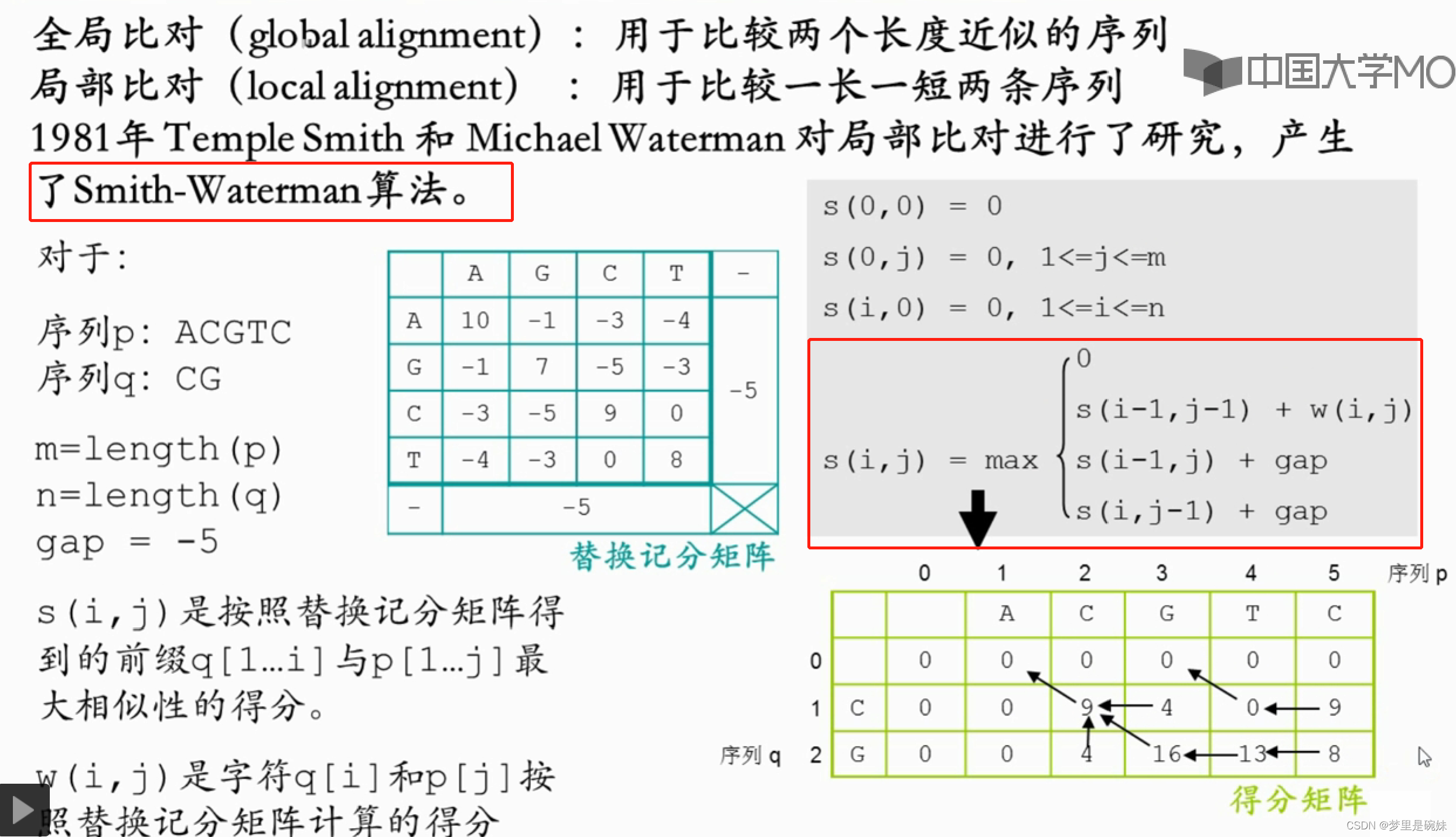
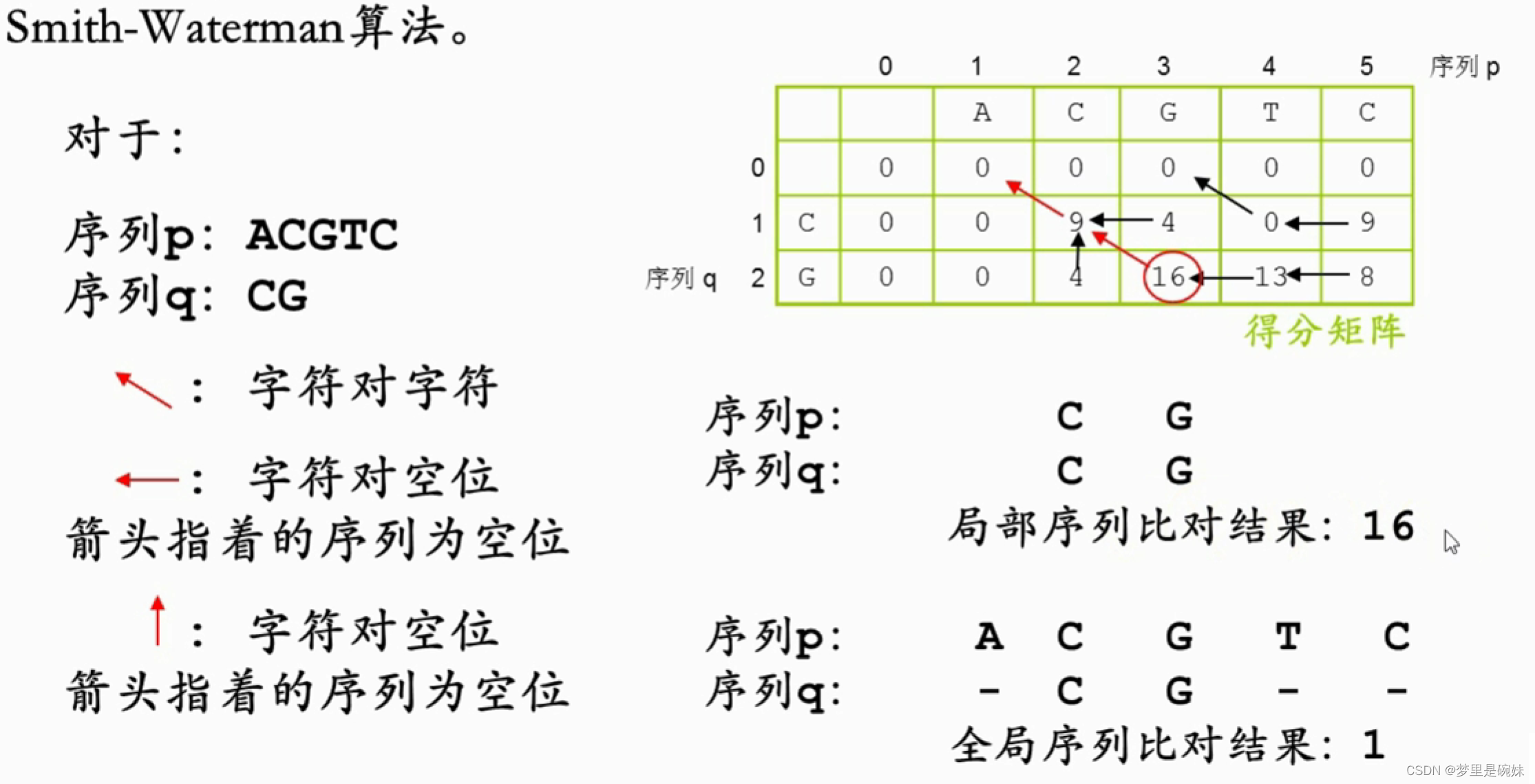
4、在线序列比对工具
EMBL 全局双序列比对工具
Pairwise Sequence Alignment Tools < EMBL-EBI
页面上有有全局比对工具(Global Alignment)、 局部比对工具(Local Alignment)、还有基因组比对工具(Genomic Alignment)。

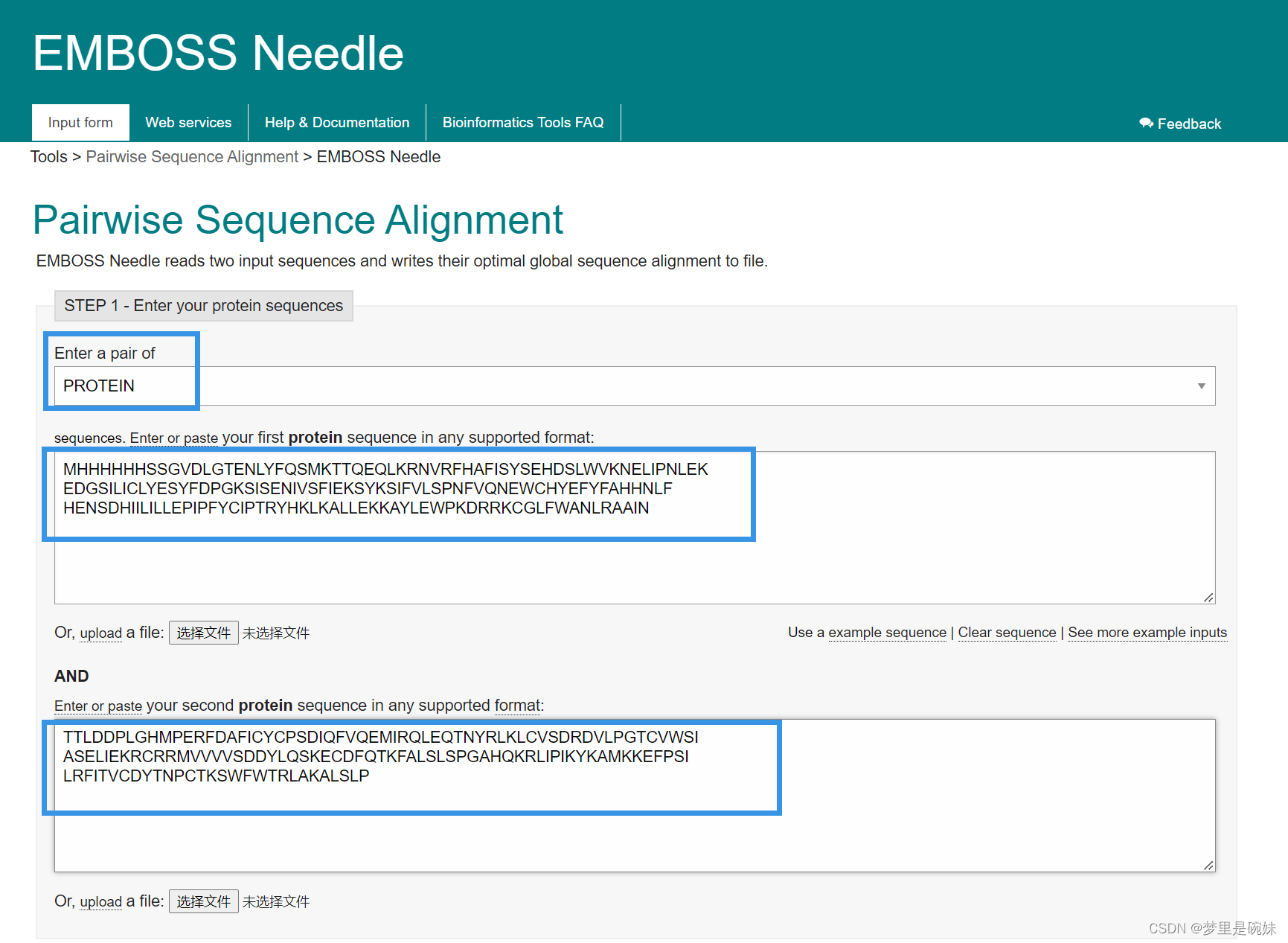
>seq1_for_global_alignment
MHHHHHHSSGVDLGTENLYFQSMKTTQEQLKRNVRFHAFISYSEHDSLWVKNELIPNLEK
EDGSILICLYESYFDPGKSISENIVSFIEKSYKSIFVLSPNFVQNEWCHYEFYFAHHNLF
HENSDHIILILLEPIPFYCIPTRYHKLKALLEKKAYLEWPKDRRKCGLFWANLRAAIN
>seq2_for_global_alignment
TTLDDPLGHMPERFDAFICYCPSDIQFVQEMIRQLEQTNYRLKLCVSDRDVLPGTCVWSI
ASELIEKRCRRMVVVVSDDYLQSKECDFQTKFALSLSPGAHQKRLIPIKYKAMKKEFPSI
LRFITVCDYTNPCTKSWFWTRLAKALSLP
将上面的两个序列进行两两比较。
More options这里可以选择使用哪种替换记分矩阵。 下拉菜单里默认选的就是 BLOSUM62。
可以设置空位罚分,也就是 gap 的分值。这里实际是让你选空位对字母的情况 罚几分,所以显示的是正数,但在计算的过程中还是按照负数处理。
EMBL 比对工具将 gap 分为两种,一种叫“gap 开头(GAP OPEN)”,另一种叫“gap 延长(GAP EXTEND)”。gap 开头就是连续的一串 gap 里面打头的那一个,可以当 它是队长。gap 延长就是剩下的那些 gap,也就是队长后面跟着的小兵。
这一串里,第一个 gap 是 gap 开头,后面的都是 gap 延长。单独的一个 gap 按 gap 开头算。gap 开头和 gap 延长 可以分别定义不同的罚分。默认情况下,gap 开头罚分多,gap 延长罚分少。

当 gap 开头罚分小,gap 延长罚分大的时候,做出来的比对里面,gap 很分散,极少有连续长串 的 gap 出现。
当 gap 开头罚分大,gap 延长罚分小的时候,说明在连 续的字母里插入一个 gap 打开一个缺口要付出很大的代价,因为 gap 开头罚分大。gap 都集中连成长串出现。
所以,可以通过调整 gap 开头和 gap 延长的罚分,可以把序列比对做成我们期待的样子。
如果你对结果没有什么预期,那就请保持默认的参数。

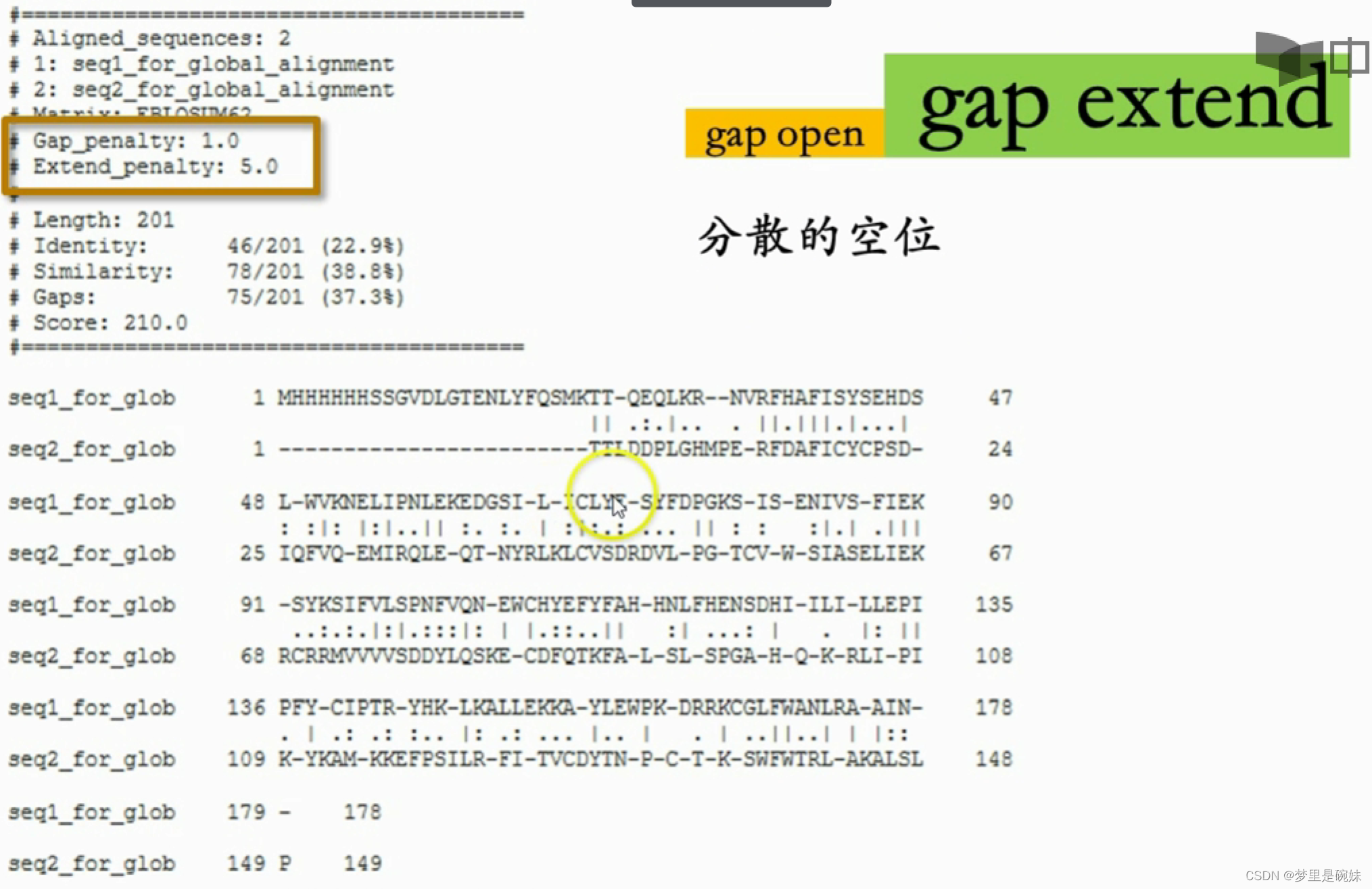
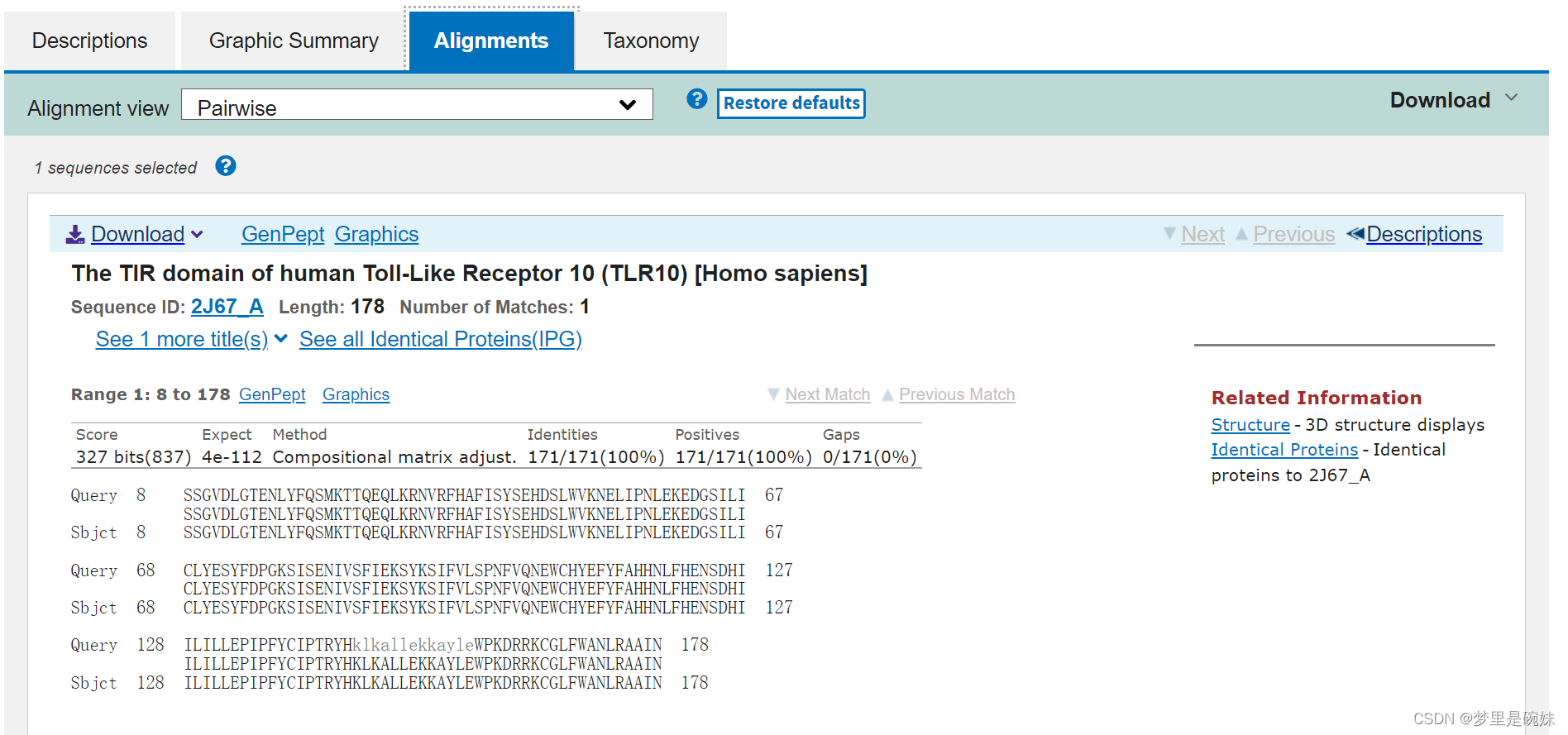
如何看序列对比结果:上下两行是序列,里面插入了 许多的空位。中间这行标记出了哪些位置上下两个字母是完全相同的,用竖线表示。上下两 个字母相似,用双点表示。上下不相似,用单点表示。字母对空位的情况,用空格表示。
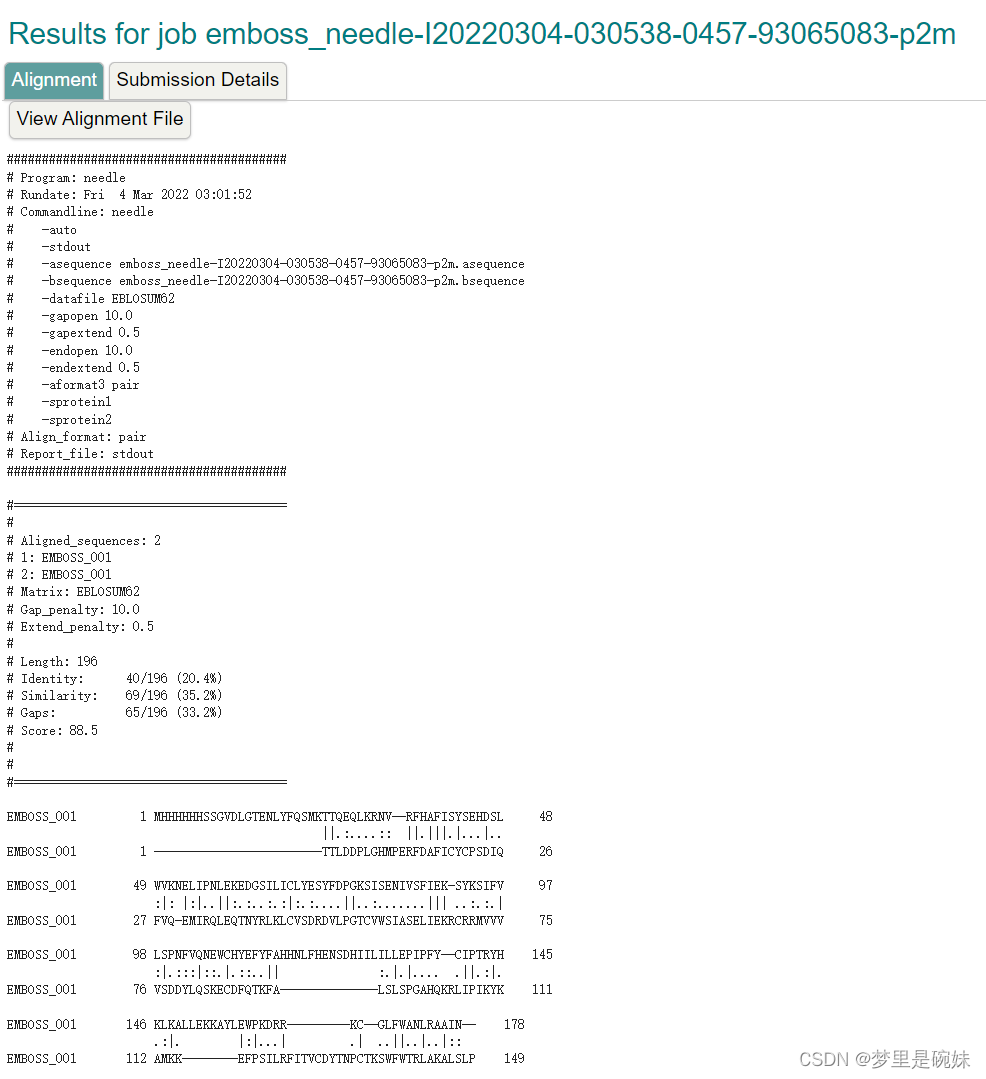
全局比对结果:
默认参数产生的:
局部对比的结果:
EMBL 的局部双序列比对工具可以选择经典的 Smith-Waterman 算法。More options 里面的参数设置和全局比对是一样 的。
默认参数产生的结果: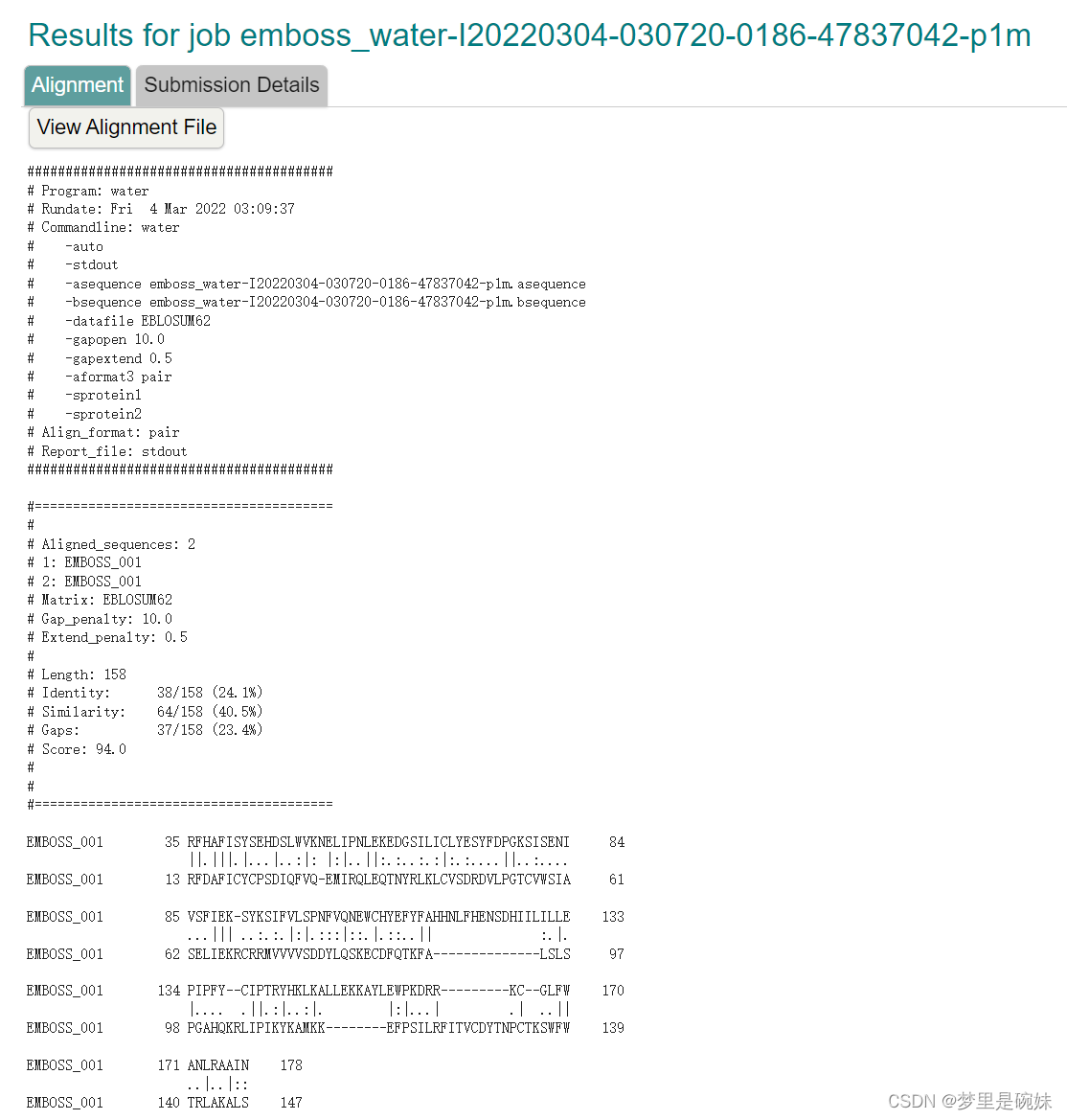
除了一长一短两条序列适合做局部比对,有的时候两条差不多长的序列也可以做局部比对,以找出它们最相似的局部片段。
有时候两条序列并不同源,它们只是有一个功能相似的区域, 这时用局部比对我们就能很快找到这一区域在两条序列中的位置。但是如果做全局比对的话, 结果就不如局部比对明显了。

Biotools 的双序列比对工具
Biotools 的双序列比对工具无论是核酸序列还是蛋白质序列都能做。全局比对,局部比 对,还包括蛋白质二级结构辅助的序列比对。关键的是还能给出得分矩阵。

其他在线序列比对工具:

5、BLAST在线工具
(1)BLAST介绍
BLAST(Basic Local Alignment Search Tool)是快速的数据库相似性搜索工具。它可 以在尽可能准确的前提下,快速的从数据库中找到跟某一条序列相似的序列。
BLAST 的基本原理很简单,要点是片段对的概念。所谓片段对是指两个给定序 列中的一对子序列,它们的长度相等,且可以形成无空位的完全匹配。图 1-A 中方框里的就 是两个片段对。BLAST 从头至尾将两条序列扫描一遍并找出所有片段对,并在允许的阈值 范围内对片段对进行延伸,最终找出高分值片段对(high-scoring pairs, HSPs)(图 1-B)。这 样的计算复杂度是 n 的一次方(n 是序列的长度)。如果做双序列比对话需要构建一个 n 乘 以 n 的表格,计算复杂度是 n 的二次方。所以找高分值片段对比做双序列比对节省了大量的 时间,当然,前提是牺牲了一定的准确度。
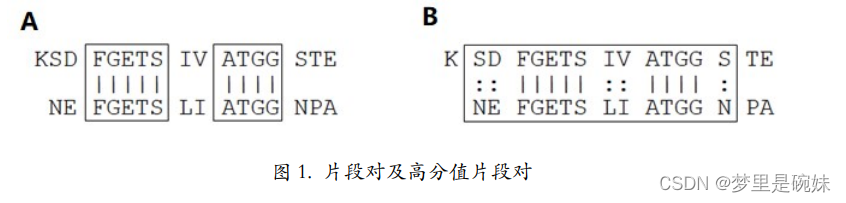
很多数据库都有blast功能。
(2)BLAST的分类
BLAST 实际上是综合在一起的一组工具的统称,它不仅可用于直接对蛋白质序列数据 库和核酸序列数据库进行搜索,而且可以将待搜索的核酸序列翻译成蛋白质序列后再进行搜 索,或者反之,以提高搜索效率。因此 BLAST 可以分为 BLASTp,BLASTn,BLASTx,tBLASTn 和 tBLASTx。
BLASTp 也就是用蛋白质序列搜索蛋白质序列数据库,常用的 BLAST。
BLASTn 是用核酸序列搜索核酸序列数据库,常用的 BLAST。
BLASTx 是将核酸序列按 6 条链翻译成蛋白质序列后搜索蛋白质序列数据库。
为什么是 按 6 条链翻译?
在无法得知翻译起始位点在情况下,翻译可能是从第一个碱基开始,三个三 个的往后翻译,也可能是从第 2 个碱基开始,也可能从第 3 个碱基开始。另外还有可能是从 这条链的互补链上开始,这样又有三个可能的开始位置,加起来一共会产生 6 条可能被翻译 出来的蛋白质序列。这 6 条中有些是真实存在的,有些是不存在,但是谁真谁假我们无从知 晓,所以 6 条序列都要到数据库中去搜索一下试试。
为什么要使用BLASTx?
比如,从核酸序列数据库里找不到跟你手里这条核酸序列相似的序列,或 找到了相似的序列但这些找到的序列无法提供有意义的注释信息。这时,就可以去蛋白质数 据库试试,看看这条核酸序列的翻译产物能不能从蛋白质数据库里找到相似的序列以及有意 义的注释信息。或者说,你不是想找跟你这条核酸序列相似的核酸序列,而是想找跟你这条 核酸序列编码蛋白质相似的蛋白质序列,这时就要做 BLASTx。
tBLASTn是用蛋白质序列 搜核酸序列数据库,核酸数据库中的核酸序列要按 6 条链翻译成蛋白质序列后再被搜索。
为什么要用tBLASTn?
当你不是想找跟你手上这条蛋白质序列相似的蛋白质序列,而是想找跟编码这条 蛋白质序列的核酸序列相似的核酸序列的时候,就要做 tBLASTn。
核酸数据库里有些序列是已经注释了某条核酸序列能够翻译成什么蛋白质序列,但还存在一些还有没注释的。
而且基因可以重叠,注释上说某段 DNA 序列可以编码某个蛋白,但是可能某个未被发现的基因也用到了这段 DNA 序列。而你要搜索的这个蛋白质序列可能 刚好就是这个未被发现的基因的翻译产物。这样就必须把核酸序列所有可能的翻译产物都翻 译出来,才能搜索得到。
tBLASTx是将核酸序列按 6 条链翻译成蛋白质序列后 搜索核酸序列数据库,核酸数据库中的所有核酸序列也要按 6 条链翻译成的蛋白质序列后再被搜索。
用 BLASTn 搜不着的,用 tBLASTx 就能搜着。
(3)BLAST 搜索——NCBI BLASTp
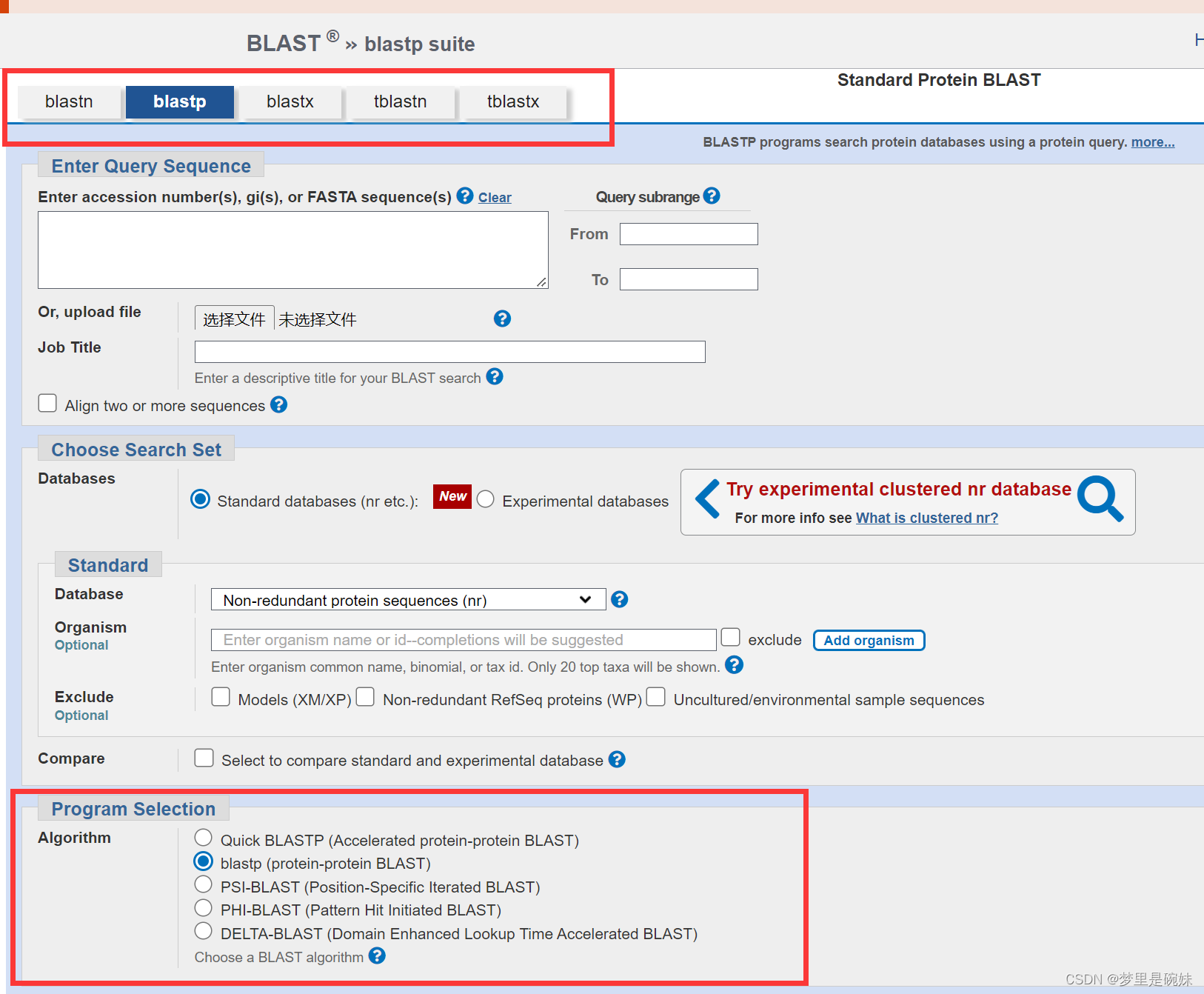
界面中的功能框的设置方法:
1)输入待搜索的蛋白质序列,这条序列可以在示例文 件 blast.fasta 里面找到。
2)指定搜索跟输入序列哪部分相似的序列,如果空着就是全长搜索。
3)给搜索任务起一个名字,如果输入的是 FASTA 格式的序列,那么在输入框里面点一下, 序列的名字就会被自动识别出来。
4)如果在 Align two or more sequences 前面打勾的话,可 以同时提交多个 BLAST 任务。
5)database。虽然是 NCBI 的 BLAST 工具,可以选择的数据库却不只 NCBI 下属的数据库,还包括其他组织的数据库,比如 PDB, Swissprot。事实上,各大数据库网站的 BLAST 工具都可以实现跨平台搜索。我们这次用 NCBI 的 BLAST 工具搜索 SwissProt 数据库。
6)Organism 可以把搜索范围限定在某一特定物种内,或者排除某一物种。
7)Algorithm这一栏里,有之前提到的三种不同的 BLAST 算法,标准 BLAST,PSI-BLAST 和 PHI-BLAST。
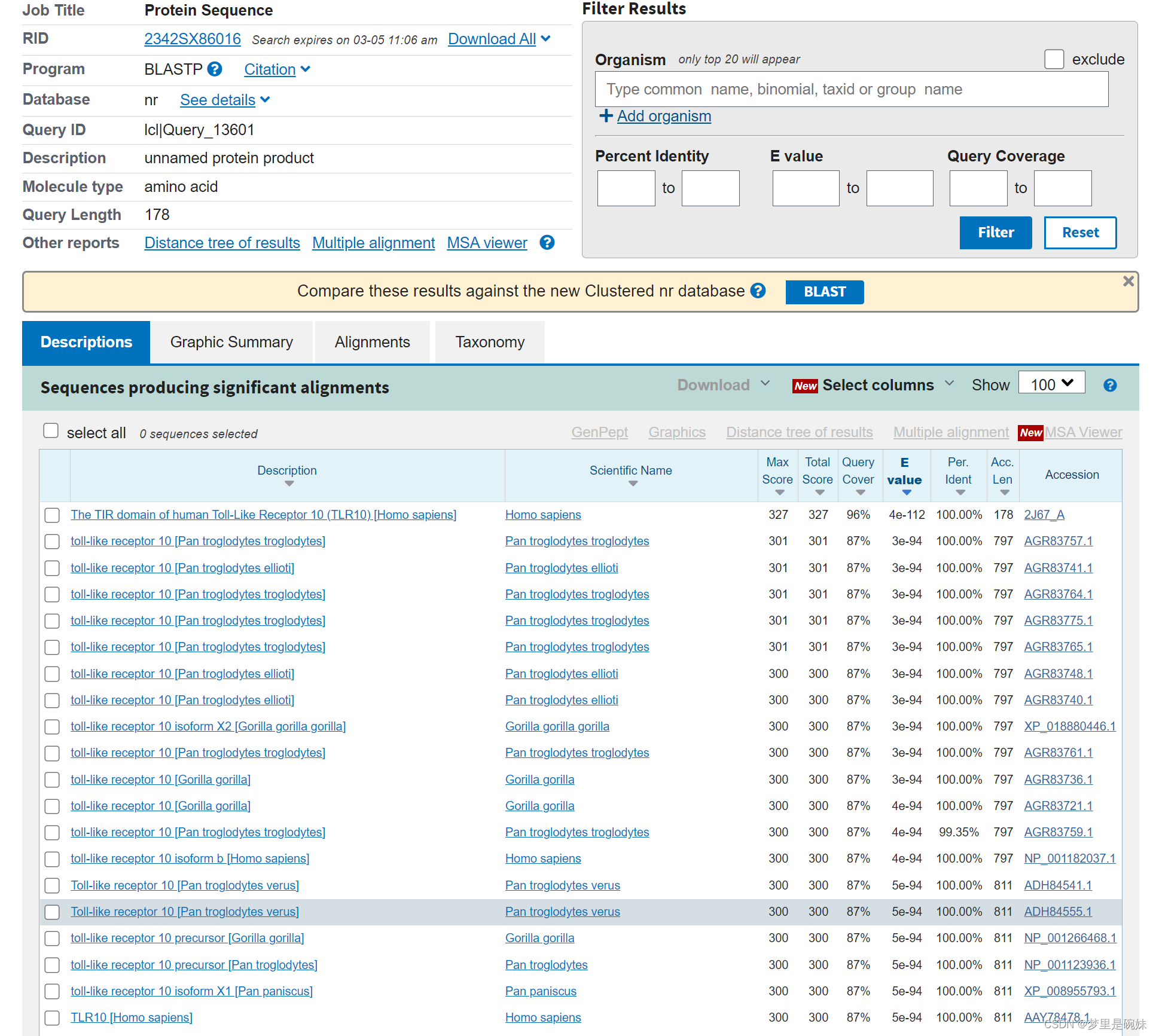

第一部分descripton搜索任务描述部分。
第二部分Graphic Summary是图形化搜索结果部分。
第三部分alignment是序列比对信息。
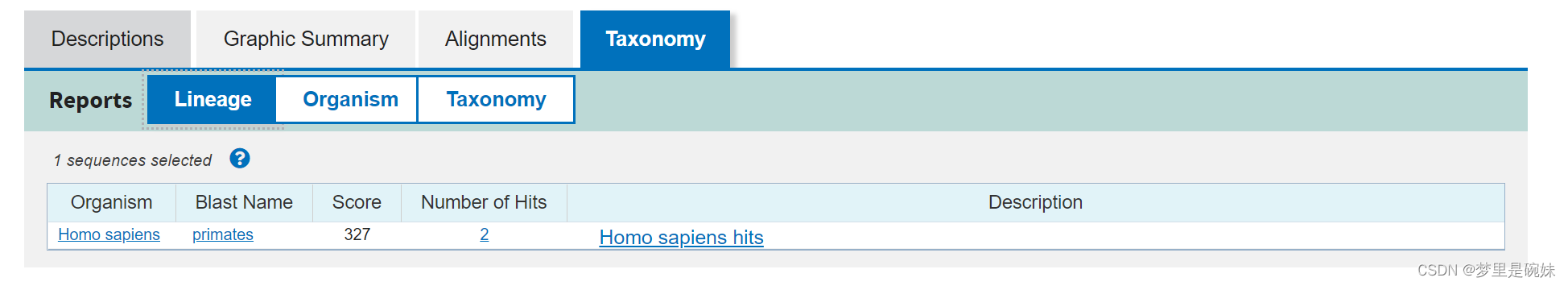
第三部分taxonomy是分类信息。
(4)BLAST 搜索:NCBI PSI-BLAST
PSI-BLAST ( Position-Specific Iterated)可以搜罗出一个庞大的蛋白质家族, 当然也包括标准 BLAST 不小心漏掉的那些远房亲戚。
PSI-BLAST 的特色:是搜完一遍再搜一遍,且从第二次搜索开始,每次搜索前都利 用上一次搜索到的结果创建一个位置特异权重矩阵以扩大本次搜索的范围。如此反复直至没 有新的结果产生为止。
位置特异权重矩阵(Position-Specific Scoring Matrix,简称 PSSM)是 以矩阵的形式,统计一个多序列比对中,每个位置上不同残基出现的百分比。假设 A 的朋 友只有 B,B 朋友除 A 外还有 C。如果输入序列的第一个位置是 A,那么在第一轮没有 PSSM 辅助的情况下,只有第一个位置是 A 或 B 的序列被找到了。它们是图 1-A 中所示的四条序 列。根据这四条序列创建的 PSSM(图 1-B)得知,第一个位置可以是 A,也可以是 B,那么 在第二轮搜索中,除了 A 的朋友 B 之外,B 的朋友 C 也可以出现在第一个位置了。这样如 此反复,我们就可以找到朋友的朋友了。


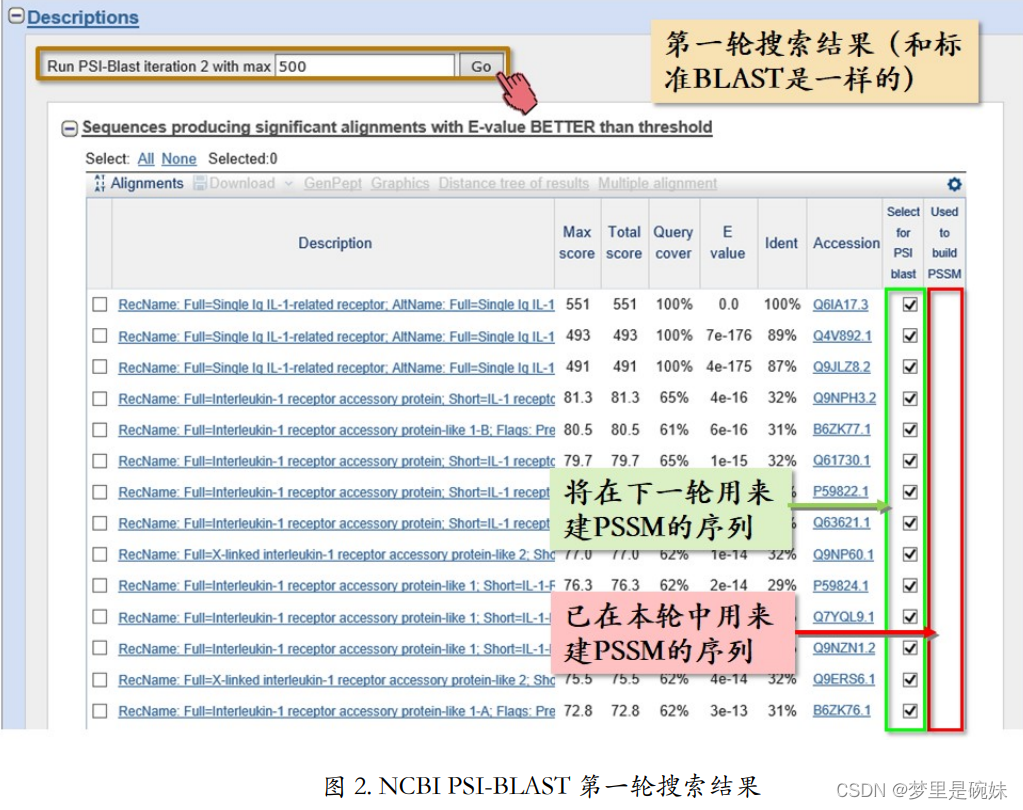
第一轮搜索:
第一轮搜索结果(图 2)和标准 BLAST 是一样的。
第二轮的搜索:
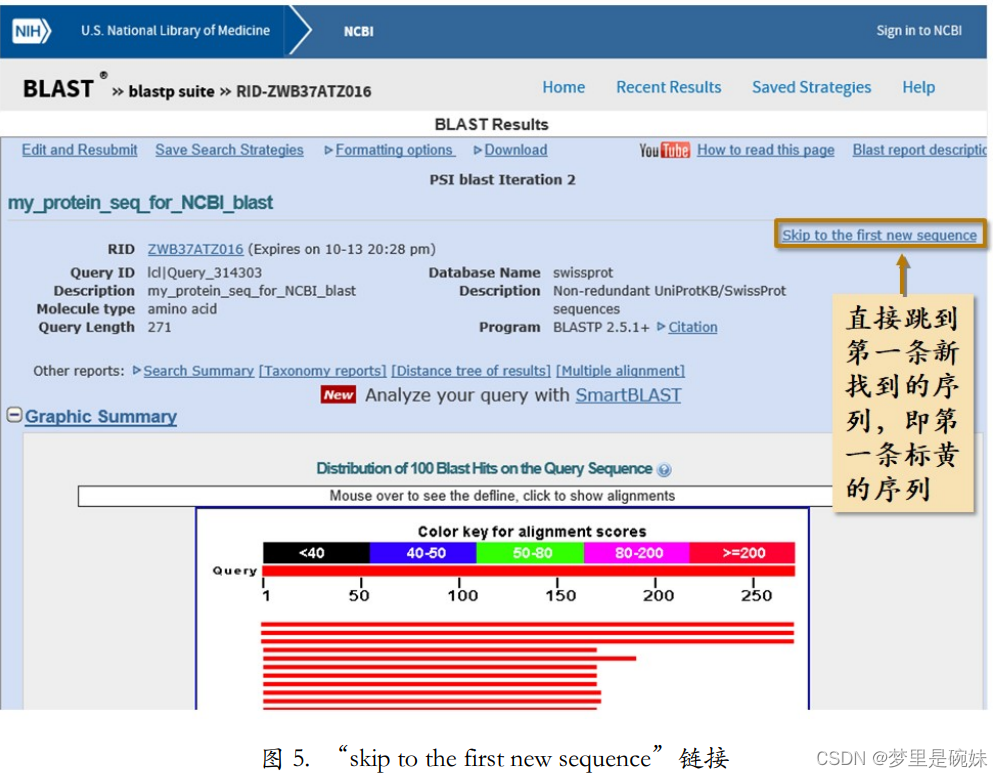
二轮搜索结果里(图 3),红色标记的一列,也就是已在第二轮搜索中被用来创建 PSSM 的序列,大部分都打勾了。但是,再往后面会看到有些标黄的序列没有打勾。这些没有打勾的标黄的序列就是在第二轮搜索中新找到的序列。
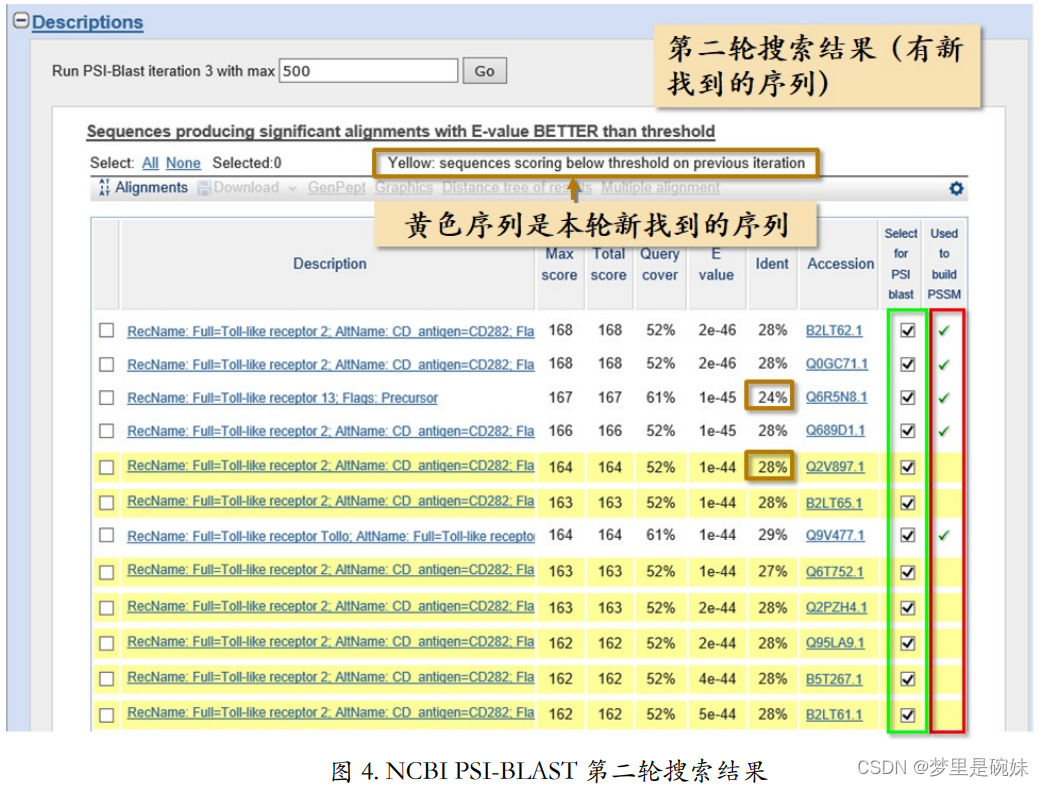
如果想要快速找到本轮搜索新找到的序列,也就是第一条标黄的序列,可以点击结果页 面上方“skip to the first new sequence”链接。
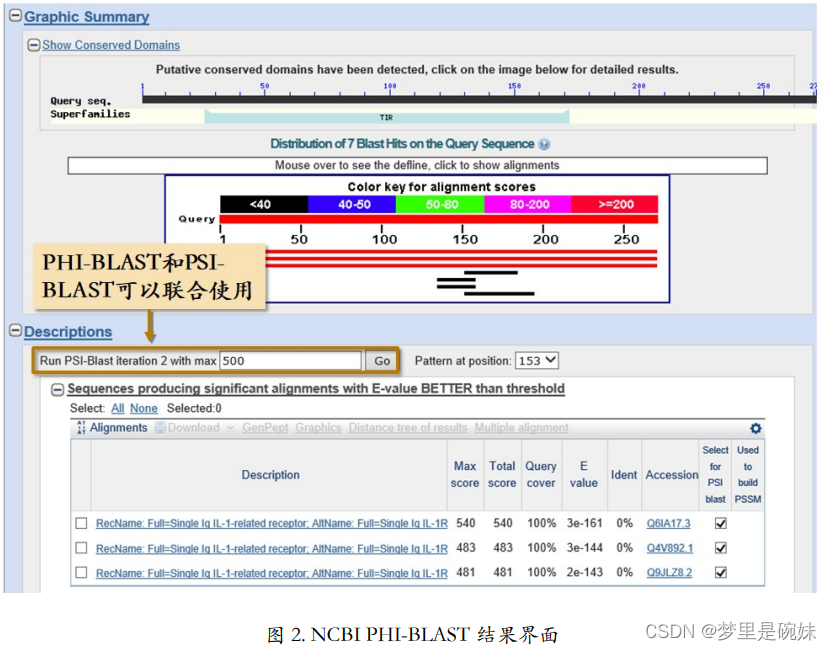
(5)BLAST 搜索:NCBI PHI-BLAST
PHI-BLAST (Pattern-Hit Initiated )和 PSI-BLAST 不同,PSI-BLAST 是撒大网搜索,而 PHI-BLAST 则是精准搜索。PHI-BLAST 能找到与输入序 列相似的并符合某种特征模式的蛋白质序列。
我们需要熟悉一下正则表达式,{}代表除什么以外,[]代表其中之一,x 代表任意字 母,(3,7)代表 3 到 7 个某字符。那么正则表达式{L}GEx[GAS][LIVM]x(3,7)的意思是, 除 L 以外的任意一个字母,紧接 G,再紧接 E,再接一个任意字符,之后是 GAS 中的任意 一个,再接 LIVM 中的任意一个,最后再接 3 到 7 个任意字符。据此,VGEAAMPRI 这条序 列是符合这条正则表达式的,而 VGEAAYPRI 这一条则不符合。这种序列特征模式可能代表 某个翻译后修饰的发生位点,也可以代表一个酶的活性位点,或者一个蛋白质家族的结构域、 功能域等有重要意义的地方。

在 NCBI BLAST 工具的输入页面,当算法选择了PHI-BLAST 之后,会自动出现模式输入框(图 1)。输入正则表达式 S[IVFL]TPS(2)(含义为:一个 S 后面紧接 IVFL 中的任意 一个字母,再接 T,再接 P,再接两个S)。这次搜索找到的相似序列中,只有符合该模式的 才会被作为结果返回。


(6) BLAST 搜索:其他 BLAST
除了前面讲的这三种 BLAST,NCBI 还开发了一个 SMART-BLAST(聪明的 BLAST)。SMART-BLAST 可谓是标准 BLASTp 的简约强化版。操作极其简单,简单到只需要输入序列。
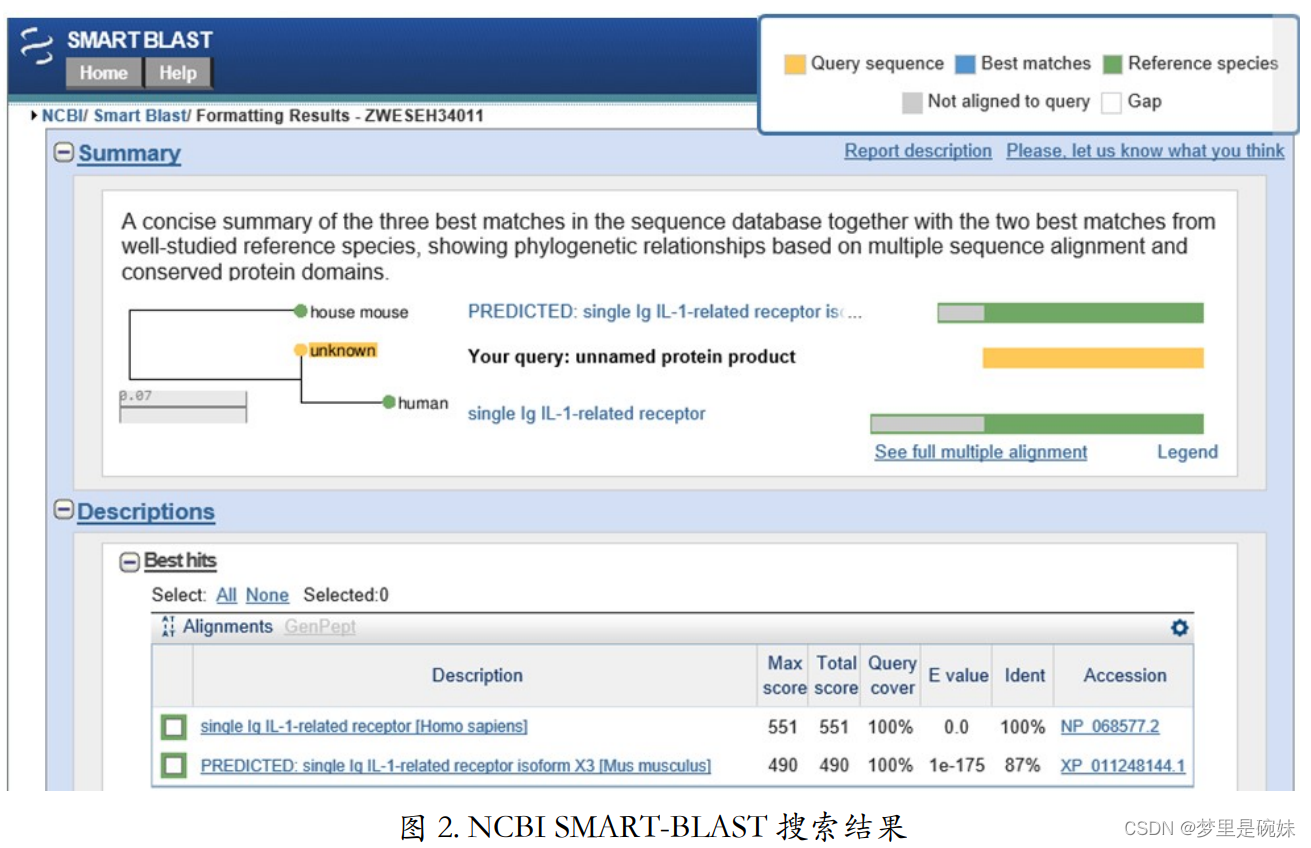
它返回的结果包括数据库中与输 入序列最相似的三条序列,以及研究的最透彻的物种中可以展现一定进化关系的两条相似序 列。图 2 中黄色的是你输入的序列,绿色的是研究的最好的模式物种中与你输入序列相似的 序列。旁边还直接给出了这三条序列的系统发生树。总之,SMART-BLAST 可以帮你从一大 堆结果中挑选出你最想要的。
更多推荐
 已为社区贡献9条内容
已为社区贡献9条内容








所有评论(0)