华大研究院:基于知识且自主多组学解释的基础代理
尽管人工智能(AI)已实现生物信息学工作流的自动化,但生物解释仍较为零散,且往往与机制性洞察脱节。现有AI存在2极分化:❶是缺乏逻辑基础的统计「黑盒」 模型,❷是局限于浅层知识检索的简单代理。为填补这一鸿沟,提出1款整合多组学数据与自适应知识进行生物解释的基础代理BiOmics。BiOmics创新采用双轨架构,包括用于知识接地逻辑推理的协调显式推理空间,以及用于高维关联映射的统一潜在嵌入空间。该架
摘要
尽管人工智能(AI)已实现生物信息学工作流的自动化,但生物解释仍较为零散,且往往与机制性洞察脱节。现有AI存在2极分化:❶是缺乏逻辑基础的统计「黑盒」 模型,❷是局限于浅层知识检索的简单代理。为填补这一鸿沟,提出1款整合多组学数据与自适应知识进行生物解释的基础代理BiOmics。BiOmics创新采用双轨架构,包括用于知识接地逻辑推理的协调显式推理空间,以及用于高维关联映射的统一潜在嵌入空间。该架构支持1种变革性的「检索-推理-预测」范式,可实现跨尺度推理,贯穿从分子变异到疾病表型的生物层级。实证评估表明,BiOmics性能超越当前最先进的AI代理和专用算法,显著提升了生物洞察的粒度与深度。具体而言,BiOmics在挖掘间接致病变异、实现无参考细胞注释以及针对特定数据集优先筛选药物重定位候选物方面展现出独特优势。此外,BiOmics借助其基于推理的知识图谱,进一步拓展了生物实体的解释维度。最终,BiOmics提供了1个多功能工程基础,推动科学人工智能(AI4S)从描述性「数据拟合」向自主、知识驱动的解释转变。
#BiOmics #多组学解释 #知识图谱 #人工智能代理 #生物推理
fangshuangsang@genomics.cn (Shuangsang Fang)
zhangyong2@genomics.cn (Yong Zhang)
liyuxiang@genomics.cn (Yuxiang Li)
结果
BiOmics系统概述
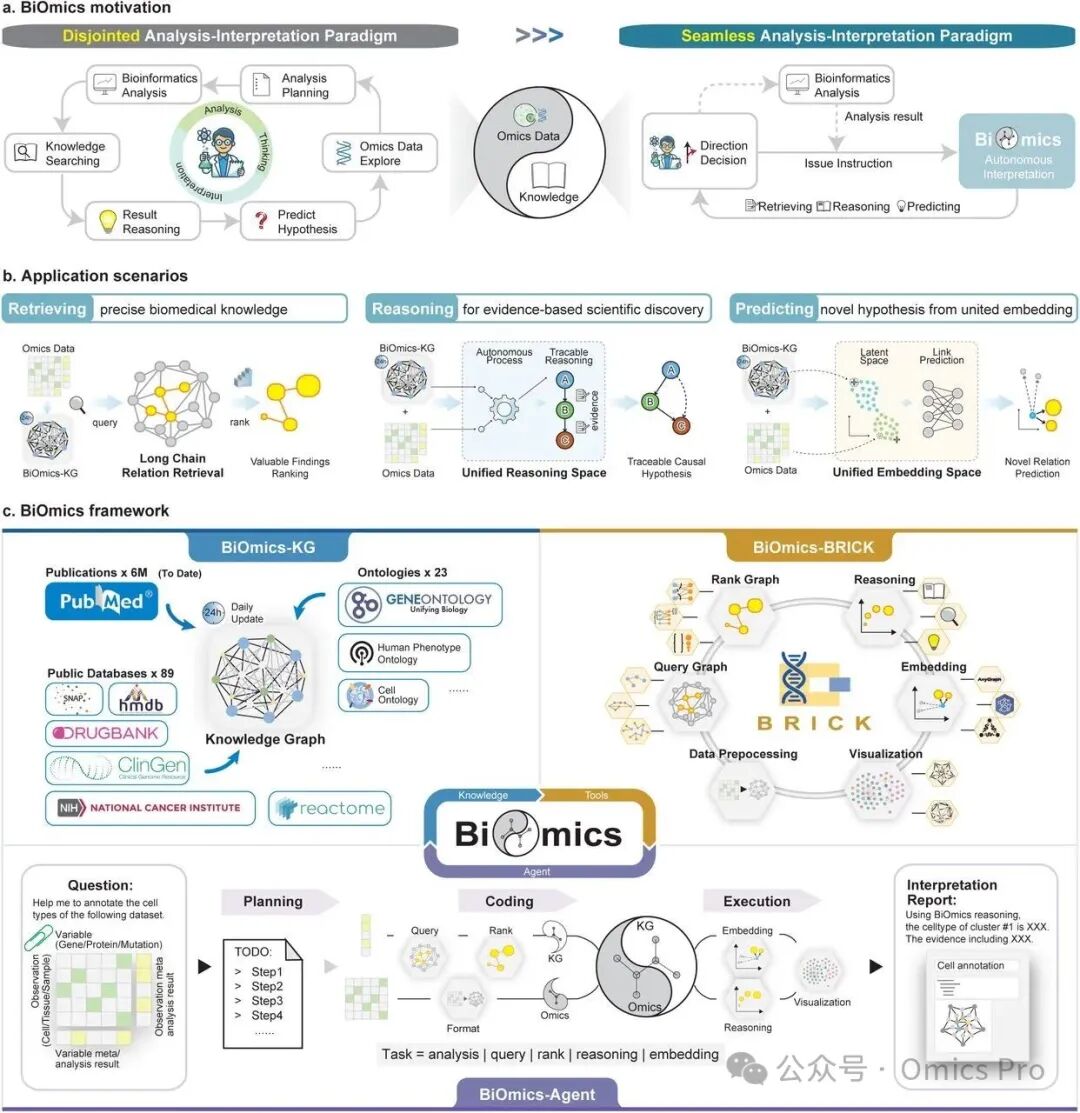
图1 BiOmics系统概述
a. BiOmics 的设计初衷:BiOmics 旨在整合多组学数据与先验知识,将传统多组学知识发现的复杂流程简化为仅通过人机交互即可完成的过程,实现多组学数据的快速自动化挖掘。
b. 应用场景:BiOmics 从3个方面解读多组学数据——检索精准的生物医学知识、推理基于证据的科学发现、基于统一嵌入空间预测新假设。
c. BiOmics 框架:BiOmics 框架由 BiOmics 生物医学知识图谱(左上)、BRICK 工具包(右上)和 BiOmics 多代理系统(底部)组成。BiOmics 知识图谱整合了本体、公共数据库和文献资源;BRICK 是款可配置的模块化工具包,用于整合知识图谱和多组学数据,包含数据预处理、图谱查询、图谱排序、推理、统一嵌入和可视化6个模块;BiOmics 多代理系统能根据用户问题和多组学数据,自动规划解读步骤、调用 BRICK 工具、执行 BRICK 代码并生成解读报告。知识图谱、工具调用与多代理系统的融合,为生物解释任务奠定了基础。
BiOmics为知识驱动的发现和多尺度多组学解释设立新基准
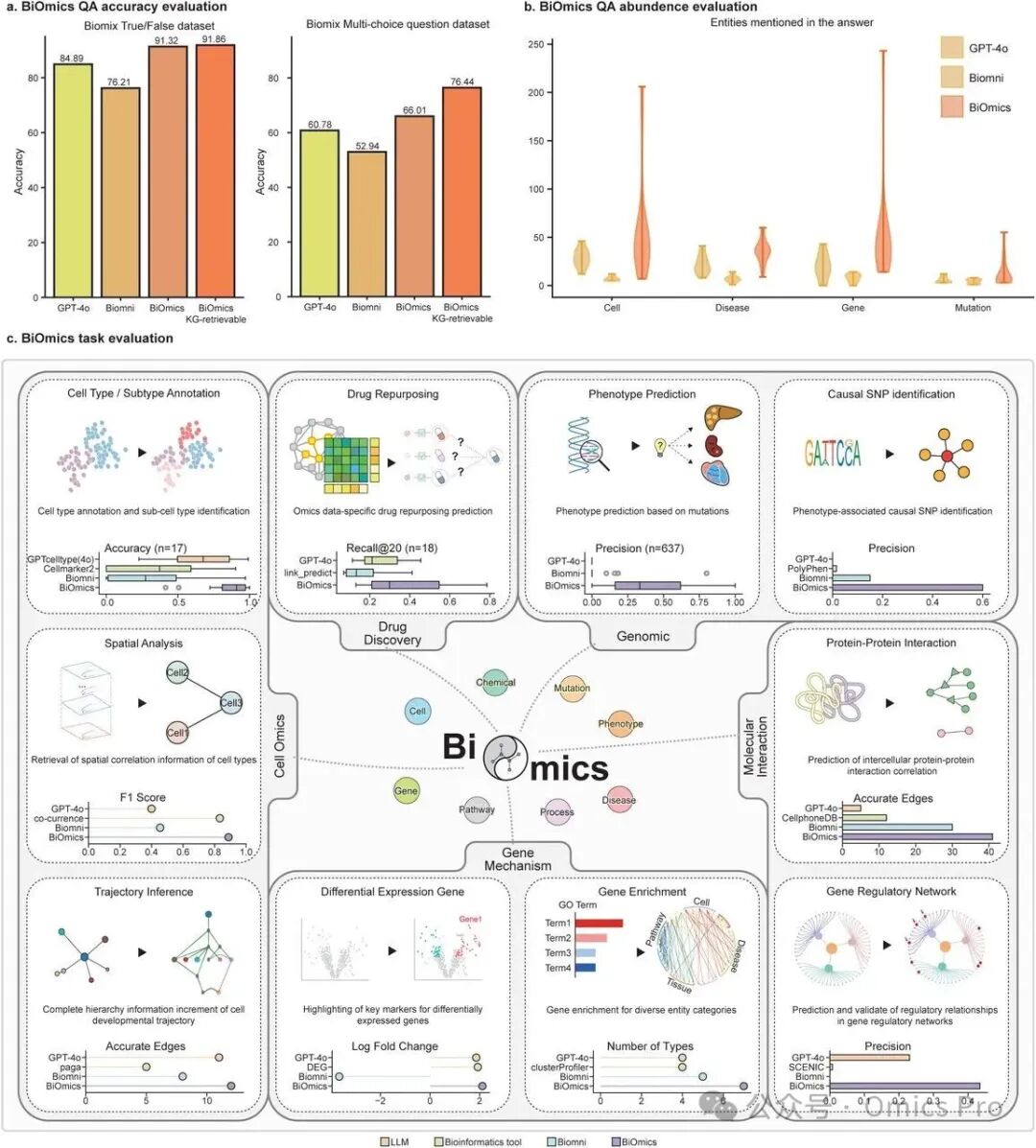
图2 BiOmics性能基准测试
a. BiOmics 问答准确率评估:利用 Biomix 问答数据集,评估各模型在判断题(左图)和多选题(右图)中的准确率。对比模型包括 GPT-4o 和 Biomni,最后1列显示 BiOmics 在可通过知识图谱检索到相关信息的问题中的准确率。
b. BiOmics 问答丰富度评估:纵轴表示答案中提及的实体数量,对比了 GPT-4o、Biomni 和 BiOmics 在可通过知识图谱检索到信息的问题中的表现,涵盖细胞、疾病、基因、变异4类查询对象。
c. BiOmics 生物信息学任务评估:每个子图对应 10 项生物信息学任务,每个子图中包含 BiOmics 与传统分析方法的核心差异说明、凸显 BiOmics 创新点的关键信息,以及与大语言模型(LLM)、传统生物信息学方法和 BiOmni 的性能对比。
BiOmics支持从突变到表型的精准双向推理
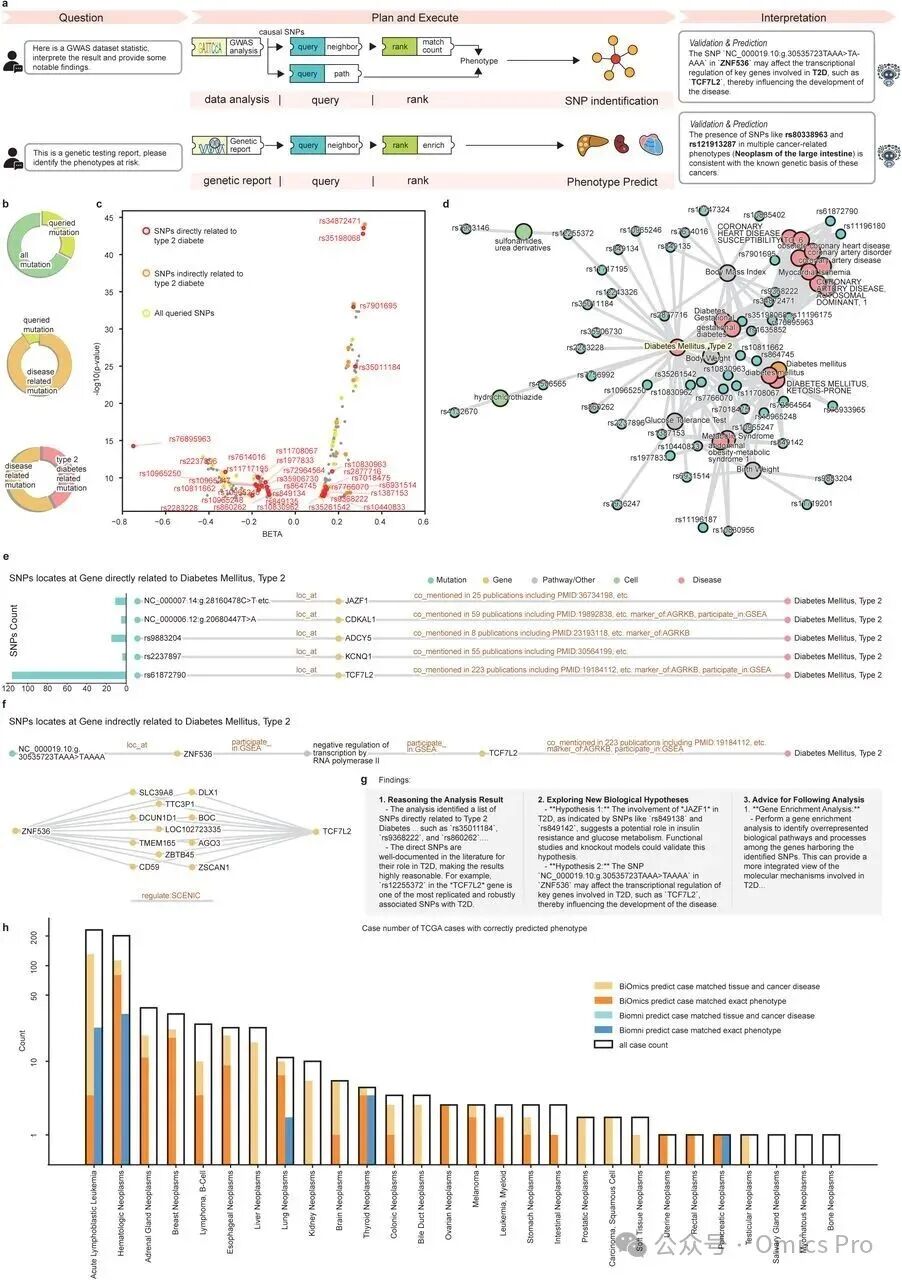
图3 BiOmics支持从基因型到表型的精准双向推理
a. 致病变异识别场景中,BiOmics 优化传统多组学分析的流程。
b. 所有单核苷酸多态性(SNP)中致病变异的占比饼图。
c. 致病变异的散点图:横轴表示全基因组关联研究(GWAS)分析的效应量(BETA);纵轴表示 - log10(p 值);黄色、橙色和红色圆圈分别代表所有查询到的 SNP、与 2 型糖尿病间接相关的 SNP 和与 2 型糖尿病直接相关的 SNP。
d. 查询到的图谱可视化结果。
e. 位于 2 型糖尿病(T2D)相关基因上的 SNP:左图条形图表示每行对应基因上的 SNP 数量。
f. 位于与 2 型糖尿病间接相关基因上的 SNP:上图为 ZNF536 基因与 2 型糖尿病的查询路径,下图为 ZNF536 与 TCF7L2 之间的调控基因。
g. BiOmics 解读报告摘要。h. TCGA 案例表型预测的条形图:横轴为各类疾病类型;纵轴为计数(采用对数压缩)。
BiOmics阐明转录组学中的多尺度基因和细胞图景
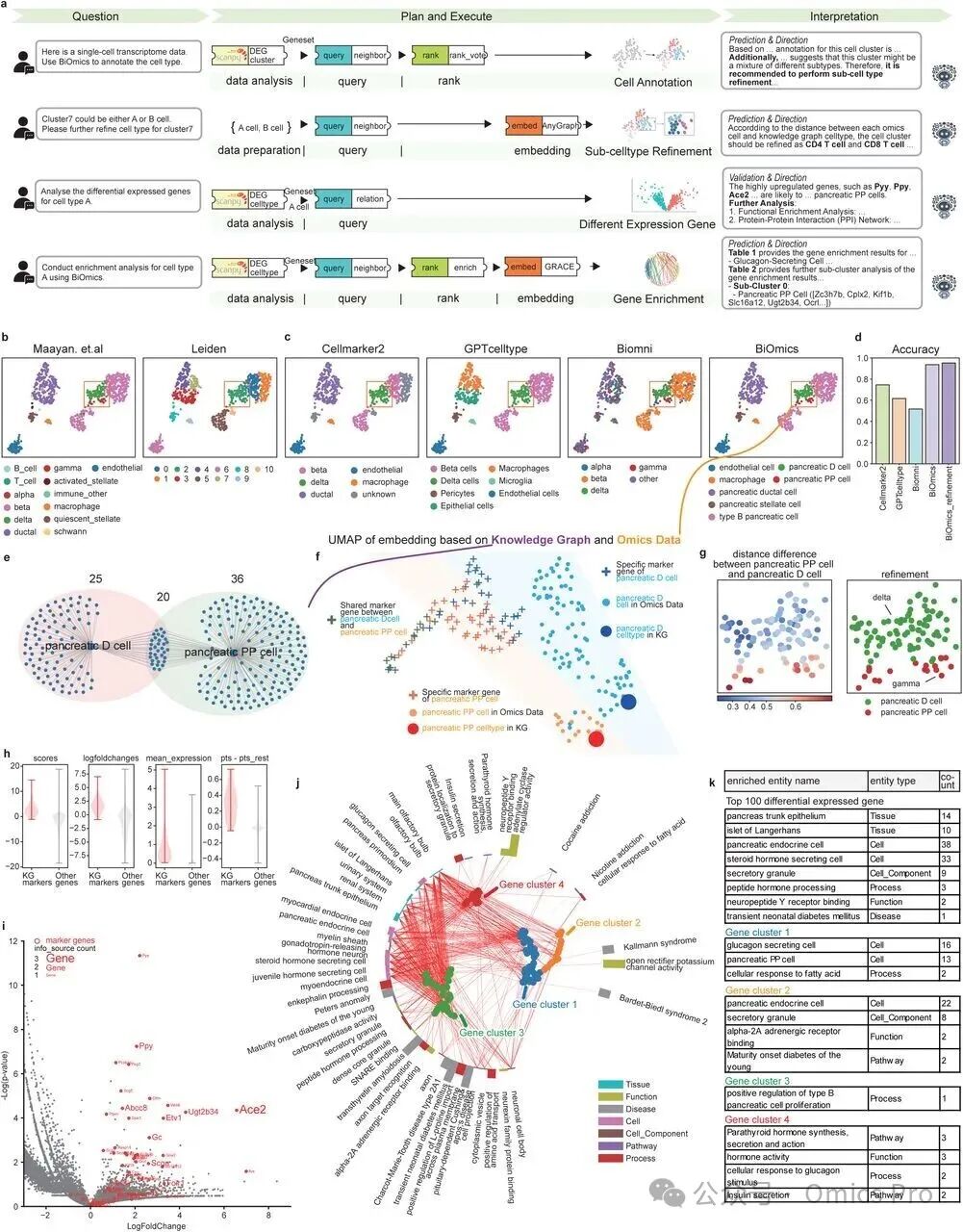
图4 BiOmics提升多组学分析深度
a. BiOmics 优化传统多组学分析的流程,涵盖细胞注释(第1行)、细胞类型细化(第2行)、差异表达基因分析(第3行)和基因富集分析(第4行)4类场景。
b. 小鼠胰岛细胞真实注释结果与聚类标签的 UMAP 图。
c. BiOmics 与其他方法的注释结果 UMAP 图:从左到右依次为 CellMarker2、GPTcelltype、Biomni 和 BiOmics 的注释结果。
d. BiOmics 与其他细胞注释方法的准确率条形图:最后1列为 BiOmics 的细化结果准确率。
e. 胰腺 D 细胞(delta 细胞)和胰腺 PP 细胞(gamma 细胞)查询到的标记基因韦恩图:散点图直接展示查询到的图谱,韦恩图可视化2种细胞类型的标记基因数量。
f. 基于多组学数据集和知识图谱查询结果统一嵌入的 UMAP 图。
g. 胰腺 D 细胞和胰腺 PP 细胞的细化结果 UMAP 图:左图为胰腺 PP 细胞与胰腺 D 细胞的欧氏距离差异,右图为细化结果。
h. 知识图谱标记基因与其他差异表达基因的差异表达基因指标小提琴图:红色代表知识图谱标记基因,灰色代表其他基因;指标包括得分、对数倍变化(logfoldchanges)、平均表达量以及特定聚类与其他细胞聚类间的表达细胞百分比差异。
i. 胰腺 PP 细胞差异表达基因的火山图(已知标记基因高亮显示):横轴为对数倍变化,纵轴为 - log(p 值),每个点代表1个基因,已知标记基因用红色圆圈标注并显示基因名,基因名字体大小代表该基因与胰腺 PP 细胞标记关系的信息来源数量。
j. 胰腺 PP 细胞标记基因的径向富集图:条形颜色区分富集实体类型,条形高度代表富集实体中的基因数量,相邻条形对应的基因集具有更高相似性;中心 UMAP 散点图中每个点代表1个基因,基因颜色代表分组,UMAP 中距离较近的基因表示功能相关性更高,红色线条将每个基因与其对应的富集术语连接。
k. 胰腺 PP 细胞所有差异表达基因(DEG)及各差异表达基因亚群的富集术语表格。
BiOmics增强细胞轨迹的合理预测与验证
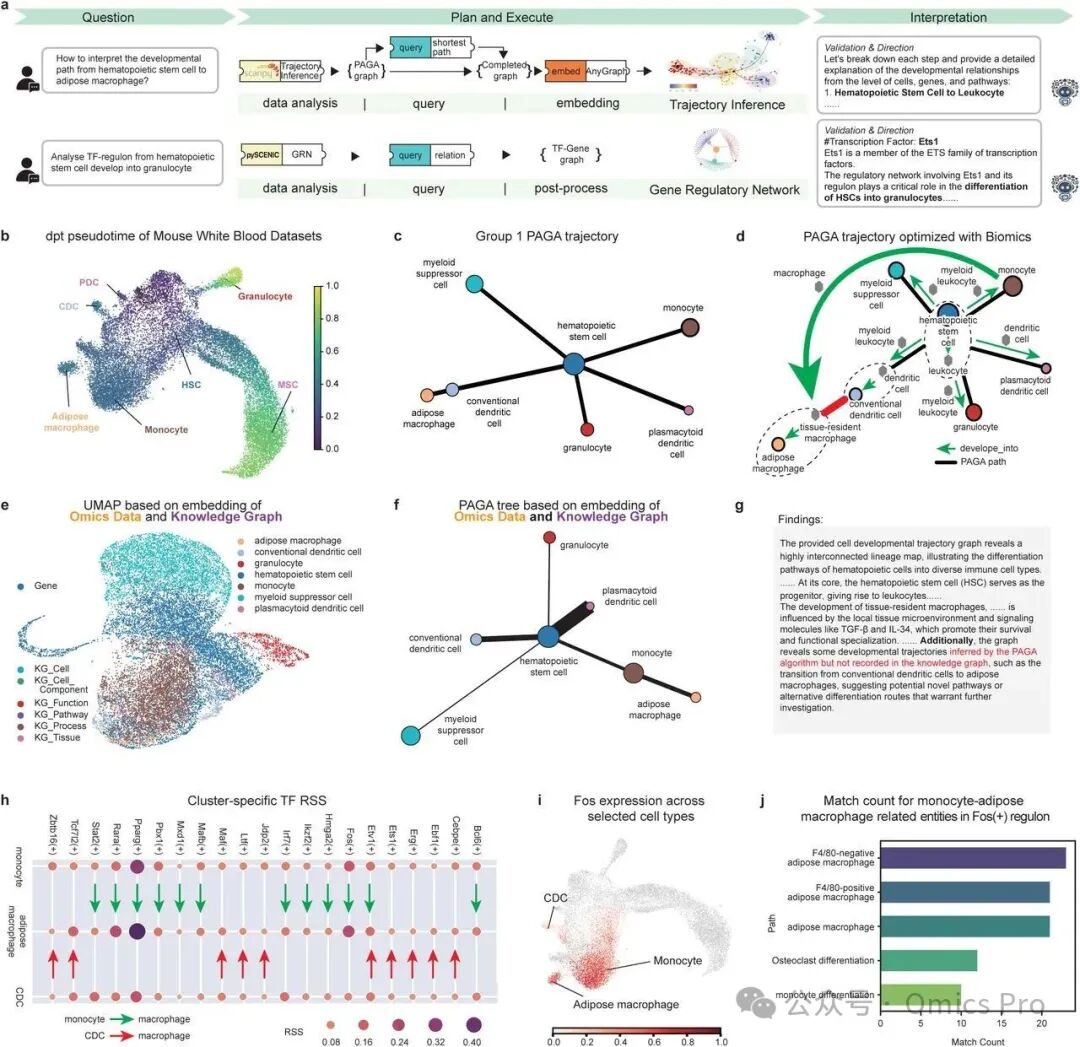
图5 BiOmics引导多组学分析路径
a. BiOmics 优化传统多组学分析的流程,涵盖轨迹分析(第1行)和基因调控网络分析(第2行)2类场景。
b. 以造血干细胞(HSC)为根节点的细胞群 1 的 Dpt 伪时间分析结果 UMAP 可视化。
c. 白细胞(WBC)数据集的 PAGA 轨迹图。
d. BiOmics 利用知识图谱完善 PAGA 图后,骨髓中细胞群(粒细胞、造血干细胞、间充质干细胞、浆细胞样树突状细胞、常规树突状细胞、单核细胞和脂肪组织巨噬细胞)的简化轨迹图:彩色圆形节点代表数据集中的细胞类型,灰色6边形节点代表知识图谱中的细胞类型;绿色路径代表已验证路径,红色路径代表未验证路径。
e. 基于 BiOmics 统一嵌入的知识图谱和多组学数据嵌入 UMAP 图。
f. 基于 BiOmics 知识图谱和多组学数据统一嵌入的细胞群 1 PAGA 轨迹图。
g. 基于 BiOmics 的细胞群 1 轨迹解读内容。
h. 3种细胞类型(单核细胞、常规树突状细胞、脂肪组织巨噬细胞)中聚类特异性转录因子调控子特异性得分(RSS)的气泡图:箭头指示具有更相似 RSS 的细胞类型,绿色箭头指向单核细胞和巨噬细胞,红色箭头指向常规树突状细胞和巨噬细胞。
i. 3种细胞类型(单核细胞、常规树突状细胞、脂肪组织巨噬细胞)中 Fos(+)调控子强度的 UMAP 图。
j. 基于 Fos(+)调控子基因富集的筛选术语。
BiOmics通过动态知识整合推进多组学驱动的药物重定位
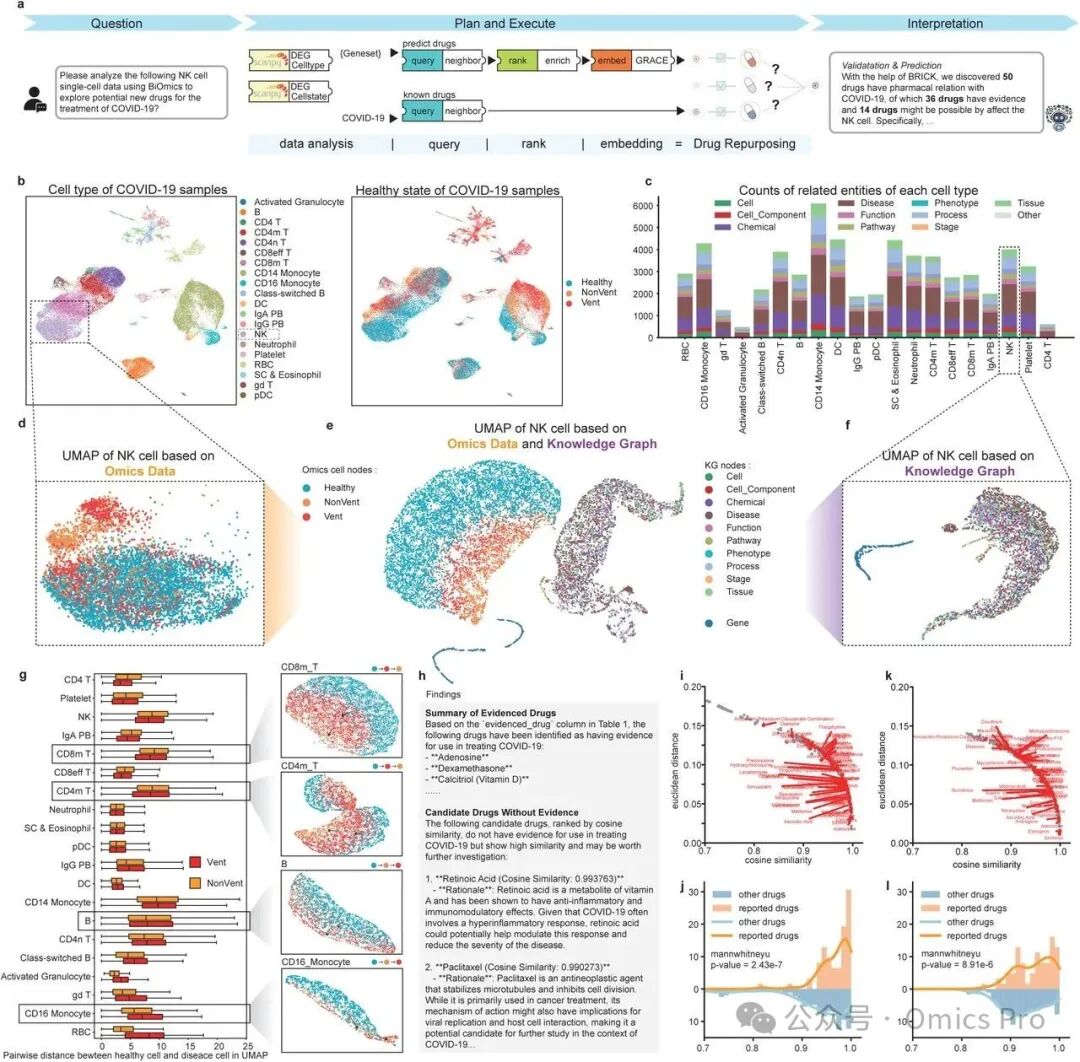
图6 BiOmics助力生物医学发现
a. 药物重定位场景中,BiOmics 优化传统多组学分析的流程。
b. COVID-19 外周血单个核细胞(PBMC)数据集的 UMAP 图:左图按细胞类型着色,右图按健康状态着色。
c. 各细胞类型查询到的实体类型堆叠条形图。
d. 基于多组学数据表达的 NK 细胞 UMAP 图。
e. 经表示学习后,基于多组学数据集和知识图谱查询结果嵌入的 NK 细胞 UMAP 图(左图);经表示学习后,基于知识图谱查询结果嵌入的 UMAP 图(右图)。
f. 基于表示学习后知识图谱查询结果嵌入的 UMAP 图。
g. 基于知识图谱和多组学数据统一嵌入的多组学细胞嵌入结果:左图为各细胞类型中通气(Vent)状态细胞与健康细胞、非通气(NonVent)状态细胞与健康细胞的嵌入距离箱线图;右图为部分细胞类型的嵌入示例。
h. BiOmics 在药物重定位任务中的解读内容。
i. 基于知识图谱和多组学数据的各化合物与 COVID-19 的距离和相似性散点图:横轴为余弦相似度,纵轴为欧氏距离,已报道的 COVID-19 相关化合物用红色标记并标注名称。
j. 基于知识图谱和多组学数据的已报道化合物与其他化合物的余弦相似度直方图:Mann-Whitney U 检验的 p 值为 2.43e-7。
k. 仅基于知识图谱(无多组学数据)的各化合物与 COVID-19 的距离和相似性散点图:横轴为余弦相似度,纵轴为欧氏距离,已报道的 COVID-19 相关化合物用红色标记并标注名称。
l. 仅基于知识图谱(无多组学数据)的已报道化合物与其他化合物的余弦相似度直方图:Mann-Whitney U 检验的 p 值为 1.58e-6。
BiOmics在多组学和蛋白质组学领域展现出强大的通用性
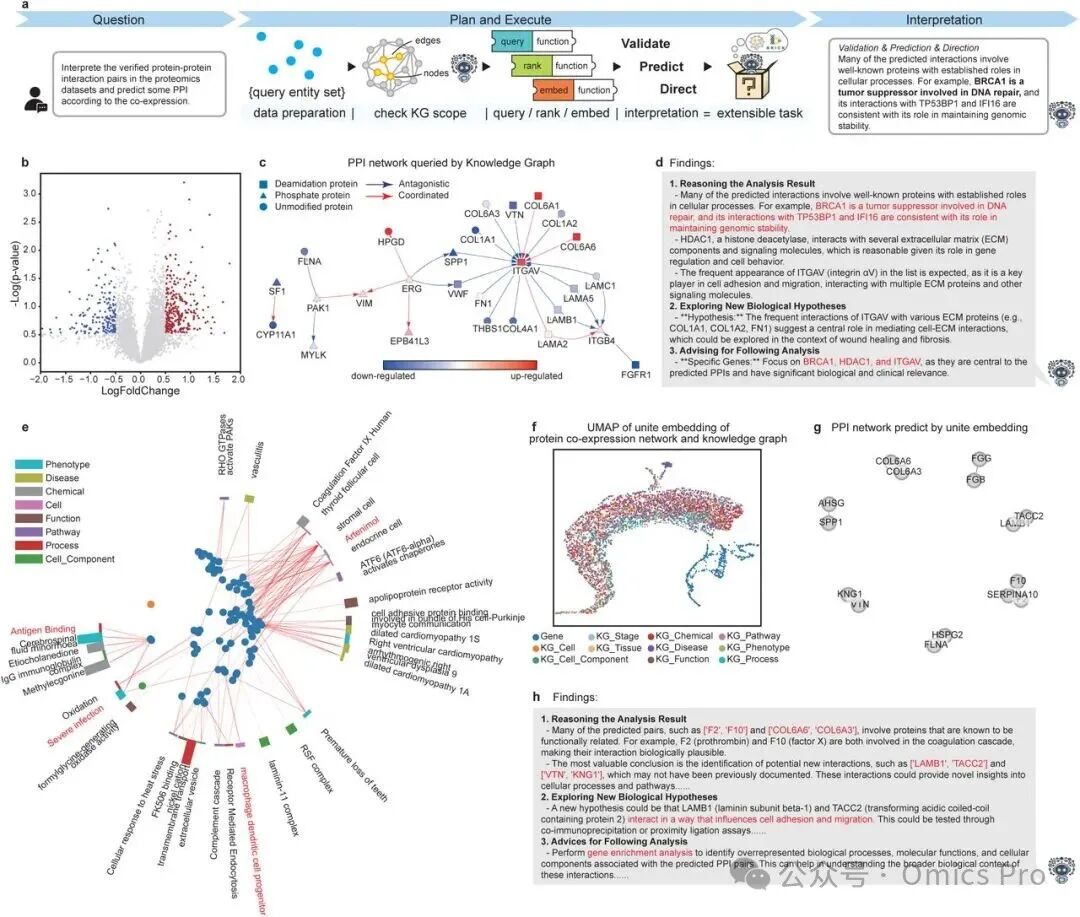
图7 BiOmics具备多组学适应性
a. BiOmics 在其他多组学分析中的可扩展应用流程。
b. 感染样本差异表达蛋白的火山图:横轴为对数倍变化,纵轴为 - log(p 值),蓝色和红色分别标记阳性标记物和阴性标记物。
c. 蛋白质 - 蛋白质相互作用(PPI)网络可视化:圆形、三角形和正方形节点分别代表正常蛋白质、磷酸化蛋白质和糖基化蛋白质;红色和蓝色代表差异表达蛋白质的对数倍变化。
d. 基于 BiOmics 查询结果的 PPI 任务解读内容。
e. 差异表达蛋白标记物的径向富集图:条形颜色区分富集实体类型,条形高度代表富集实体中的基因数量。
f. 经表示学习后,基于蛋白质组学数据集和知识图谱查询结果统一嵌入的 UMAP 图。
g. 基于蛋白质组学数据集统一嵌入的预测 PPI 网络。
h. 基于 BiOmics 统一嵌入结果的 PPI 任务解读内容。
数据
BiOmics整合的多组学数据分为2个不同子集:用于基准测试的评估数据和用于案例研究的分析案例数据。评估子集相关数据来源如下:Biomix多选题和判断题数据集取自Hugging Face平台(BiomixQA,https://huggingface.co/datasets/kg-rag/BiomixQA);用于细胞注释的组织特异性cellxgene数据集为内部构建;支持药物重定位分析的数据集为GSE150728;用于表型预测的是639个可公开获取的 TCGA 案例(含对应标识符);用于致病变异识别的是「All of Us 2024 T2D WGS」数据集;用于差异表达基因和基因富集分析的是GSM2230761数据集;用于轨迹推理和基因调控网络(GRN)研究的是GSE228590数据集;用于空间分析的是PRJNA668433数据集;用于蛋白质-蛋白质相互作用研究的是PXD008079数据集。分析案例子集包括:「All of Us 2024 T2D WGS」数据集(人类/2型糖尿病)、GSM2230761数据集(小鼠/胰岛)、GSE228590数据集(小鼠/胚胎白细胞)、GSE150728数据集(人类/外周血单个核细胞)、PRJNA668433数据集(小鼠/睾丸)以及PXD008079数据集(人类/胎盘)。
详细总结

思维导图(mindmap)
3大核心组件详解

参考
BiOmics: A Foundational Agent for Grounded and Autonomous Multi-omics Interpretation, https://www.biorxiv.org/content/10.64898/2026.01.17.699830v1
注:AI辅助创作,如有错误欢迎指出。内容仅供参考,不构成任何建议。
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容







所有评论(0)