邻近标记酶谱解析:基于BioID家族「BioID/TurboID/AirID/ultraID」和过氧化物酶家族「APEX/APEX2」的深度对比
在邻近标记酶中,BioID家族与过氧化物酶家族在酶学特性、标记效率与应用场景中的差异,常令研究者难以抉择。今天,我们将基于权威文献,对这些标记酶进行详尽的酶学特性剖析,以及应用场景的深度探讨,全方位解析如何为特定的生物医学问题选择最合适的工具。
引言
邻近标记(PL)技术通过基因编码的酶,在活细胞原位将标签共价连接到邻近蛋白,无需直接物理结合即可捕获弱相互作用、瞬时互作及空间邻近蛋白,显著提升了蛋白质组学研究在原位空间图谱绘制中的分辨率,为解析细胞器接触位点、无膜细胞器(如相分离液滴)及动态信号转导复合物提供了高分辨率技术支撑。
面对邻近标记酶,BioID家族(BioID/BioID2/TurboID/AirID/ultraID)与过氧化物酶家族(APEX/APEX2)各有千秋,其酶学特性、标记效率与应用场景的差异,常令研究者难以抉择。
为此,我们将基于PubMed、Web of Science、Protocols.io等权威数据库的文献,对这些常用和新兴的标记酶进行详尽的酶学特性剖析,以及应用场景的深度探讨,全方位解析“如何为特定的生物医学问题选择最合适的工具”。
01 邻近标记的生化机理与酶学分类
邻近标记技术依据催化反应化学性质及所需底物,主要分为两大类:生物素连接酶衍生标记系统和过氧化物酶衍生标记系统,理解两类酶的核心生化逻辑是实验设计与结果解读的基础。
1. 生物素连接酶家族:从BirA到TurboID的定向进化
该类技术源于大肠杆菌的生物素连接酶BirA,其天然功能是将生物素与ATP结合生成高活性、不稳定的生物素酰-5'-腺苷酸中间体,并特异性转移至乙酰辅酶A羧化酶(BCCP)亚基的特定赖氨酸残基。通过定向改造BirA的结构与动力学特性,衍生出系列功能变体:
① BioID:经R118G单点突变,使中间体“泄漏”并标记邻近蛋白(伯胺反应),标记半径约10nm,但催化效率极低,需18-24小时标记,仅适用于稳态复合物研究。
② BioID2:删除BirA的N端DNA结合结构域,分子量从35kDa降至27kDa,减少空间位阻,优化生物素亲和力,仍需16小时以上标记,时间分辨率无突破。
③ TurboID/miniTurbo:经定向进化获得的高活性变体,催化效率比BioID高100倍,10分钟即可获得强信号,时间分辨率达“分钟级”;miniTurbo(28kDa)体积更小,背景噪音低但稳定性较差。二者的不足是高活性易导致内源生物素消耗,引发背景标记与细胞毒性。
2. 过氧化物酶家族:自由基介导的秒级标记
与生物素连接酶的温和反应不同,过氧化物酶系统(APEX/APEX2)源自抗坏血酸过氧化物酶,经改造后采用激进的自由基化学机制催化苯酚衍生物氧化。
▶ 反应机理:在过氧化氢(H2O2)存在下,催化生物素-苯酚生成生物素-苯氧基自由基,该自由基半衰期<1毫秒,扩散距离<20nm,快速与邻近蛋白的酪氨酸、色氨酸等富电子残基共价交联。
▶ 核心优势:标记仅需45-60秒,具备“快照”式秒级时间分辨率,是瞬态过程(如神经突触传递)研究的关键工具。
▶ 标记偏好差异:与生物素连接酶家族(主要标记赖氨酸,富集RNA加工、蛋白定位相关蛋白)相比,APEX2侧重标记酪氨酸,在代谢通路相关蛋白富集中更具优势,二者形成蛋白质组覆盖互补。
3. 新兴酶学工具:AirID、ultraID与其他变体
随着邻近标记技术的普及,研究者们不断开发新型酶以解决BioID(慢)和TurboID(背景高、毒性大)之间的矛盾,力求在活性、特异性和生物兼容性之间找到最佳平衡点。
① AirID:祖先序列重构的低毒高效工具
▶ 设计思路:未依赖大肠杆菌BirA突变,而是通过宏基因组数据与祖先序列重构算法,推导出BirA家族的祖先序列,创新设计逻辑。
▶ 核心特性:分子量约35kDa,标记动力学介于BioID与TurboID之间(1-3小时即可获得强信号);生物兼容性优异,长时间表达与标记对细胞毒性极低,适配小鼠、发育生物学模型等长时程体内实验;低生物素浓度下表现优于BioID,融合蛋白稳定性高于miniTurboID,减少假阴性。
② MicroID与UltraID:极致小型化的高性能变体
▶ 结构突破:MicroID通过截去BioID2的C端非结构域并引入突变,分子量降至20kDa以下;UltraID(约19kDa)在MicroID基础上新增R40G/L41P突变,进一步优化性能。
▶ 核心优势:UltraID兼具TurboID的速度(10分钟标记)与BioID2的低背景特性,19kDa的小巧体积使其成为膜蛋白、核孔复合物等空间受限区域研究的理想选择,部分膜结合蛋白相互作用研究中,信噪比优于TurboID和BASU。
③ BASU与Pup-IT:其他技术路径的探索
▶ BASU:源自枯草芽孢杆菌的生物素连接酶,早期文献声称动力学极快(1分钟),但后续独立研究证实其表现不稳定,需较长标记时间才能达到BioID水平,应用存在争议;
▶ Pup-IT:基于原核生物类泛素化系统的标记技术,利用PafA酶将小蛋白Pup连接到邻近蛋白上,无内源性背景(哺乳动物细胞无Pup系统),但Pup分子量较大可能干扰底物识别,操作流程较生物素系统更复杂。
02 特殊应用场景与案例分析
1. 神经科学:突触蛋白组与活体脑图谱
神经元极性结构复杂,突触间隙蛋白质组难以通过传统生化方法分离。
▶ APEX2的应用:对培养神经元突触间隙进行1分钟标记,成功鉴定神经递质受体复合物的动态变化;
▶ TurboID的在体突破:通过立体定位注射Cre依赖性TurboID的AAV病毒或构建Rosa26-TurboID转基因小鼠,实现特定脑区、特定细胞类型(如多巴胺能神经元、星形胶质细胞)的在体蛋白质组标记,喂食含生物素饲料/饮水7天即可获得强信号,揭示不同脑区星形胶质细胞异质性。
2. 植物生物学:温度是关键
BioID最适温度为37°C,而拟南芥等模式植物生长温度多为22°C,低活性限制了其在植物研究中的应用。
▶ TurboID的革命:在室温(25°C)及更低温度下仍保持极高活性,成为植物领域首选工具;
▶ 案例:利用TurboID标记植物免疫受体NLR(如烟草N蛋白),成功鉴定病原菌诱导下的动态互作网络,这是传统BioID无法实现的。
3. 细胞器接触位点:Split系统的精妙应用
内质网-线粒体接触位点是脂质转运和钙信号传导的枢纽,结构脆弱易被传统分离方法破坏。
▶ Split-TurboID方案:将TurboID拆分为无活性的N端和C端片段,分别融合到内质网膜蛋白和线粒体外膜蛋白上,仅在两者物理接触的纳米尺度界面上,酶片段重组恢复活性,可特异性标记并纯化接触位点蛋白质组,排除非接触区域干扰。
4. 病毒学:宿主-病原体相互作用
病毒感染过程中,病毒蛋白与宿主蛋白的相互作用多为瞬时且发生于特定复制区室。
▶ HIV-1研究:利用BioID融合的HIV-1蛋白,揭示病毒核输入过程中与宿主核孔蛋白的相互作用机制;
▶ AirID的应用:低细胞毒性且耐受抑制剂的特性,使其被用于筛选阻断病毒蛋白-宿主因子相互作用的小分子药物,为药物筛选提供新平台。
03 综合比较与选择指南:决策矩阵
面对如此众多的酶,研究者应如何做出选择?以下是基于关键实验参数的决策矩阵。
表1. 主流邻近标记酶特性详表
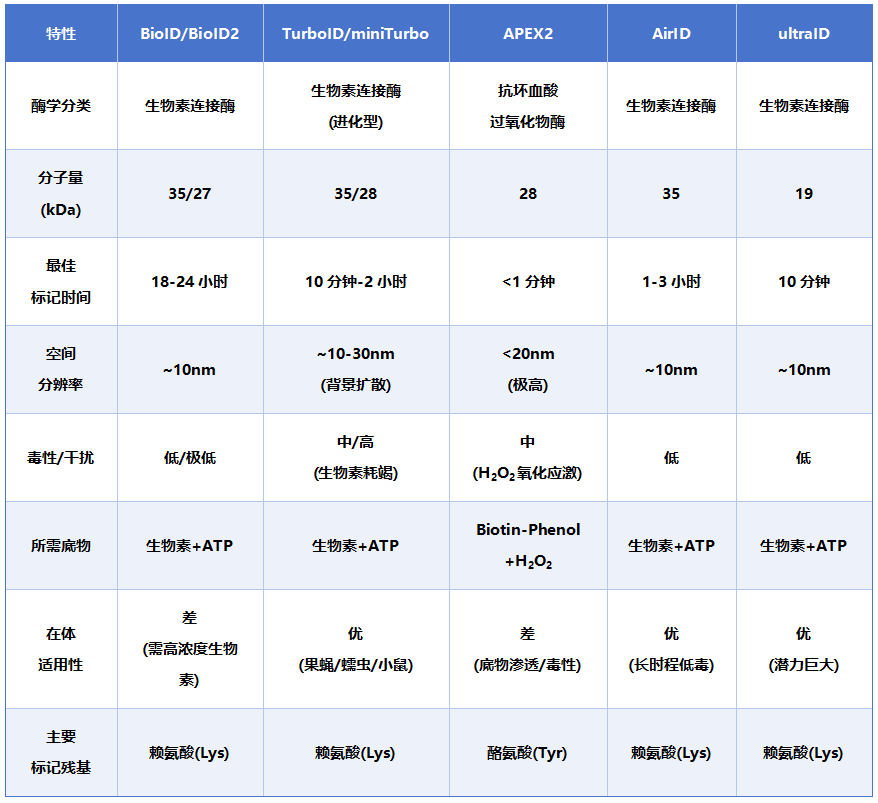
04 场景导向的选择建议
1. 需要极高的时间分辨率(秒级):
▶ 首选:APEX2。适用于捕捉GPCR激活、离子通道开关或快速囊泡运输。
▶ 警告:需确认细胞能耐受H2O2,且Biotin-Phenol能渗透到目标区域(植物或厚组织切片慎用)。
2. 活体动物或植物实验:
▶ 首选:TurboID。其高活性克服了体内生物素分布不均和低温(植物)的限制。
▶ 备选:AirID。如果实验周期长(如数周的发育观察),AirID的低毒性更具优势。
3. 对融合蛋白大小敏感(如标记小蛋白或病毒衣壳):
首选:ultraID或BioID2。小于20kDa的ultraID是目前的最小选择,能最小化空间位阻。
4. 关注背景控制与特异性(如低丰度蛋白):
▶ 首选:miniTurbo或ultraID。虽然TurboID最快,但其高背景可能淹没低丰度信号。miniTurbo和ultraID提供了更好的信噪比。
▶ 策略:配合3-4小时的生物素耗竭预处理。
5. 研究特定的亚细胞接触结构:
首选:Split-TurboID。相比Split-BioID,其信号强度更高,能够在合理的时间内(<4小时)富集到接触位点蛋白,避免了过长时间标记导致的非特异性扩散。
05 结论与展望
邻近标记技术已经从一个单纯的“互作伙伴发现工具”演变为解析细胞空间组织的“显微镜”。从BioID的温和但缓慢,到TurboID的激进与高效,再到APEX2的精准快照以及AirID/ultraID的理性设计,工具箱的丰富使得研究者可以根据生物学问题的具体需求(时间尺度、空间尺度、生理环境)量体裁衣。
未来的技术演进将聚焦于三个方向:一是化学探针的革新:开发具有可光解、可点击化学特性的新型生物素衍生物,以进一步提高富集的特异性;二是酶的定向进化:创造出更小、更快且对内源生物素不敏感的“正交”酶系统;三是与单细胞测序及超分辨成像的融合:实现从单细胞维度同时解析转录组与空间蛋白质组的多模态分析。
对于生物医学研究者而言,掌握这些工具不仅意味着能画出一张更全的“蛋白网络图”,更意味着能动态地看到这些网络如何在疾病、药物或发育过程中重编程,以此揭示生命的分子机制。
⬇️⬇️⬇️
研究动态、瞬时、弱相互作用的最佳工具—PL-MS蛋白邻近表达筛选解决方案
基于邻近标记技术联合质谱推出PL-MS蛋白邻近表达筛选解决方案,提供从细胞构建培养、亲和富集实验到质谱高通量筛选(阶段可选):①目标序列获取和质粒克隆构建、②基因编辑和细胞系构建、③细胞培养和临近标记、④生物素标记蛋白的亲和纯化富集、⑤蛋白提取和酶解、⑥质谱检测和⑦数据分析和互作网络构建。
▶ 弱/动态/瞬时互作:基于空间临近标记的特殊原理,可以动态捕获更多弱相互作用和瞬时相互作用的蛋白;
▶ 高亲和性:链霉亲和素的亲和效价强,且无需严格保证非变性条件,可以捕获更多膜蛋白的互作;
▶ 避免阴性结果:基于质谱定量数据进行无偏见的高通量筛选,可以获得大量蛋白相互作用信息,避免阴性结果;
▶ 精准筛选:基于定量差异、蛋白功能注释、互作网络核心节点等多维度分析,提供候选临近表达蛋白的清单;
▶ 原位精准表达:可选目标基因的原位标签表达,保留基因天然表达模式,提升研究科学性。
参考文献
1. Bosch JA, Chen CL, Perrimon N. Proximity-dependent labeling methods for proteomic profiling in living cells: An update. Wiley Interdiscip Rev Dev Biol. 2021;10(1):e392. doi:10.1002/wdev.392
2. Dumrongprechachan V, Salisbury RB, Soto G, Kumar M, et al. Cell-type and subcellular compartment-specific APEX2 proximity labeling reveals activity-dependent nuclear proteome dynamics in the striatum. Nat Commun. 2021;12(1):4855. Published 2021 Aug 11. doi:10.1038/s41467-021-25144-y
3. Xu Y, Fan X, Hu Y. In vivo interactome profiling by enzyme-catalyzed proximity labeling. Cell Biosci. 2021;11(1):27. Published 2021 Jan 29. doi:10.1186/s13578-021-00542-3
4. Pfeiffer CT, Paulo JA, Gygi SP, Rockman HA. Proximity labeling for investigating protein-protein interactions. Methods Cell Biol. 2022;169:237-266. doi:10.1016/bs.mcb.2021.12.006
5. Kido K, Yamanaka S, Nakano S, et al. AirID, a novel proximity biotinylation enzyme, for analysis of protein-protein interactions. Elife. 2020;9:e54983. Published 2020 May 11. doi:10.7554/eLife.54983
6. Schaack GA, Sullivan OM, Mehle A. Identifying Protein-Protein Interactions by Proximity Biotinylation with AirID and splitAirID. Curr Protoc. 2023;3(3):e702. doi:10.1002/cpz1.702
7. Rayaprolu S, Bitarafan S, Santiago JV, et al. Cell type-specific biotin labeling in vivo resolves regional neuronal and astrocyte proteomic differences in mouse brain. Nat Commun. 2022;13(1):2927. Published 2022 May 25. doi:10.1038/s41467-022-30623-x
8. Li Y, Zhang Y, Dinesh-Kumar SP. TurboID-Based Proximity Labeling: A Method to Decipher Protein-Protein Interactions in Plants. Methods Mol Biol. 2024;2724:257-272. doi:10.1007/978-1-0716-3485-1_19
9. Yamada K, Yamanaka S, Yamakoshi H, et al. In-cell proximity target validation methods for heterobifunctional molecules with CRBN- or VHL-binder using AirID. Commun Biol. 2025;8(1):1323. Published 2025 Aug 30. doi:10.1038/s42003-025-08761-x
10. Kido K, Yamanaka S, Nakano S, et al. AirID, a novel proximity biotinylation enzyme, for analysis of protein-protein interactions. Elife. 2020;9:e54983. Published 2020 May 11. doi:10.7554/eLife.54983
更多推荐
 已为社区贡献7条内容
已为社区贡献7条内容







所有评论(0)