学习通过皮层发育连续性迁移实现全生命周期脑解剖对应/文献速递-基于人工智能的医学影像技术
2026.1.26本文提出了一种新颖的皮层发育连续性(CDC)迁移学习框架,通过利用大脑不同年龄组间的发育连续性,在数据量最大的年龄组(成人组)上训练模型,并沿皮层发育轨迹逐步迁移到其他年龄组,有效解决了皮层折叠模式个体差异大、不同神经发育阶段异质性强以及早期发育阶段数据稀缺等挑战,实现了跨生命周期大脑精细解剖结构的鲁棒对应,并能有效捕捉群体特异性模式和保留个体差异。Title题目01Learni
2026.1.26
本文提出了一种新颖的皮层发育连续性(CDC)迁移学习框架,通过利用大脑不同年龄组间的发育连续性,在数据量最大的年龄组(成人组)上训练模型,并沿皮层发育轨迹逐步迁移到其他年龄组,有效解决了皮层折叠模式个体差异大、不同神经发育阶段异质性强以及早期发育阶段数据稀缺等挑战,实现了跨生命周期大脑精细解剖结构的鲁棒对应,并能有效捕捉群体特异性模式和保留个体差异。
Title题目
01
Learning lifespan brain anatomical correspondence via cortical developmental continuity transfer
学习通过皮层发育连续性迁移实现全生命周期脑解剖对应
文献速递介绍
02
人类大脑皮层在早期发育过程中形成复杂的折叠模式,其正常生长和折叠对大脑回路和功能组织至关重要。皮层折叠异常与多种脑功能障碍、认知缺陷和精神疾病密切相关,因此理解皮层折叠模式的发展是重要研究课题。然而,皮层折叠模式在个体间差异显著,在不同神经发育阶段也表现出巨大的异质性。建立可靠的全生命周期解剖对应关系,即在不同神经发育阶段的个体大脑间建立纵向跨受试者对应,是揭示大脑发育轨迹或评估脑解剖-功能关系纵向研究的必要前提。传统脑图谱方法可能忽略个体差异。研究人员已探索使用更精细的沟回皮层地标,如沟底和不可分割的沟根来提高对齐精度。最近,三铰链脑回(3HG)被识别为一种新的精细尺度皮层折叠模式地标,它在多个灵长类物种中进化保守,在人类大脑中普遍存在,并兼具共同和个体模式。然而,现有的3HG对应方法主要针对单一成人队列,应用于全生命周期研究时,要么混合所有阶段训练单一模型(忽略异质性),要么为每个阶段独立训练(需要大量数据且未能利用跨阶段固有关系),而早期发育阶段数据获取尤其困难。受迁移学习成功的启发,本文旨在设计一种新颖的迁移策略,利用不同年龄组间的固有关系,促进共享模式在神经发育阶段间的传递,提出了一种皮层发育连续性(CDC)迁移框架。该框架从数据量最大的成人组训练模型,然后沿着皮层发育轨迹逐步迁移到其他阶段,并设计了新的损失函数以确保共同模式的提取和保留,同时捕捉阶段特异性新模式。
Aastract摘要
02
在整个生命周期内识别人类大脑的解剖对应关系是研究大脑发育和老化的重要前提。然而,由于皮层折叠模式巨大的个体差异、不同神经发育阶段的异质性以及神经影像数据的稀缺性,在更精细的尺度上推断可靠的全生命周期解剖对应关系十分困难。为解决此问题,本研究利用大脑皮层的发育连续性,提出了一种新颖的迁移学习策略:模型首先在样本量最大的成人年龄组上从头开始训练,然后遵循皮层发育轨迹,逐步迁移并适应其他年龄组。本研究设计了一种新的损失函数,以确保在迁移过程中能够提取并保留共同模式,同时捕捉特定年龄组的新模式。该框架使用涵盖四个生命周期年龄组(从34孕周到青年成人)的1000多个大脑的多数据集进行评估。实验结果表明:1) 所提出的迁移策略能够显著提高训练样本数量非常有限的群体(例如早期神经发育阶段)的模型性能;2) 借助迁移学习,我们能够稳健地推断不同神经发育阶段不同大脑之间复杂的“多对多”解剖对应关系。
Method方法
03
CDC框架基于皮层持续发育的原理,利用不同年龄组间的固有关系,促进共享模式在神经发育阶段间的传递。该框架采用分步学习过程,首先在样本量最大的年龄组上训练,然后将学到的知识沿着皮层发育轨迹迁移到其他年龄组。研究首先介绍了3HG的识别过程。3HG识别包括五个步骤:首先,根据脑回高度将大脑皮层分割为脑回顶部和脑沟底;其次,使用树形行进算法连接脑回顶点的所有顶点,形成图表示;然后,修剪短于预定义阈值的冗余分支,保留主干形成GyralNet,3HG被定义为GyralNet上具有三个分支的连接点;最后,每个半球皮层被划分为75个感兴趣区域(ROI),每个3HG属于一个特定ROI。在3HG解剖嵌入方面,将3HG网络表示为图G=(A,X),其中A是邻接矩阵,X是one-hot编码矩阵。为了捕获不同跳数(l-hop)的间接连接,l-hop特征Fli被定义为包含0-hop到l-hop的连接模式。然后,一个自监督嵌入框架用于学习每个ROI的有意义的特征嵌入,其编码和解码过程通过一系列非线性函数和可学习的ROI嵌入矩阵E实现。目标函数采用MSE损失和两个解码层的加权和。CDC迁移框架在此基础上,首先使用成人组数据从头训练模型,学习ROI嵌入E。然后,遵循皮层发育轨迹,将E迁移并适应到其他年龄组。迁移过程中,E中的共同模式被提取并与新数据中学到的特定年龄组新模式相结合。为指导此过程,提出了两个正则化项:Lspe旨在确保两个组特异性嵌入之间保持最小距离,而Lcom则鼓励共同部分与两个组的ROI嵌入保持一致,从而在保留组特异性模式的同时促进共同知识的传递。评估方法包括三个方面:评估ROI嵌入的有效性,通过3HG的l-hop连接强度来衡量其捕捉解剖模式规律性的能力;评估全生命周期3HG对应推断的有效性,衡量3HG嵌入向量在提供可靠的跨受试者和跨组解剖对应方面的能力;以及通过消融研究比较不同迁移策略和不同粒度脑图谱的影响。
Discussion讨论
04
本研究提出的CDC迁移框架旨在解决全生命周期脑解剖对应中的数据稀缺和异质性问题。它与现有医学图像分析中的其他迁移学习方法存在显著差异。当前医学图像分析的迁移学习主要集中在分类和分割任务,利用CNN等架构处理图像特征,并通常采用权重初始化和微调两种方式。这些方法依赖于源任务和目标任务之间特征的相似性,如共享的图像相关特性。然而,本研究采用3HG网络作为输入,处理的是全生命周期脑解剖对应问题,其数据格式和模型应用场景与现有主流迁移学习研究不同,难以直接进行公平比较。因此,本研究没有直接与其他文献中的迁移学习模型进行比较。CDC迁移框架具有良好的可扩展性和适用性。通过在一个大型独立数据集ADNI上验证,模型能够为新采集的个体生成3HG嵌入向量,并准确推断跨生命周期的3HG对应关系,即使在不同年龄组和病理状态下(例如ADNI数据集中未区分正常老年人与MCI/AD患者)。这表明该框架具有良好的泛化能力,能够应用于新的独立数据集。未来的工作可以进一步探索3HG在患者群体与正常个体之间分布模式的差异。
Conclusion结论
05
本研究引入了皮层发育连续性(CDC)迁移框架,旨在利用皮层折叠发育的连续性以优化模型性能。该方法通过遵循大脑皮层发育轨迹,将感兴趣区域(ROI)特征嵌入在不同年龄组之间进行迁移,从而实现共同模式的积累和传播,同时捕捉特定年龄组的模式。为有效指导迁移过程,本研究引入了两个新颖的正则化器,以促进共同模式和组特异性模式的捕获。通过在包含四个年龄组(从34孕周到青年成人)的1000多个大脑的多数据集上进行实验评估,结果表明,该CDC迁移框架显著改善了训练样本有限人群的模型性能。此外,CDC迁移学习方法能够稳健地推断相同或不同神经发育阶段不同大脑之间复杂的“多对多”解剖对应关系。CDC迁移框架为解决跨多个阶段的复杂问题提供了有价值的见解,这些问题特点是具有共享的内在关系和显著的跨阶段异质性。
Results结果
06
实验使用四个年龄组(成人、2岁、6个月、34孕周),共1000多个大脑,首先从成人组训练模型,然后依次迁移到2岁、6个月和34孕周组。在ROI嵌入评估中,结果显示3-hop连接由于覆盖范围广,可能导致连接强度表示不准确,因此1-hop和2-hop嵌入效果更佳。可视化结果表明,与从头训练相比,CDC迁移框架获得的嵌入相似性矩阵能更准确地捕捉ROI之间的连接差异,与真实连接强度矩阵高度相似,特别是在脑回-脑回连接区域。定量分析(余弦相似度、结构相似性指数和皮尔逊相关系数)也证实了CDC迁移策略在捕捉群组特异性模式方面的显著优势。在3HGs纵向对应推断方面,CDC迁移策略识别的对应3HG在不同年龄组个体间与共同解剖地标一致性高,错误率远低于从头训练的模型。例如,1-hop嵌入下CDC仅有2个不准确错误和2个缺失错误,而从头训练有14个不准确错误和27个缺失错误。CDC策略在不同年龄组间的对应质量没有显著差异,而从头训练的模型则差异显著,成人组结果尤其差。此外,CDC迁移框架学习的3HG嵌入能有效保留个体变异性,即在不同个体中能找到数量不等的对应3HG(从零到多个),准确反映个体大脑皮层的折叠模式。消融研究显示,CDC迁移框架中Lspe和Lcom两个正则化项对于准确捕捉群组特异性模式是必需的,且有益于加快收敛速度。在不同迁移策略比较中,遍历所有先前年龄组的迁移策略在捕捉群组特异性模式方面表现更优,并且训练过程中收敛速度更快。对于不同脑图谱的比较,随着粒度增加(ROI数量增多),每个ROI的平均3HG数量减少,平均余弦相似度降低,但3HG的识别精度提高。当粒度过高(如1000个ROI)时,可能会出现遗漏现有对应3HG的情况,表明需要平衡粒度以获得最佳效果。
Figure图
07
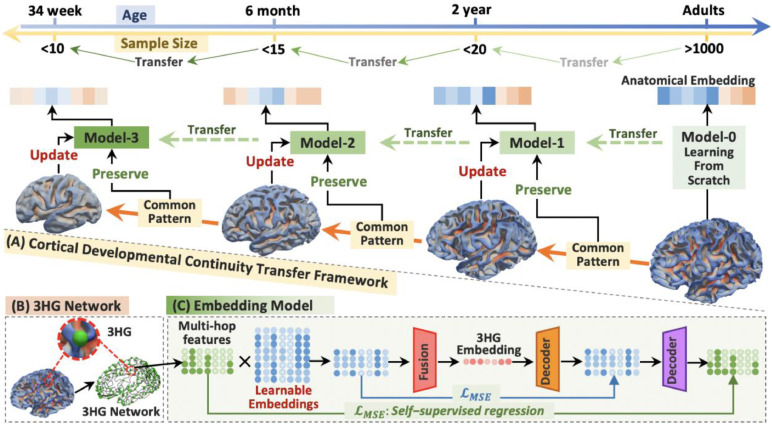
图1. (A) 所提出框架的总体方案。嵌入模型由具有最多数据样本的成人组从头开始训练,然后遵循大脑皮层发育轨迹(第3.2节)依次迁移并适应其他年龄组。嵌入模型的详细架构显示在(C)中。(B) 我们使用3HG网络来描述大脑解剖结构。每个3HG是三个方向的脑回交界处,同一半球上的3HG通过脑回铰链连接成网络(第3.1节)。对于每个3HG,我们使用其所在的解剖区域及其与其他3HG的多跳连接作为两个关键特征来生成多跳特征,该特征在学习过程中用作数据样本(第3.1节)。(C) 嵌入模型采用两级编码架构,将输入的每个3HG多跳特征分层映射到潜在表示(第3.1节)。
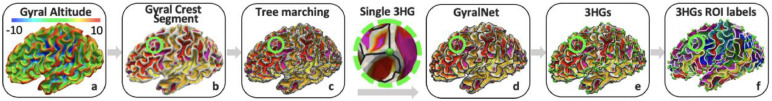
图2.3HGs识别流程。a:重建白质表面,并按脑回高度进行颜色编码。b:在脑回高度图上应用分水岭算法,将表面分为脑回顶部(白色区域)和脑沟底(彩色区域)。c:使用树形行进算法连接脑回顶部区域的顶点。d-e:修剪长度小于预定义阈值的冗余分支,保留的主干是3HG网络,在(Zhang et al. (2020c))中称为GyralNet。f:每个3HG根据其所属的感兴趣区域(ROI)进行标记。
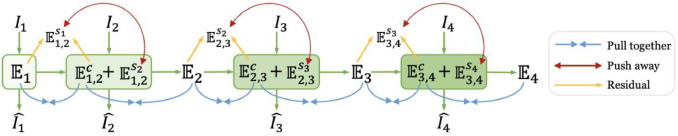
图3.所提出的皮层发育连续性(CDC)迁移框架中迁移过程的图示。该图以四个组为例展示了框架内的迁移过程。每个组(标记为组1、组2、组3和组4)都拥有自己的一组输入样本(Ii)并获得相应的输出(Ii^)。每个年龄组学习自己的融合层和两个解码层(WF、WD1和WD2),而嵌入Ei的学习过程由CDC迁移策略指导。具体而言,嵌入框架使用组1的输入样本从头开始训练,生成ROI特征嵌入(E1)。在训练过程中,从E1中提取一个共同部分,表示为E1,2c,代表组1和组2之间的共享特征。该共同部分与组2学习的组特异性嵌入(E1,2s2)相结合,生成组2的ROI特征嵌入。此过程由每对相邻组重复进行,包括组1和组2、组2和组3等,依次将学习到的ROI嵌入从一个组迁移到最近的相邻组。
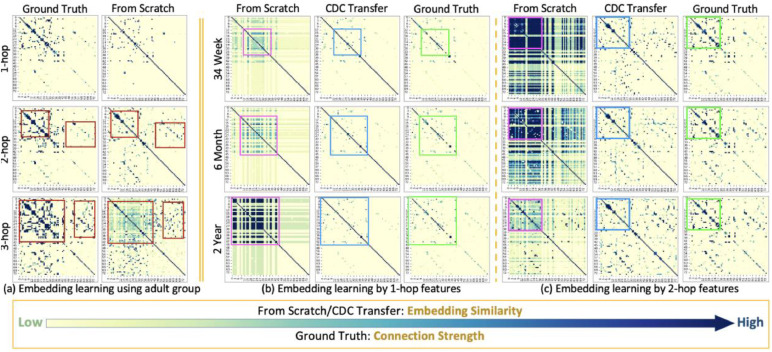
图4.使用3HG的l-hop连接强度(定义见3.1节)评估学习到的ROI嵌入。(a) 第一列显示了3HG的1-hop、2-hop和3-hop连接强度,这些强度是根据由1,064名受试者组成的成人组的全部人口计算得出的。三个不同hop的结果显示在三行中。第二列显示了嵌入相似性矩阵,这些矩阵是通过计算成对ROI嵌入之间的余弦相似度获得的。这些ROI嵌入是使用成人组的训练数据集从头开始学习的。(b) (a)中获得的1-hop嵌入的训练模型通过CDC迁移策略依次迁移到2岁组,然后是6个月组,最后是34孕周组。学习到的ROI嵌入的嵌入相似性矩阵显示在第二列。此外,对于每个组,使用相同的训练数据集从头训练了一个模型,学习到的ROI嵌入的嵌入相似性矩阵显示在第一列。第三列显示了连接强度矩阵(真实值),这些矩阵是根据每个年龄组的全部人口计算得出的。(c) 类似于(b),该子图显示了2-hop嵌入的结果。(a)、(b)和(c)中所有矩阵的脑区顺序对应于Destrieux图谱(Destrieux et al. (2010))中定义的顺序,其中前44个区域主要代表脑回,其余31个区域代表脑沟。
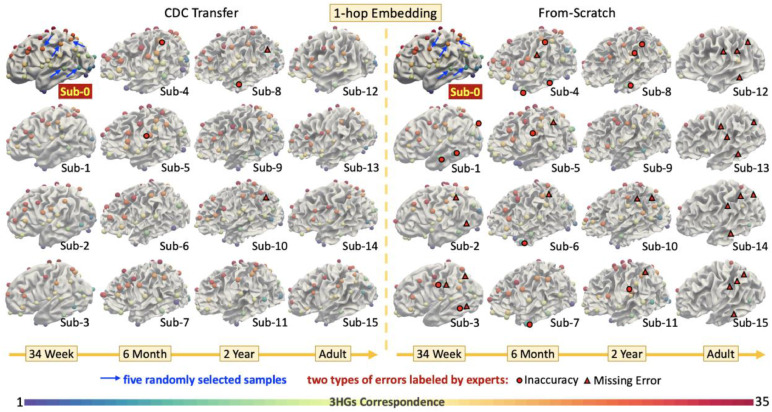
图5.通过1-hop嵌入的3HGs生命周期对应关系。选择34W组中随机选取的受试者(sub-0)的3HG作为示例3HG。为了在其他受试者身上找到对应的3HG,采用了以下流程:1)对于不同年龄组不同受试者中的每个3HG,计算其嵌入向量与示例3HG嵌入向量之间的余弦相似度;2)对于每个受试者,通过选择与示例3HG余弦相似度为1.0的3HG来识别对应的3HG;3)如果没有3HG的余弦相似度为1.0,则选择余弦相似度最高(高于预定义阈值)的3HG作为对应的3HG。遵循这些步骤,获得了不同受试者中每个示例3HG的对应3HG。为了更好地可视化,我们从sub-0中选择了35个跨越整个大脑皮层的示例3HG,并显示了使用不同方法获得的来自4个年龄组的15个随机选择的受试者(sub-1到sub-15)中的对应3HG。不同受试者中的对应3HG用相同的颜色表示。两名专家参与评估对应结果,两种类型的错误:不准确/缺失错误,分别用红色圆圈/黑色边框的红色三角形标记。
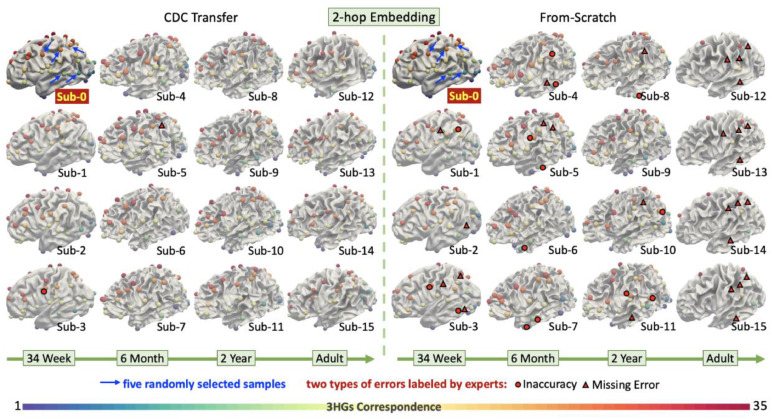
图6通过2-hop嵌入的3HGs生命周期对应关系。详细描述请参考图5。
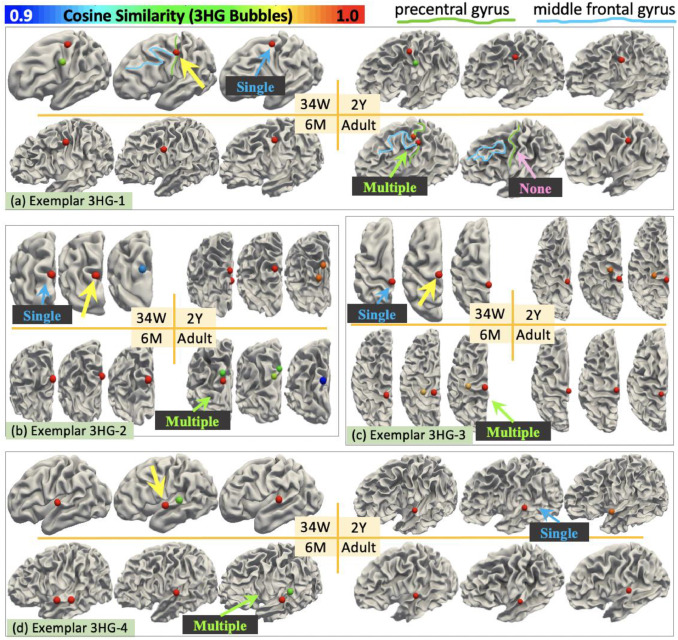
图7跨受试者的个体差异和变异性。我们选择了四个示例3HG(由黄色箭头指示)来检查它们在来自四个不同年龄组(34W、6M、2Y和成人)的受试者中的对应关系。对于每个年龄组,我们随机选择了三名受试者,并基于0.9的余弦相似度阈值建立了对应关系。对应的3HG以气泡形式可视化,气泡的颜色表示余弦相似度。鉴于大脑皮层的强烈变异性,在不同受试者中可能会找到不同数量的对应关系,从零到多个。
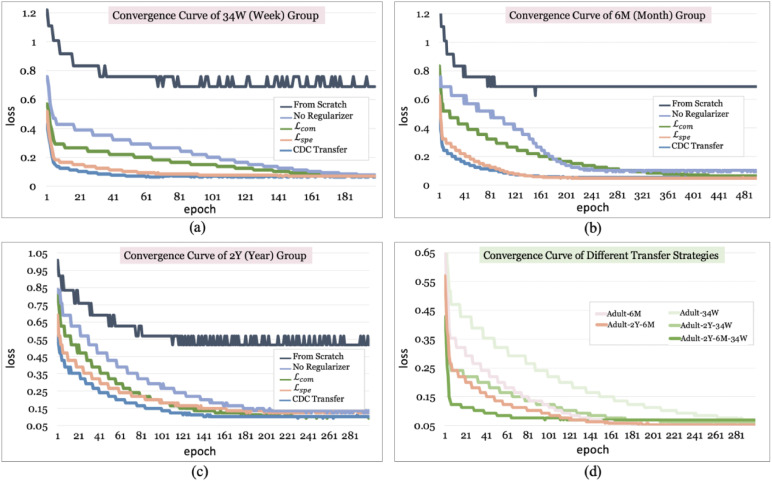
图8不同正则化器设置(a-c)和不同迁移策略(d)的收敛曲线。
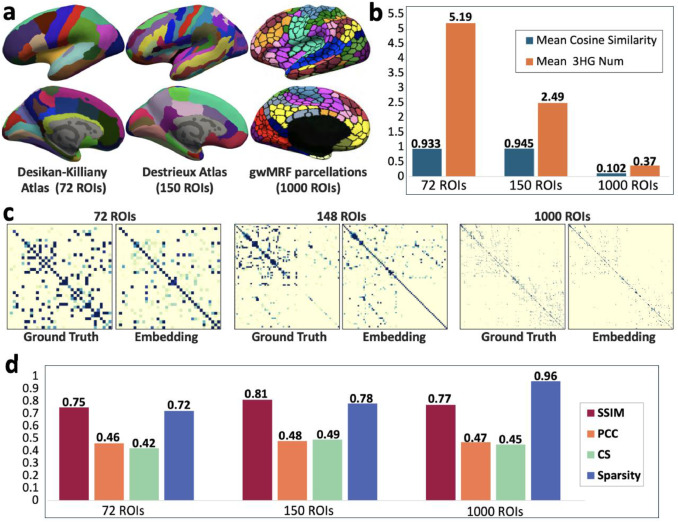
图9不同图谱的比较。a. 三种不同粒度级别的图谱。b. 三种图谱的top-1对应3HG的平均余弦相似度和每个ROI的平均3HG数量。c. 三种不同图谱的学习到的嵌入相似性矩阵和相应的真实值矩阵。d. 四种度量用于测试真实值和学习到的嵌入矩阵之间的相似性以及这些矩阵的稀疏性。
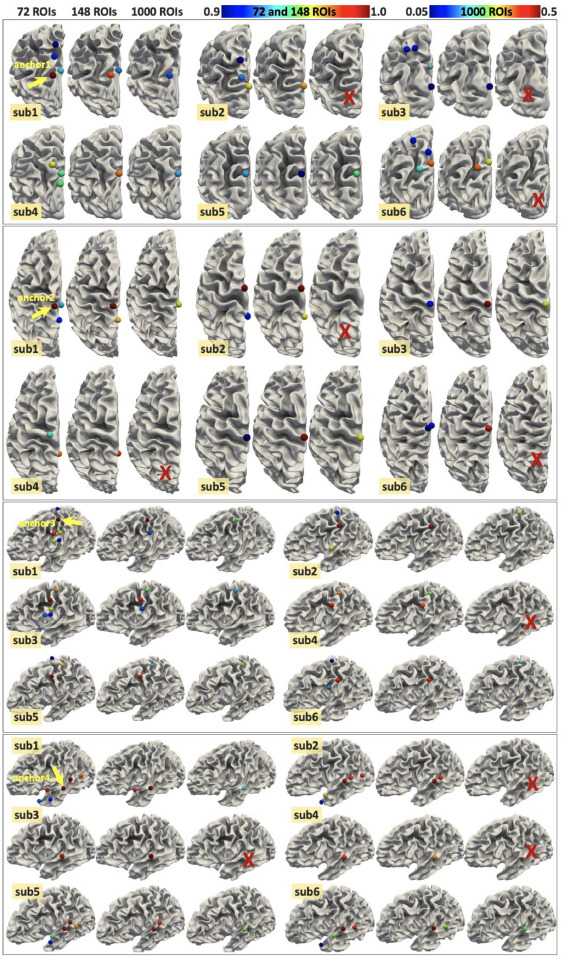
图10使用不同脑图谱在6名随机选择的受试者中识别的4个锚定3HG的对应3HG结果。
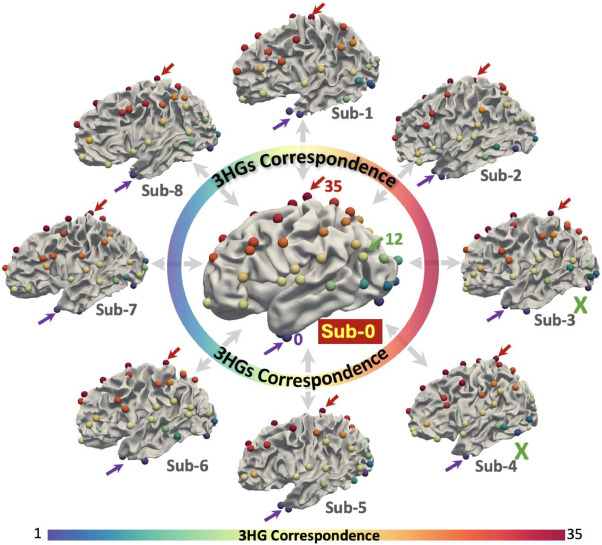
图11新的独立ADNI数据集的3HG生命周期对应结果。
更多推荐
 已为社区贡献20条内容
已为社区贡献20条内容








所有评论(0)