MONAI Label: A framework for AI-assisted Interactive Labeling of 3D Medical Images论文精读
MONAI Label 是一个端到端的标注与学习平台交互式与非交互式标注主动学习策略多前端支持可扩展的AI模型集成DICOM兼容与PACS集成MONAI Label 是一个功能全面、开源灵活、临床可用的AI辅助标注平台,显著降低了3D医学图像标注的门槛与时间成本,推动了医学影像AI模型的快速迭代与临床应用。
·

这篇题为《MONAI Label:一个用于3D医学图像AI辅助交互式标注的框架》的论文,系统性地介绍了一个开源医学图像标注平台。以下是对该论文的详细解析:
一、研究背景与问题
- 核心问题:医学图像标注(尤其是3D图像)耗时耗力,专业标注资源稀缺,成为训练监督式深度学习模型的主要瓶颈。
- 现有方案:传统的全自动分割方法在临床中尚未达到理想鲁棒性;交互式分割方法虽更可靠,但缺乏统一、易用的开源平台。
- 解决方案:提出MONAI Label,一个免费、开源、支持AI辅助交互式标注的框架,旨在降低标注成本、提升标注效率。
二、MONAI Label 框架概述
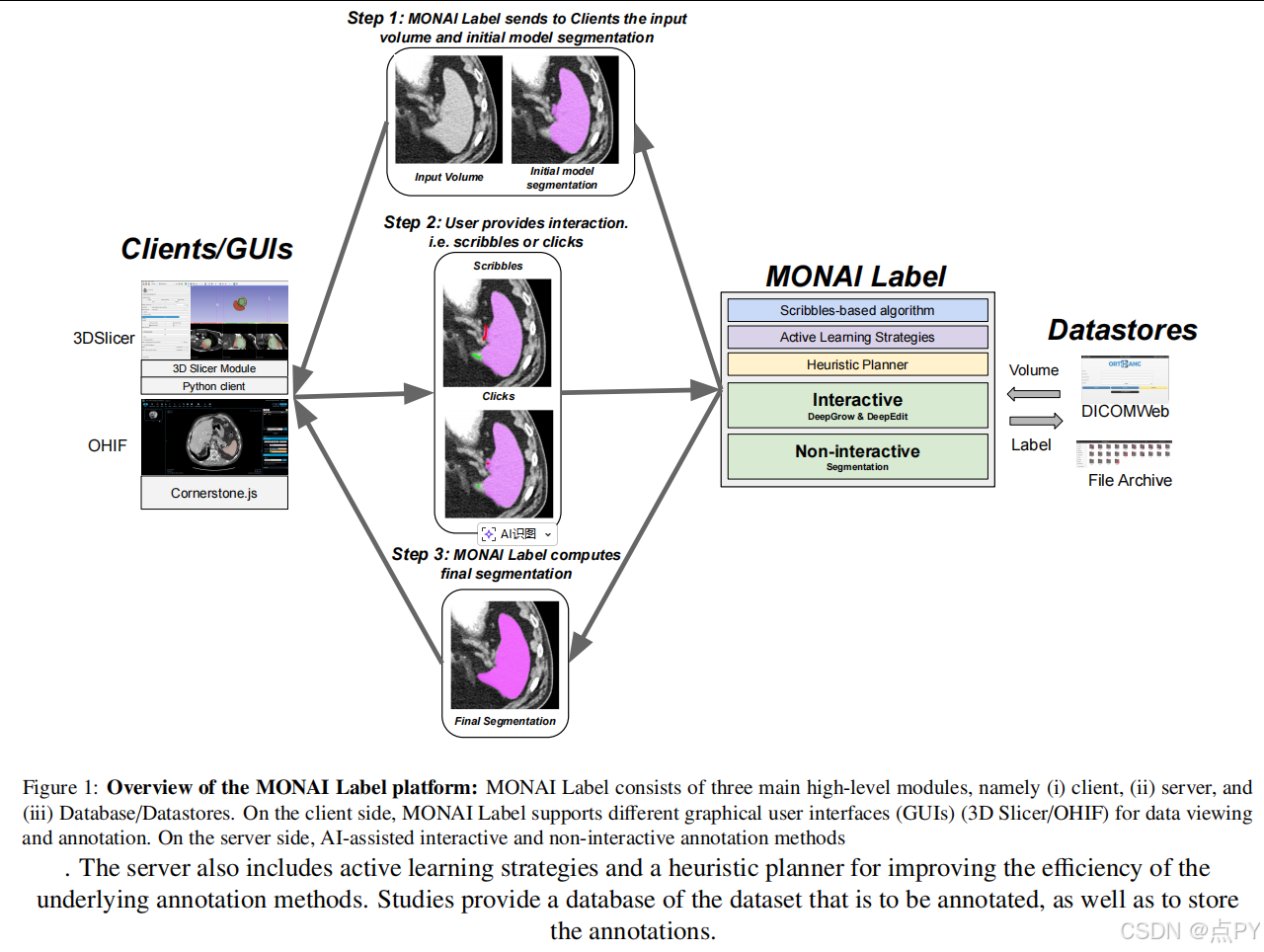
MONAI Label 是一个端到端的标注与学习平台,支持:
- 交互式与非交互式标注
- 主动学习策略
- 多前端支持(3D Slicer、OHIF)
- 可扩展的AI模型集成
- DICOM兼容与PACS集成
主要架构模块:
- 客户端:支持3D Slicer(本地)和OHIF(Web)两种GUI。
- 服务器:提供AI模型推理、训练、主动学习策略等服务。
- 数据库/数据存储:支持本地存储与DICOM服务器。
三、核心标注方法
1. 交互式标注
- DeepGrow:基于用户正负点击的交互式分割模型,支持2D/3D标注。
- DeepEdit:扩展DeepGrow,支持“无点击”自动推理 + “点击”交互编辑,便于主动学习集成。
- Scribbles(涂鸦):基于能量优化的交互分割,支持在线似然分割与CNN分割优化。
2. 非交互式标注
- 自动分割模型:基于标准CNN(如UNet)的全自动分割,可作为基准或预处理步骤。
四、主动学习策略
MONAI Label 集成了主动学习机制,用于智能选择待标注样本,提升标注效率:
- 不确定性估计:支持基于Dropout的认知不确定性估计。
- 采集函数:支持选择最不确定样本,用户也可自定义策略。
- 适用于DeepEdit:其自动推理部分可直接用于未标注图像的排序。
五、实验结果
论文在两个公开数据集上验证了标注效率:
1. DeepGrow 在脾脏分割数据集(MSD)
- 标注时间随训练阶段增加而显著减少。
- 第4阶段使用DeepGrow 2D/3D组合,标注一个3D体积仅需1–2.5分钟,比传统方法快10倍。
2. DeepEdit 在心脏MRI左心房分割
- 医生仅需标注1–2张图像即可启动训练,后续标注可通过模型辅助交互完成。
- 显著降低总标注时间,实现“边标注边训练”的持续学习流程。
六、实际应用案例
-
Telestroke(远程卒中服务):
- 用于脑出血CT图像的自动分割与量化。
- 模型在标注过程中持续优化,支持临床决策。
-
Neurosurgical Atlas(神经外科图谱):
- 用于脑肿瘤及周围结构的多模态MR分割。
- 标注时间减少50%–80%,支持手术规划。
七、系统特点与创新
- 开源与可扩展:基于Python/PyTorch,支持自定义模型与交互方式。
- 多模态支持:支持3D医学图像、病理图像(WSI)等。
- 主动学习集成:内置不确定性估计与样本选择策略。
- 临床友好:提供3D Slicer与OHIF插件,便于集成到临床工作流。
- 启发式规划器:自动优化GPU使用与训练配置。
八、未来工作方向
- 支持更多交互方式(如ROI、闭合曲线)。
- 扩展病理图像分析功能。
- 社区驱动开发,欢迎反馈与贡献。
九、总结
MONAI Label 是一个功能全面、开源灵活、临床可用的AI辅助标注平台,显著降低了3D医学图像标注的门槛与时间成本,推动了医学影像AI模型的快速迭代与临床应用。
十、开源代码
更多推荐
 已为社区贡献50条内容
已为社区贡献50条内容







所有评论(0)