大模型工具的引入
将疾病非结构化文本转化为统一格式的结构化数据,以支持后续疾病知识图谱的构建与图数据库存储。结合前一阶段爬取的疾病文本内容特点,并遵循“不过度推断、尽量贴近原文”的原则,实验最终定义了以下 10 个结构化抽取字段:disease_name(疾病名称)affected_part(累及部位)department(就诊科室)symptoms(症状表现)risk_groups(高风险人群)infectiou
本实验围绕“文本型疾病描述数据的结构化建模与图数据库智能查询”这一核心目标,结合大模型技术与图数据库技术,构建从非结构化医学文本 → 结构化知识 → 图数据库 → 自然语言问答的完整实验流程。实验整体设计分为数据获取、信息抽取、图数据库构建以及大模型图查询四个阶段,逐步验证大模型在医学知识建模与智能检索场景下的应用效果。
一、实验数据与预处理
本实验所使用的数据来源于公开医学健康网站——39 健康网,数据内容主要为各类疾病的文字介绍信息。该类数据以自然语言文本形式呈现,具有信息密集、语义丰富但结构松散的特点,适合作为大模型进行医学文本结构化抽取与知识建模的实验数据来源。
在数据采集阶段,实验采用后羿采集器作为爬虫工具,对 39 健康网中疾病相关页面进行定向爬取。爬取内容主要包括疾病名称及其对应的文本描述信息。
输入对应网址,爬取数据:
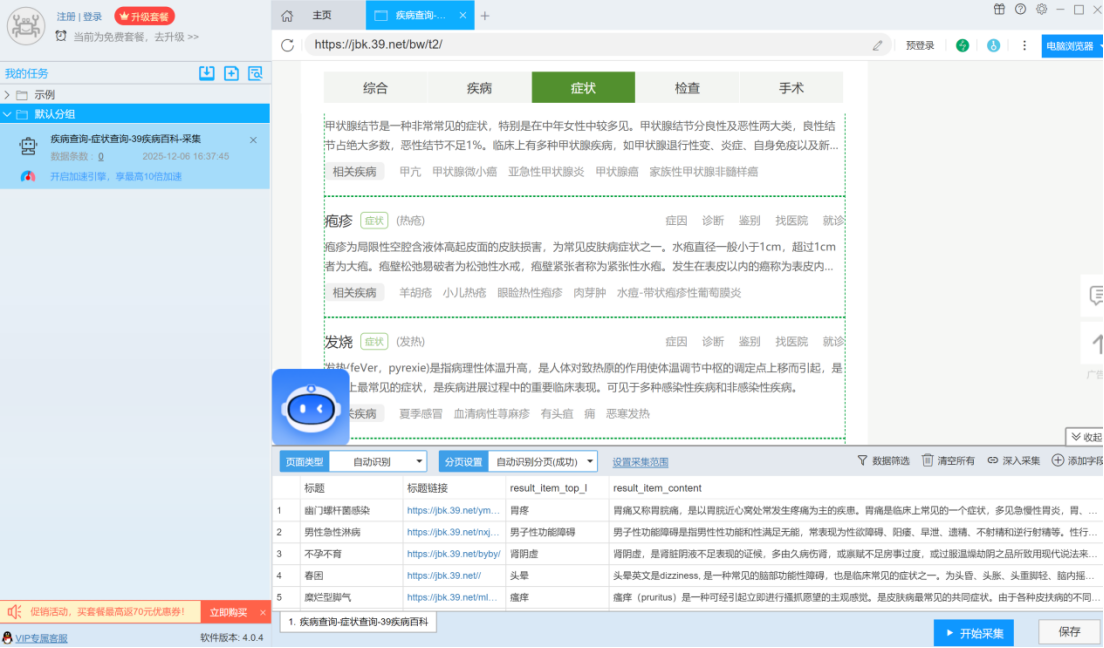
由于原始采集数据属于典型的非结构化文本数据,在正式进入大模型处理之前,需要进行必要的数据预处理操作。首先,对爬取结果进行格式整理,统一存储为 CSV / Excel 等结构化文件格式,确保每条记录对应一类疾病,并保留完整的原始文本描述。

其次,对文本内容进行基础清洗,包括去除网页冗余信息、无关标签、特殊符号及重复段落,减少噪声对模型抽取结果的干扰。此外,在预处理过程中对数据进行了初步规范化处理,如统一疾病名称表达方式,删除明显缺失或内容异常的样本,确保数据语义清晰、文本完整。经过上述步骤处理后,随机抽取其中50种疾病,最终形成了一份质量较高的疾病非结构化文本数据集,为后续基于大模型的结构化信息抽取实验提供了可靠的数据输入。清洗数据的具体代码prepare_disease_texts.py 如下:
import pandas as pd
import re
import random
# 1. 读取疾病文件
df = pd.read_csv("disease1.csv", encoding='utf-8')
# 2. 对 description 长度进行初筛(过滤掉无意义/太短内容)
df["desc_length"] = df["description"].astype(str).apply(len)
candidate = df[df["desc_length"] > 50].copy() # 可按需调整阈值
# 3. 文本清洗函数:去噪、去前缀、规范空格
def clean_text(text):
text = str(text)
# 去掉可能存在的常见前缀
text = re.sub(r"^(疾病:|病因:|描述:|答:|医生:)", "", text)
# 去掉 HTML 标签
text = re.sub(r"<.*?>", "", text)
# 去掉多余空格/换行
text = re.sub(r"\s+", " ", text).strip()
# 去掉常见噪声符号
noise_chars = r"[★☆◆■●▲▼□◇…→←↑↓]"
text = re.sub(noise_chars, "", text)
return text
# 4. 应用清洗
candidate["clean_description"] = candidate["description"].apply(clean_text)
# 5. 再次过滤:清洗后长度不足的去除
candidate = candidate[candidate["clean_description"].apply(len) > 60]
# 6. 去重
candidate = candidate.drop_duplicates(subset=["clean_description"])
# 7. 随机抽取50条
sample_n = min(50, len(candidate))
final_samples = candidate.sample(n=sample_n, random_state=42)[["name", "clean_description"]]
final_samples = final_samples.reset_index(drop=True)
final_samples.columns = ["disease_name", "disease_text"]
# 8. 保存输出
final_samples.to_csv("disease_50_samples.csv", index=False, encoding="utf-8-sig")
print("完成!已生成 disease_50_samples.csv,共 {} 条疾病描述文本".format(sample_n))
print(final_samples.head())
通过对实验数据的合理采集与预处理,有效保证了疾病文本数据的可用性与一致性,为后续大模型抽取、知识图谱构建以及图数据库查询实验奠定了数据基础。
二、大模型结构化抽取实验设计与对比分析
1. 大模型平台选择与服务配置
本实验采用阿里云“百炼”大模型服务平台作为大模型调用环境,使用其中的通义千问(Qwen-Plus)模型完成医学文本的结构化信息抽取任务。百炼平台为用户提供标准化的大模型 API 接口,并支持通过兼容 OpenAI 协议的方式进行调用,便于与现有 Python 生态及 LangChain 框架集成。
实验前,首先在阿里云官网完成账号注册与实名认证,并在控制台中开通“百炼”大模型服务。平台为新用户提供一定的免费调用额度,使得实验能够在不额外增加成本的情况下完成多轮模型调用与参数对比测试。随后,通过配置环境变量的方式,将 DashScope API Key 安全地加载至实验环境中,确保模型调用过程的稳定性与安全性。
2. 实验任务定义与字段设计
本部分的核心任务是:将疾病非结构化文本转化为统一格式的结构化数据,以支持后续疾病知识图谱的构建与图数据库存储。结合前一阶段爬取的疾病文本内容特点,并遵循“不过度推断、尽量贴近原文”的原则,实验最终定义了以下 10 个结构化抽取字段:
-
disease_name(疾病名称)
-
affected_part(累及部位)
-
department(就诊科室)
-
symptoms(症状表现)
-
risk_groups(高风险人群)
-
infectious(是否具有传染性)
-
check_method(检查方式)
-
drug(常用药物)
-
rate(治愈率或控制率)
-
insurance(医保情况)
上述字段既能够覆盖疾病文本中的核心医学信息,又保持了较好的通用性,避免因字段过于细碎而导致模型抽取失败率升高。
3. Prompt 设计与对比方案
为系统评估不同提示策略与模型随机性参数对抽取效果的影响,本实验设计了3 种 Prompt 形式 × 3 种 Temperature 参数的对比实验,共形成 9 种实验组合。
实验中采用了以下三类Prompt:
-
简洁指令型 Prompt
仅给出任务目标、字段定义和输出格式,强调直接从文本中抽取信息,适合测试模型在最少约束条件下的抽取能力。 -
详细指令型 Prompt
在简洁指令基础上进一步强调“不得推断、不补充、不解释”,引导模型严格对齐原文内容进行信息抽取,以降低幻觉风险。 -
示例引导型 Prompt
在正式抽取前提供完整示例,通过“示例—文本—JSON 输出”的方式,帮助模型更好地理解字段含义和输出格式,增强对复杂文本的适应能力。
Temperature 用于控制模型生成结果的随机性,本实验选取以下三种典型取值:
-
0.0:生成结果最稳定、确定性最强
-
0.5:在稳定性与多样性之间折中
-
1.0:生成随机性较高,语言灵活度增强
通过对不同 Temperature 下抽取结果的对比,分析模型随机性对结构化抽取质量的影响。
4. 实验流程与结果分析
实验采用 Python 编程实现,基于 LangChain 框架对通义千问模型进行调用。
文件名:extraction_experiment.py
import pandas as pd
import os
import json
import time
from tqdm import tqdm
from dotenv import load_dotenv
from langchain_openai import ChatOpenAI
# 1. 环境 & API Key
load_dotenv()
if not os.environ.get("DASHSCOPE_API_KEY"):
api_key = input("请输入你的 DashScope API-Key: ").strip()
os.environ["DASHSCOPE_API_KEY"] = api_key
print("API Key 状态:", "已就位" if os.environ.get("DASHSCOPE_API_KEY") else "缺失")
# 2. 加载数据
try:
df = pd.read_csv("disease_50_samples(1).csv")
assert "disease_text" in df.columns
print(f"成功加载 {len(df)} 条文本")
except Exception as e:
print("❌ 数据加载失败:", e)
exit(1)
# 3. 字段定义(与文本严格对齐)
FIELDS = [
"disease_name",
"affected_part",
"department",
"symptoms",
"risk_groups",
"infectious",
"check_method",
"drug",
"rate",
"insurance"
]
# 4. Prompt 构造函数
def build_prompt(prompt_type, text):
json_template = """{
"disease_name": "",
"affected_part": "",
"department": "",
"symptoms": "",
"risk_groups": "",
"infectious": "",
"check_method": "",
"drug": "",
"rate": "",
"insurance": ""
}"""
if prompt_type == "简洁指令":
return f"""
请从以下医学文本中抽取结构化信息,仅返回 JSON。
字段未出现请返回空字符串 ""。
JSON格式:
{json_template}
文本:
{text}
"""
if prompt_type == "详细指令":
return f"""
你是医学文本结构化抽取专家。
任务:从文本中“照原文抽取”信息,不允许推断或补充。
不要输出解释,只返回 JSON。
JSON格式:
{json_template}
文本:
{text}
"""
if prompt_type == "示例引导":
return f"""
请参考示例,从文本中抽取疾病结构化信息,仅返回 JSON。
示例:
文本:
水痘是一种常见疾病,主要累及皮肤。就诊于传染科。
症状包括发烧、水泡。传染性:有。治愈率95%。医保:是。
JSON:
{{
"disease_name": "水痘",
"affected_part": "皮肤",
"department": "传染科",
"symptoms": "发烧,水泡",
"risk_groups": "",
"infectious": "有传染性",
"check_method": "",
"drug": "",
"rate": "95%",
"insurance": "是"
}}
现在处理以下文本:
{text}
"""
raise ValueError("未知 Prompt 类型")
# 5. 后处理 & 评分函数
def normalize_fields(data):
return {k: data.get(k, "") or "" for k in FIELDS}
def score_extraction(data):
return sum(1 for k in FIELDS if data[k].strip())
# 6. 主实验循环(3×3)
PROMPTS = ["简洁指令", "详细指令", "示例引导"]
TEMPERATURES = [0.0, 0.5, 1.0]
summary_results = []
all_structured_records = []
for prompt_name in PROMPTS:
for temp in TEMPERATURES:
print(f"\n===== Prompt={prompt_name} | Temp={temp} =====")
llm = ChatOpenAI(
model="qwen-plus",
temperature=temp,
base_url="https://dashscope.aliyuncs.com/compatible-mode/v1",
api_key=os.environ["DASHSCOPE_API_KEY"]
)
total_score = 0
success = 0
for _, row in tqdm(df.iterrows(), total=len(df)):
text = row["disease_text"]
prompt = build_prompt(prompt_name, text)
try:
response = llm.invoke(prompt).content.strip()
data = json.loads(response)
data = normalize_fields(data)
score = score_extraction(data)
total_score += score
success += 1
data["Prompt"] = prompt_name
data["Temperature"] = temp
data["Score"] = score
all_structured_records.append(data)
time.sleep(0.1)
except Exception:
continue
avg_score = total_score / success if success > 0 else 0
summary_results.append({
"Prompt": prompt_name,
"Temperature": temp,
"成功抽取条数": success,
"平均得分(满分10)": round(avg_score, 2)
})
# 7. 输出结果
summary_df = pd.DataFrame(summary_results)
detail_df = pd.DataFrame(all_structured_records)
summary_df.to_excel("3x3_抽取效果对比(1).xlsx", index=False)
detail_df.to_excel("疾病结构化结果_全部组合(1).xlsx", index=False)
print("\n✅ 实验完成")
print("已生成:")
print(" - 3x3_抽取效果对比.xlsx")
print(" - 疾病结构化结果_全部组合.xlsx")导入大模型:

运行结果:
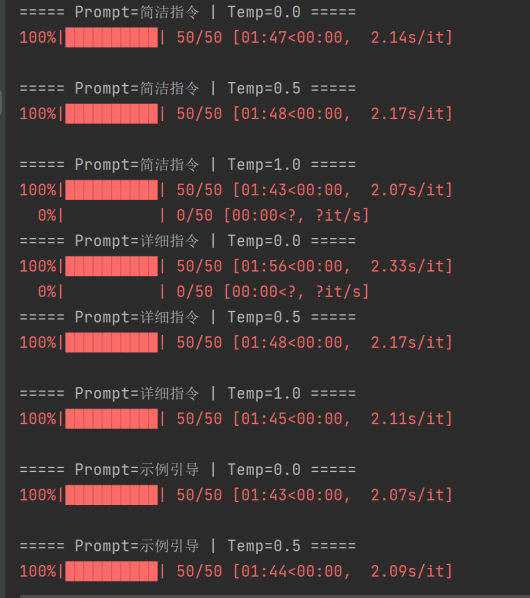
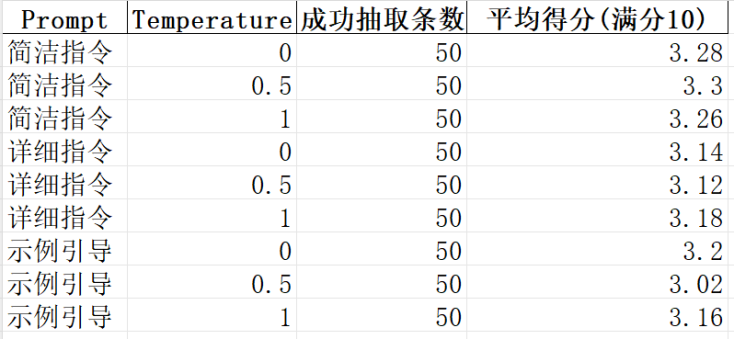
从实验结果可以看出,在 3 种 Prompt 形式与 3 种 Temperature 组合下,模型均能够对全部 50 条疾病文本完成结构化抽取,说明通义千问模型在该任务上的整体稳定性较好,不同参数设置不会导致抽取失败。从平均得分来看,各组合的得分集中在 3.0~3.3 分之间,整体差异不大,表明在当前字段设计与文本复杂度条件下,模型对显性疾病信息(如疾病名称、部分症状等)的抽取能力较强,但对隐含或文本中出现频率较低的字段(如治愈率、医保情况、检查方式等)覆盖有限。对比不同 Prompt 形式可以发现,简洁指令型 Prompt 的平均得分略高,说明在字段较多但文本信息有限的情况下,过于复杂的指令或示例并未显著提升抽取效果,反而可能引入一定约束。Temperature 的变化对结果影响较小,低温度下结果略为稳定,高温度下存在轻微波动,但整体未出现明显性能退化,表明该任务对模型随机性的敏感度较低。
疾病结构化结果:
三、Neo4j / TuGraph 系统架构与实现
1. Neo4j 系统实现流程
启动docker,用powershell指令进入windows命令行,输入docker images,查看是否有名字叫 docker.lms.run/library/neo4j 的镜像。命令行输入:
docker run --publish=7475:7474 --publish=7688:7687 `--volume="//c/Users/zhang/Desktop/专业综合实践/data:/data" `--volume="//c/Users/zhang/Desktop/专业综合实践/import:/import" `docker.1ms.run/library/neo4j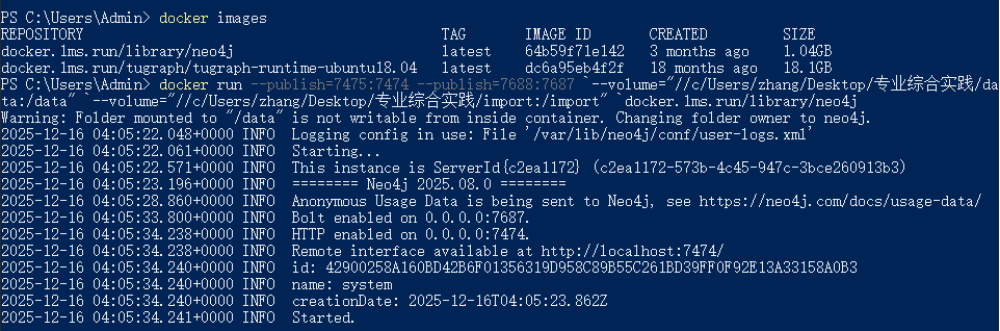
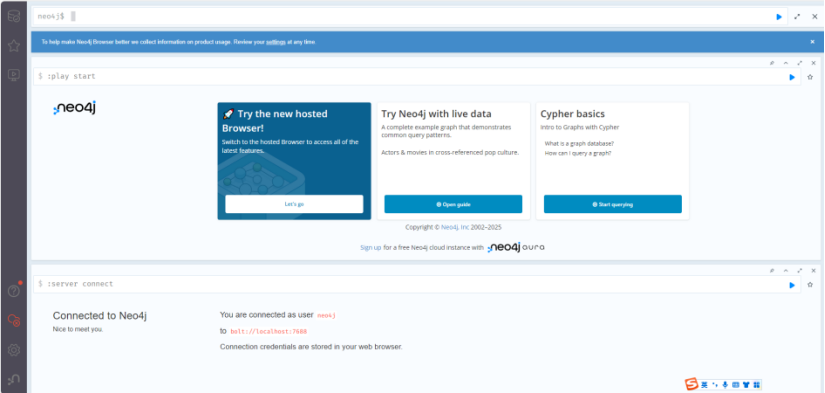
打开浏览器,在浏览器地址栏输入:localhost:7475
输入用户名和密码,进入图形化操作界面
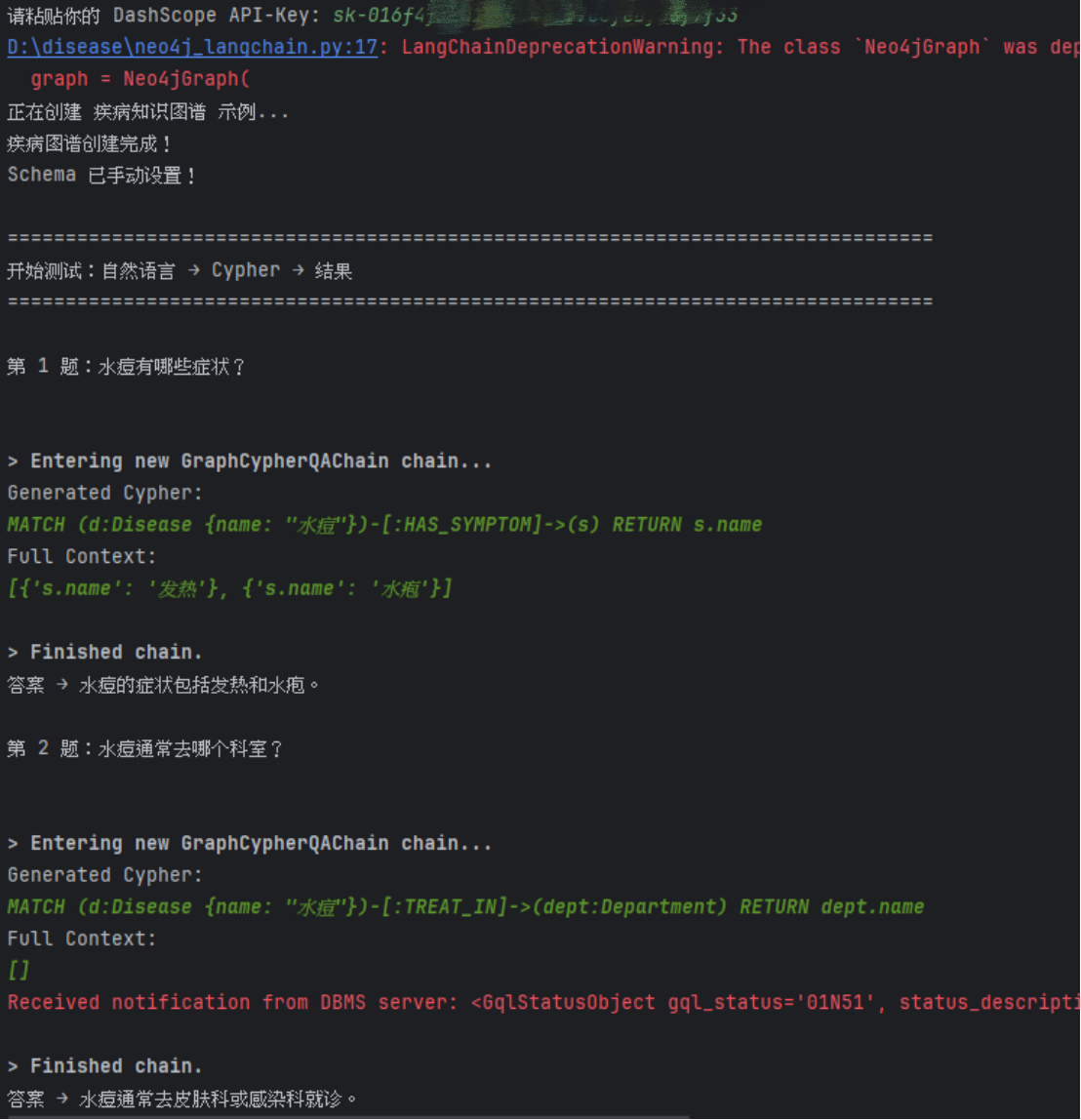
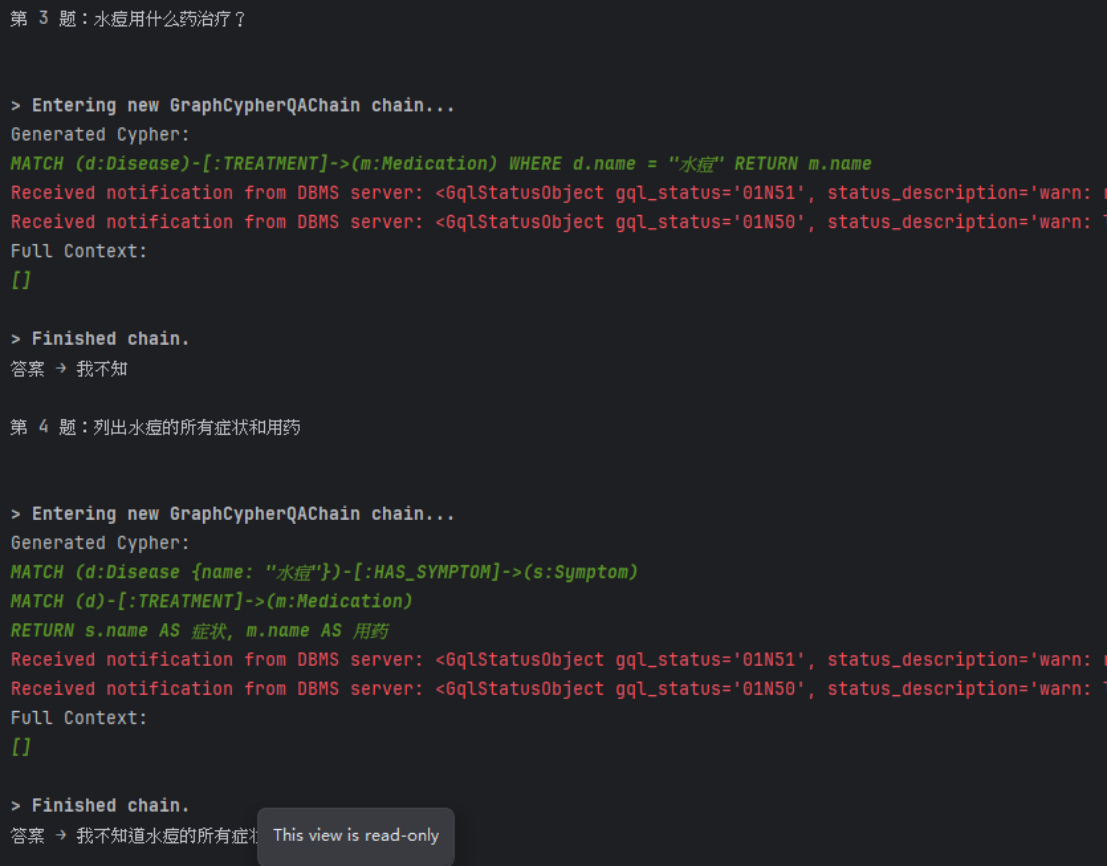
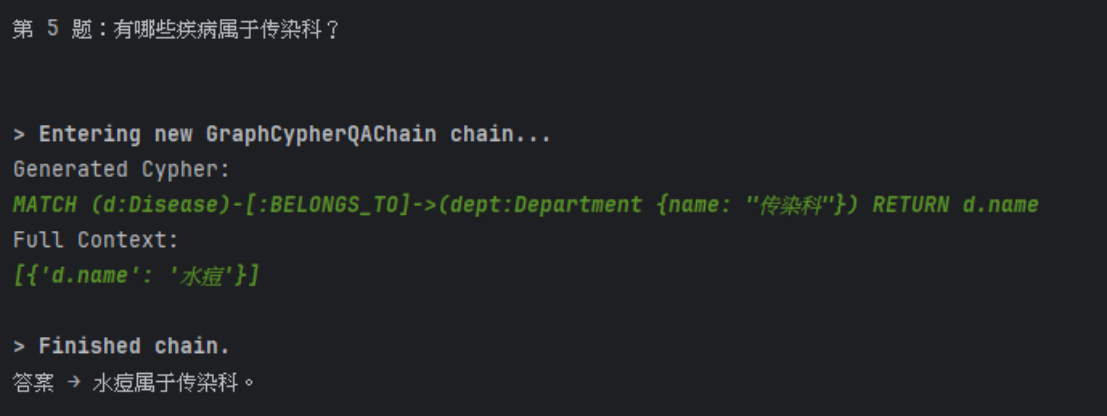
在 Neo4j 实验环境中,首先通过 Python 驱动与数据库建立连接,并构建包含疾病、症状、药物等节点及其关系的示例图谱。随后引入通义千问大模型,结合 LangChain 提供的图问答链,对用户输入的自然语言问题进行处理。
文件名:neo4j_langchain.py
from langchain_openai import ChatOpenAI
from langchain_community.graphs import Neo4jGraph
from langchain.chains import GraphCypherQAChain
import os
# 密钥处理
from dotenv import load_dotenv
load_dotenv()
if not os.environ.get('DASHSCOPE_API_KEY'):
os.environ['DASHSCOPE_API_KEY'] = input("请粘贴你的 DashScope API-Key: ").strip()
os.environ["OPENAI_API_KEY"] = os.environ["DASHSCOPE_API_KEY"]
# 连接 Neo4j
graph = Neo4jGraph(
url="bolt://localhost:7687",
username="neo4j",
password="rxt0413zC",
refresh_schema=False # 关键:关闭自动 schema
)
# 建立疾病知识图谱
print("正在创建 疾病知识图谱 示例...")
graph.query("MATCH (n) DETACH DELETE n")
graph.query("""
MERGE (d:Disease {name: "水痘"})
MERGE (s1:Symptom {name: "发热"})
MERGE (s2:Symptom {name: "水疱"})
MERGE (dpt:Department {name: "传染科"})
MERGE (drug:Drug {name: "阿昔洛韦"})
MERGE (d)-[:HAS_SYMPTOM]->(s1)
MERGE (d)-[:HAS_SYMPTOM]->(s2)
MERGE (d)-[:BELONGS_TO]->(dpt)
MERGE (d)-[:TREATED_BY]->(drug)
""")
print("疾病图谱创建完成!")
# 手动 Schema
graph.schema = """
Node properties are the following:
Disease {name: STRING}
Symptom {name: STRING}
Drug {name: STRING}
Department {name: STRING}
Relationship properties are the following:
(No relationship properties)
The relationships are the following:
(:Disease)-[:HAS_SYMPTOM]->(:Symptom)
(:Disease)-[:TREATED_BY]->(:Drug)
(:Disease)-[:BELONGS_TO]->(:Department)
"""
print("Schema 已手动设置!")
# 创建 LLM + Cypher QA Chain
llm = ChatOpenAI(
model="qwen-plus",
temperature=0,
base_url="https://dashscope.aliyuncs.com/compatible-mode/v1"
)
chain = GraphCypherQAChain.from_llm(
llm=llm,
graph=graph,
verbose=True, # 调试 Cypher
allow_dangerous_requests=True
)
# 测试问题
questions = [
"水痘有哪些症状?",
"水痘通常去哪个科室?",
"水痘用什么药治疗?",
"列出水痘的所有症状和用药",
"有哪些疾病属于传染科?"
]
print("\n" + "="*80)
print("开始测试:自然语言 → Cypher → 结果")
print("="*80)
for i, q in enumerate(questions, 1):
print(f"\n第 {i} 题:{q}")
result = chain.invoke({"query": q})
print("答案 →", result["result"])系统在运行时先由大模型根据预定义的 Schema 自动生成 Cypher 查询语句,再由 Neo4j 执行该语句并返回查询结果。通过开启 verbose 模式,可以清晰地观察到模型生成的 Cypher 语句,从而验证其与图数据库结构的一致性。实验结果表明,在 Schema 定义清晰、约束合理的前提下,大模型能够正确生成可执行的 Cypher 查询语句。
2. TuGraph 系统实现流程
在 TuGraph 平台中,由于其不完全支持 LangChain 默认的自动事务与 Schema 刷新机制,实验对系统架构进行了针对性调整。首先,通过 TuGraph 控制台或导入工具提前完成疾病知识图谱的数据导入与 Schema 创建;随后在程序中显式指定数据库名称,并采用自动提交(auto-commit)方式执行 Cypher 查询,避免显式事务引发的兼容性问题。
首先启动 TuGraph,在命令行中执行:
docker run -d -v D:\disease:/mnt -p 7070:7070 -p 7687:7687 docker.1ms.run/tugraph/tugraph-runtime-ubuntu18.04 lgraph_server
在浏览器地址栏输入 http://localhost:7070,即可进入 TuGraph 图形化操作界面。
接着将第二部分处理好的结构化文本数据导入TuGraph,并运行脚本TuGraph_langchain.py:
from langchain_openai import ChatOpenAI
from langchain_core.prompts import ChatPromptTemplate
from neo4j import GraphDatabase
import os
from dotenv import load_dotenv
# 1. 密钥
load_dotenv()
if not os.environ.get("DASHSCOPE_API_KEY"):
os.environ["DASHSCOPE_API_KEY"] = input("请输入 DashScope API Key: ").strip()
os.environ["OPENAI_API_KEY"] = os.environ["DASHSCOPE_API_KEY"]
# 2. 连接 TuGraph
driver = GraphDatabase.driver(
"bolt://localhost:7687",
auth=("admin", "73@TuGraph")
)
def run_cypher(cypher: str):
with driver.session(database="default") as session:
return session.run(cypher).data()
# 3. LLM
llm = ChatOpenAI(
model="qwen-plus",
temperature=0,
base_url="https://dashscope.aliyuncs.com/compatible-mode/v1"
)
# ==================== 4. Prompt ====================
prompt = ChatPromptTemplate.from_messages([
(
"system",
"""
你是 TuGraph 图数据库专家,需要将中文医疗问题转换为 Cypher 查询。
图数据库 Schema:
节点:
- Disease(name)
- Drug(name)
- Symptom(name)
- Department(name)
- Age(name)
关系:
- (Disease)-[:HAS_Drug]->(Drug)
- (Disease)-[:HAS_SYMPTOM]->(Symptom)
- (Disease)-[:IS_OF_Department]->(Department)
- (Disease)-[:IS_OF_AGE]->(Age)
【生成规则(必须严格遵守)】
你只能从以下 Cypher 模板中选择并填写,不得自行创造新结构。
【模板 1:疾病列表】
MATCH (d:Disease)
RETURN d.name
LIMIT 5
【模板 2:疾病 → 药物】
MATCH (d:Disease)-[:HAS_Drug]->(m:Drug)
WHERE d.name = "X"
RETURN m.name
【模板 3:症状 → 疾病】
MATCH (s:Symptom)<-[:HAS_SYMPTOM]-(d:Disease)
WHERE s.name = "X"
RETURN d.name
【模板 4:疾病 → 症状】
MATCH (d:Disease)-[:HAS_SYMPTOM]->(s:Symptom)
WHERE d.name = "X"
RETURN s.name
【模板 5:疾病 → 科室】
MATCH (d:Disease)-[:IS_OF_Department]->(k:Department)
WHERE d.name = "X"
RETURN k.name
【模板 6:人群 → 疾病】
MATCH (d:Disease)-[:IS_OF_AGE]->(a:Age)
WHERE a.name = "X"
RETURN d.name
约束:
1. 只输出一条 Cypher
2. MATCH 开头,RETURN 结尾
3. 不输出解释、不加 markdown
4. 若问题无法明确匹配模板,使用【模板 1】
"""
),
("human", "{question}")
])
chain = prompt | llm
# 5. 测试
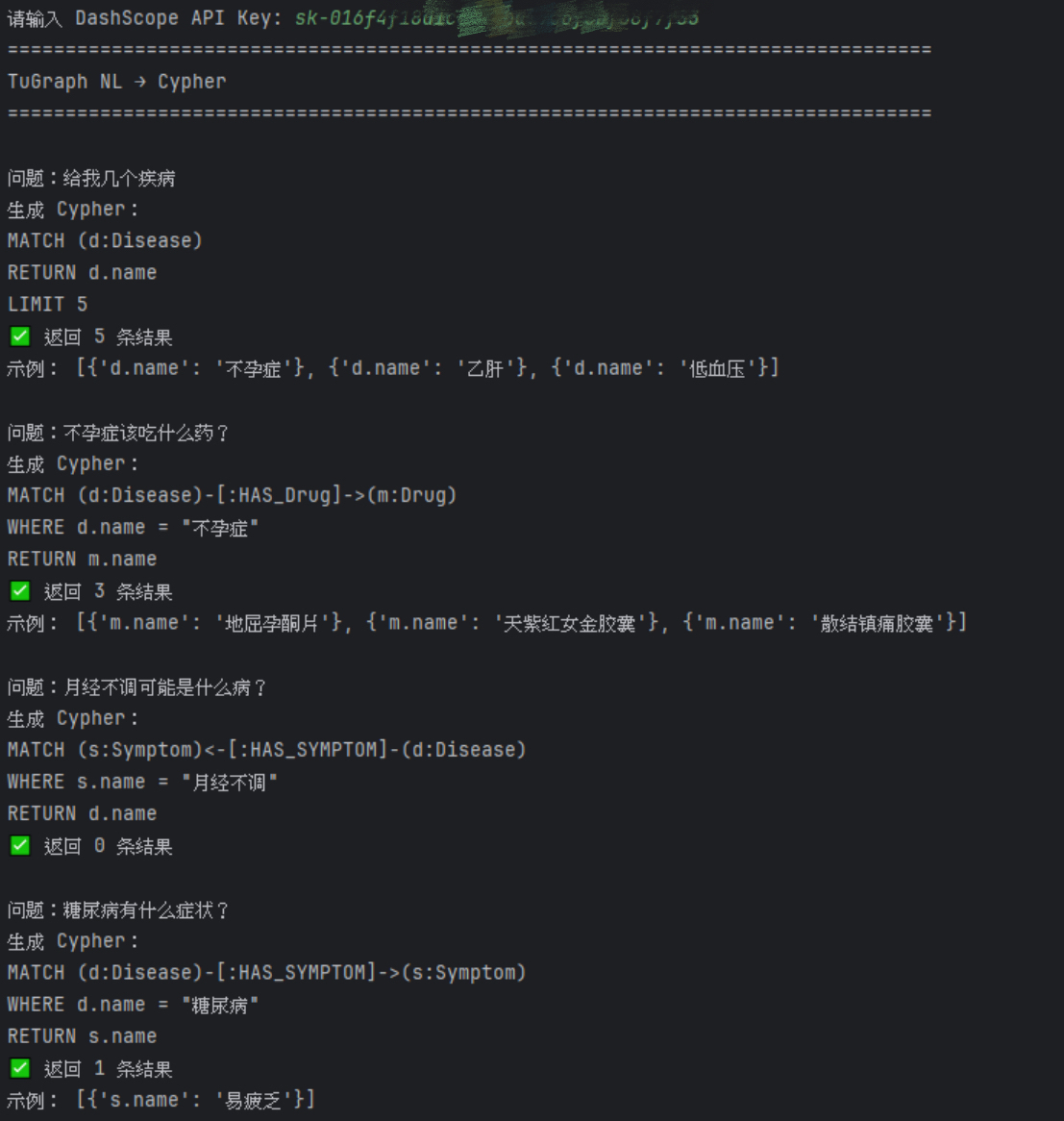
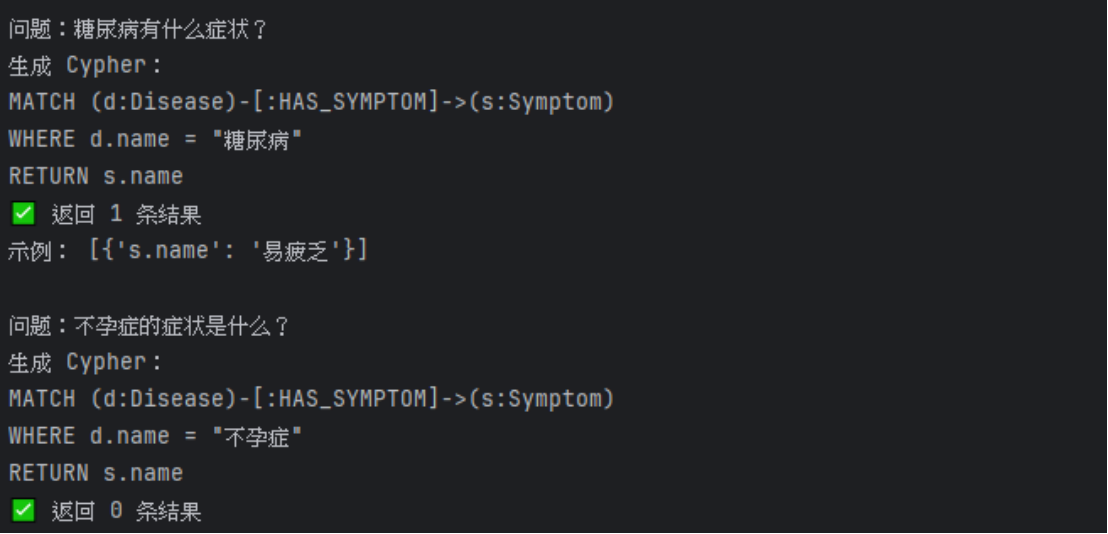
questions = [
"给我几个疾病",
"不孕症该吃什么药?",
"月经不调可能是什么病?",
"糖尿病有什么症状?",
"不孕症的症状是什么?"
]
print("=" * 80)
print("TuGraph NL → Cypher")
print("=" * 80)
for q in questions:
print(f"\n问题:{q}")
cypher = chain.invoke({"question": q}).content.strip()
print("生成 Cypher:")
print(cypher)
try:
data = run_cypher(cypher)
print(f"✅ 返回 {len(data)} 条结果")
if data:
print("示例:", data[:3])
except Exception as e:
print("❌ 执行失败:", e)在大模型交互层,采用更加严格和保守的 Prompt 设计策略,对可生成的 Cypher 语句进行约束,例如限定节点标签、关系类型以及可使用的属性字段。这种方式有效降低了模型生成非法或不兼容语句的概率,提高了查询成功率。实验过程中,通过逐步放宽 Prompt 约束,实现了从“简单节点查询”到“多实体关系查询”的渐进式测试。
四、实验总结
本实验围绕疾病文本数据,完成了从非结构化数据采集、结构化信息抽取,到疾病知识图谱构建及自然语言查询的完整流程。实验结果表明,大模型在疾病知识建模和图数据库交互任务中具有较高的实用价值,能够有效降低人工规则设计和查询编写的成本。通过 Neo4j 与 TuGraph 双平台的实践验证,实验进一步证明了大模型技术在不同图数据库系统中的可迁移性和适配潜力。
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容








所有评论(0)