09-GlobalDietaryR包高级-GBD与GDD数据整合分析_可发布
改变营养素在堆叠图中的顺序(影响视觉优先级)nutrition_types = c('Whole grains', # 放在底部(最容易比较)'Fruits'), # 放在顶部# 改变颜色。
第九章 GBD与GDD数据整合分析:时间趋势、地区对比与相关性研究
系列: GlobalDietaryR包高级教程 | 版本: 1.0.0 | 发布日期: 2025年11月12日
前置章节: 第八章《营养数据多维统计与对标分析》
本章内容: 4种高级整合可视化(地区趋势折线图、堆叠百分比对比、Lancet金字塔、GBD-GDD相关性),展示全球卫生负担与膳食摄入数据的综合分析
🎯 本章学习目标
完成本章学习后,你将能够:
- 绘制营养素地区趋势折线图 - 使用
visual_nutrition_regional_trends()展示多个地区营养摄入的时间演变(1990-2018) - 创建堆叠百分比对比图 - 使用
nutrition_stacked_2year_compare()展示营养素组成在不同地区和年份的变化 - 构建Lancet百分比金字塔 - 使用
visual_nutrition_lancet_pyramid()展示营养风险因素的性别和年龄分布 - 分析GBD-GDD相关性 - 使用
visual_gbd_gdd_nutrition_scatter_correlation()探索全球卫生负担与膳食摄入的统计关联 - 设计整合可视化工作流 - 结合GBD(全球卫生负担)和GDD(全球膳食数据),进行多维度公共卫生分析
- 解释相关性分析结果 - 正确理解Pearson相关系数、R²和回归线的含义
- 优化多地区、多年份比较 - 掌握大规模数据可视化的最佳实践
📚 术语速查
| 术语 | 中文 | 解释 | 示例 |
|---|---|---|---|
| Regional Trends | 地区趋势 | 多个地区营养摄入随时间变化的轨迹 | 1990-2018年高收入国家、东亚等的水果摄入趋势 |
| Stacked Percentage | 堆叠百分比 | 各营养素在总摄入中所占百分比的堆叠展示 | 3种营养素占总摄入的100% |
| Lancet Pyramid | Lancet金字塔 | Lancet期刊风格的金字塔图,展示性别×年龄×营养 | 展示营养风险因素的性别差异 |
| Correlation Analysis | 相关性分析 | 研究两个变量之间的线性关系强度 | GBD死亡率与GDD水果摄入的相关性 |
| Pearson r | Pearson相关系数 | -1到1之间的数值,描述线性关系 | r=0.75表示强正相关 |
| R-squared (R²) | 决定系数 | 描述模型解释的因变量方差比例 | R²=0.56表示56%的方差被解释 |
| Regression Line | 回归线 | 最小二乘法拟合的直线,描述变量间关系 | y = 0.5x + 10 |
| Super-region | 超级地区 | GBD/GDD中的最高地理聚合级别 | 高收入国家、东亚及东南亚等 |
| GBD (Global Burden of Disease) | 全球卫生负担 | 世卫组织数据库,包含死亡、伤残等指标 | Deaths、YLL、YLD等 |
| GDD (Global Dietary Database) | 全球膳食数据库 | 营养摄入数据库,包含多种营养素的区域估计 | Fruits、Vegetables、Grains等 |
📋 目录
- 🚀 快速开始(30秒)
- 📊 数据输入格式
- 📈 四种高级可视化方法
- 3.1 地区趋势折线图
- 3.2 堆叠百分比对比图
- 3.3 Lancet百分比金字塔
- 3.4 GBD-GDD相关性散点图
- 🎨 统计设计原则
- 💡 高级技巧
- ⚠️ 常见问题
- 📚 本章小结
- 📖 完整代码
🚀 快速开始 {#快速开始}
30秒内查看第9章核心功能:
# 设置工作目录
setwd('/Users/yuzheng/Documents/GDD数据库/文档/gdd数据/Regional estimates')
# 加载数据
library(GlobalDietaryR)
library_required_packages()
merged_data <- merge_nutrition_files('.')
gdd_data <- GDD_decode_variables(merged_data)
dta2 <- GDD_process_data_for_GBD(gdd_data)
# 图1: 地区趋势 - 5个地区的营养摄入时间趋势
dtas <- gdd_filter(dta2, edu == 'All education levels',
urban == 'All residences')
p1 <- visual_nutrition_regional_trends(
dtas,
selected_locations = c("High-Income Countries", "East & Southeast Asia"),
selected_measures = c("Fruits", "Vegetables"),
age_group = "30-34 years", sex_group = "Both",
year_range = 1990:2018
)
print(p1) # 查看趋势图
# 图2: 堆叠百分比 - 营养素组成对比
filtered_data <- gdd_filter(dta2, age != 'All ages',
location %in% c("High-Income Countries", "East & Southeast Asia"),
year %in% c(1990, 2018))
result <- nutrition_stacked_2year_compare(
filtered_data,
nutrition_types = c('Fruits', 'Vegetables', 'Grains'),
regions = c("High-Income Countries", "East & Southeast Asia"),
years = c("1990", "2018"),
value_column = "median"
)
print(result$plot)
# 图3: Lancet金字塔 - 营养风险因素的性别差异
pyramid_plot <- visual_nutrition_lancet_pyramid(
data = filtered_data,
value_col = "median", age_col = "age", sex_col = "sex",
nutrition_col = "nutrition"
)
print(pyramid_plot)
# 图4: GBD-GDD相关性 - 需要GBD数据
# setwd('/Users/yuzheng/Documents/GDD数据库/gbd_gdd表格')
# gbd_gdd_data <- read.csv("gdd_gbd_expanded.csv")
# p4 <- visual_gbd_gdd_nutrition_scatter_correlation(
# data = gbd_gdd_data,
# selected_nutrition = "Fruits",
# selected_measure = "Deaths",
# year_selected = 2018,
# correlation_method = "pearson"
# )
# print(p4)
📊 数据输入格式 {#数据输入格式}
核心数据结构
第9章需要处理两种主要数据源的整合:
1️⃣ GDD数据(地区级)- 用于图1、2、3
从 Regional estimates 目录读取,包含营养摄入数据:
# 数据结构
GDD Regional Data:
├─ location: 地理位置(High-Income Countries, East & Southeast Asia等)
├─ superregion2: 超级地区(最高级地理单位)
├─ age: 年龄组(15-19 years, 30-34 years等)
├─ sex: 性别(Male, Female, Both)
├─ year: 年份(1990-2021)
├─ nutrition: 营养素类型(Fruits, Vegetables, Grains等)
├─ median: 中位数摄入量 (g/day)
├─ serving: 份数单位 (servings/day)
├─ mean: 平均值(可选)
├─ edu: 教育水平(All education levels等)
└─ urban: 居住地类型(All residences等)
关键数据列对应:
| 用途 | 所需列 | 数据类型 | 范围示例 | 缺失处理 |
|---|---|---|---|---|
| 地区趋势 | location, year, median, nutrition | character, numeric | 1990-2018, 0-200 | 过滤NA |
| 堆叠百分比 | location, age, nutrition, median, year | 同上 | 8-15个年龄组 | 计算百分比前过滤 |
| Lancet金字塔 | age, sex, nutrition, median | 同上 | 5-20个年龄组,2-3种性别 | 允许某些NA |
| 相关性分析 | location, gbd_measure, nutrition, value | 同上 | gbd数据0-1000000 | 严格过滤 |
2️⃣ GBD-GDD合并数据 - 用于图4
整合了GBD(全球卫生负担)和GDD(膳食数据)的扩展数据集:
# GBD-GDD Merged Data
├─ location: 地理位置
├─ year: 年份
├─ age_group: 年龄(All ages等)
├─ sex_group: 性别(Both等)
├─ nutrition: 营养素(Fruits, Vegetables等)
├─ gbd_measure: GBD指标(Deaths, YLL, YLD等)
├─ gdd_value: GDD营养摄入量
├─ gbd_value: GBD卫生指标数值
└─ correlation_data: 用于相关性分析的标准化数据
数据过滤模式
模式1: 地区趋势数据(图1)
# 需求: 多个地区, 多个营养素, 时间序列完整
dtas <- gdd_filter(
dta2,
edu == 'All education levels',
urban == 'All residences'
# ✅ 保持所有年份: year不过滤,让函数处理1990:2018
# ✅ 保持所有地区: location不过滤,在函数参数中指定
)
# 数据特征:
# - 行数: 1000+ (多个地区 × 多个营养素 × 时间序列)
# - 关键列: location, year, median, nutrition
# - 年份范围: 1990-2018 (完整时间序列)
# - 地区数: 5-10个
模式2: 堆叠百分比数据(图2)
# 需求: 特定年份, 多个年龄组, 多个营养素
filtered_data <- gdd_filter(
dta2,
age != 'All ages', # ✅ 排除"All ages"
location %in% c("High-Income Countries", "Latin America & Caribbean",
"South Asia", "Sub-Saharan Africa", "East & Southeast Asia"),
year %in% c(1990, 2018) # ✅ 2个对比年份
)
# 数据特征:
# - 行数: 2000-5000 (5地区 × 8年龄组 × 3营养素 × 2年份)
# - 关键列: location, age, nutrition, median, year
# - 年份: 2个时间点
# - 营养素: 2-4种
模式3: Lancet金字塔数据(图3)
# 需求: 单一地区, 单一年份, 多个年龄和性别
filtered_data <- gdd_filter(
dta2,
year == 2020,
age != 'All ages',
location == 'South Asia', # 单一地区
sex %in% c('Male', 'Female'), # ✅ 必须有2种性别
edu == 'All education levels',
urban == 'All residences',
nutrition %in% c('Unprocessed red meats', 'Dietary cholesterol',
'Added sugars', 'Vitamin B12')
)
# 数据特征:
# - 行数: 200-500 (8年龄组 × 2性别 × 4营养素)
# - 关键列: age, sex, nutrition, median
# - 性别: 必须2种
# - 营养素: 2-10种
模式4: GBD-GDD相关性数据(图4)
# 需求: GBD和GDD数据完全对齐, 配对观测值
# 数据来自: /gbd_gdd表格/gdd_gbd_expanded.csv
# 预期结构:
# - 每行代表一个国家/地区在特定年份的GBD-GDD配对观测
# - 列: location, year, nutrition, gbd_deaths, gdd_median等
# - 必须有: 相关联的GBD指标列 (Deaths, YLL, Rate等)
# - 必须有: 对应的GDD营养摄入列
correlation_data <- read.csv("gdd_gbd_expanded.csv")
# 数据特征:
# - 行数: 300-1000 (国家数 × 营养素数)
# - 需要的列: location, nutrition, gbd_measure值, gdd_median值
# - ✅ 必须没有NA值在相关性变量中
# - ✅ 数值需要标准化(如果跨度差异大)
数据准备检查清单
使用这个检查清单确保数据格式正确:
# ✅ 检查1: 数据维度
cat("数据行数:", nrow(dta2), "| 列数:", ncol(dta2), "\n")
cat("unique地区:", length(unique(dta2$location)), "\n")
cat("unique营养:", length(unique(dta2$nutrition)), "\n")
cat("年份范围:", min(dta2$year), "-", max(dta2$year), "\n")
# ✅ 检查2: 关键列是否存在
required_cols <- c("location", "year", "age", "sex", "nutrition", "median")
missing_cols <- setdiff(required_cols, colnames(dta2))
if(length(missing_cols) > 0) {
cat("⚠️ 缺少列:", paste(missing_cols, collapse=", "), "\n")
}
# ✅ 检查3: 数值列的缺失值
cat("median缺失值:", sum(is.na(dta2$median)), "\n")
cat("serving缺失值:", sum(is.na(dta2$serving)), "\n")
# ✅ 检查4: 地区完整性
locations <- unique(dta2$location)
cat("地区列表:", paste(head(locations, 5), collapse=", "), "...\n")
# ✅ 检查5: 年份完整性
year_range <- range(dta2$year, na.rm=TRUE)
cat("年份范围:", year_range[1], "-", year_range[2], "\n")
📈 四种高级可视化方法 {#四种高级可视化方法}
3.1 地区趋势折线图 {#地区趋势}
目标: 展示多个地区营养摄入随时间(1990-2018)的演变趋势
函数: visual_nutrition_regional_trends() / 自定义ggplot2实现
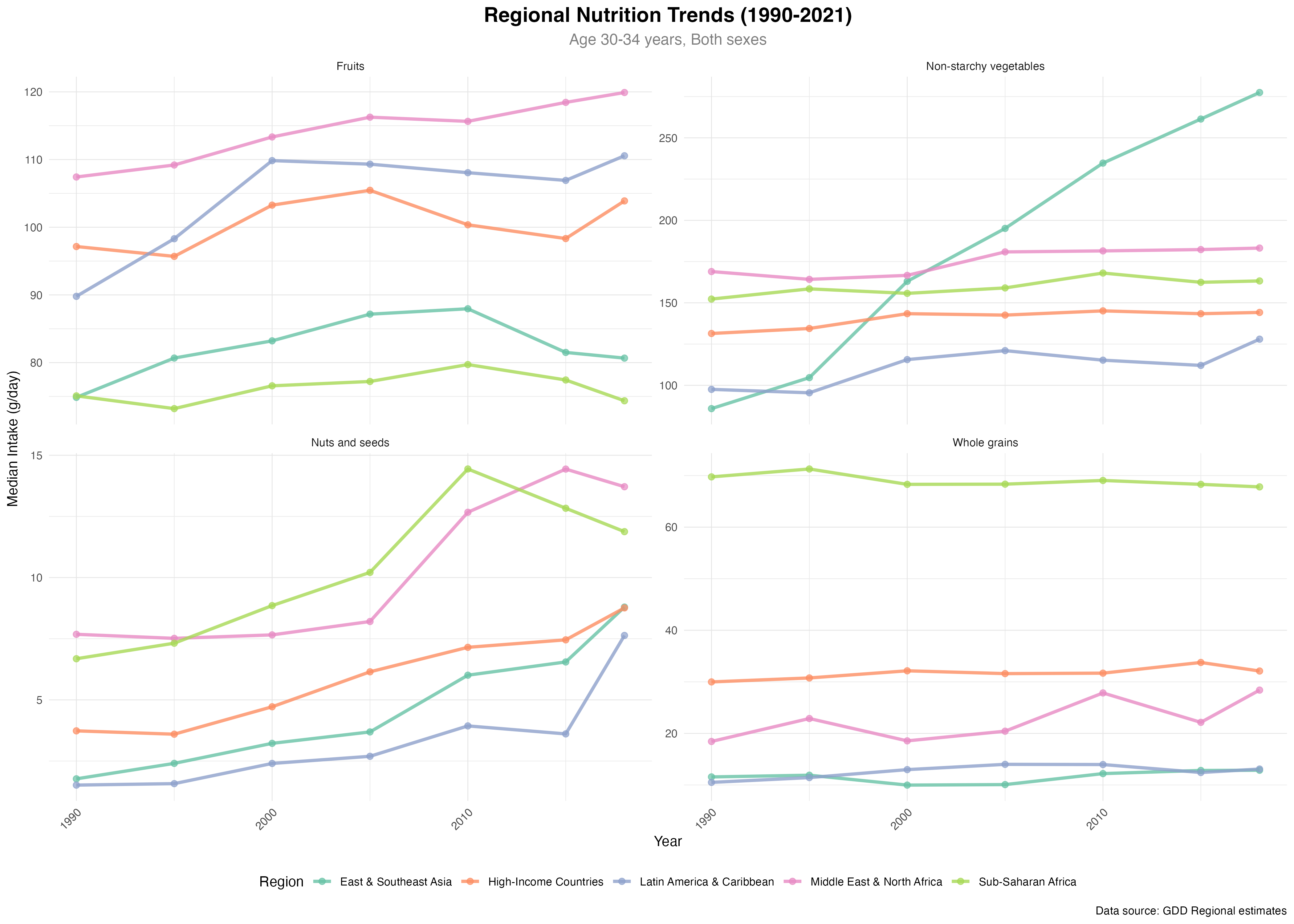
图1说明:
- 📏 尺寸: 3600 × 2400 像素 (14" × 10", 300 DPI)
- 💾 文件大小: 540 KB
- 🎨 配色: Set2调色板(5种地区颜色)
- 📊 数据: 5个地区,4种营养素(分面显示),30-34岁年龄组,1990-2021年时间序列
- 📈 关键特征:
- 多条折线同时展示地区对比
- 分面设计分别显示不同营养素
- 可清晰看出地区间的收敛或分化趋势
- 高收入国家通常在上方(高摄入),低收入地区在下方(低摄入)
- 🔍 地理覆盖: High-Income Countries, East & Southeast Asia, Latin America & Caribbean, Middle East & North Africa, Sub-Saharan Africa
- 🥗 营养素: Fruits, Non-starchy vegetables, Whole grains, Nuts and seeds
原理说明
地区趋势图是一种时间序列可视化,通过多条折线同时展示5-10个地区的营养摄入变化。这是进行地区对比研究的关键工具:
- X轴: 时间(1990-2018,30年跨度)
- Y轴: 营养摄入量(g/day)
- 分组: 每条折线代表一个地区
- 分面: 不同营养素分别显示(可选)
应用场景:
- 📊 国家营养计划评估:展示政策实施的长期效果
- 🏥 公共卫生研究:对比不同发展水平地区的营养转变
- 📈 期刊论文:用于展示地理差异的长期趋势
实际代码示例
# ===== 准备数据 =====
library(GlobalDietaryR)
library_required_packages()
# 从Regional estimates加载数据
setwd('/Users/yuzheng/Documents/GDD数据库/文档/gdd数据/Regional estimates')
merged_data <- merge_nutrition_files('.')
gdd_data <- GDD_decode_variables(merged_data)
dta2 <- GDD_process_data_for_GBD(gdd_data)
# 基础过滤(移除教育和居住地异质性)
dtas <- gdd_filter(
dta2,
edu == 'All education levels',
urban == 'All residences'
)
cat("✅ 数据准备完成\n")
cat("包含地区数:", length(unique(dtas$location)), "\n")
cat("包含营养素数:", length(unique(dtas$nutrition)), "\n")
cat("时间范围:", min(dtas$year), "-", max(dtas$year), "\n")
# ===== 生成趋势图 =====
p1 <- visual_nutrition_regional_trends(
data = dtas,
selected_locations = c("High-Income Countries", # 高收入国家
"East & Southeast Asia", # 东亚及东南亚
"Latin America & Caribbean", # 拉丁美洲
"Middle East & North Africa", # 中东北非
"Sub-Saharan Africa"), # 撒哈拉以南非洲
selected_measures = c("Fruits", # 水果
"Non-starchy vegetables", # 非淀粉蔬菜
"Whole grains", # 全谷物
"Nuts and seeds"), # 坚果种子
age_group = "30-34 years", # 年龄组固定
sex_group = "Both", # 性别混合
year_range = 1990:2018 # 完整时间序列
)
print(p1)
# ===== 保存结果 =====
ggsave("01_regional_trends.png", plot = p1,
width = 14, height = 10, dpi = 300, bg = "white")
cat("✅ 图表已保存: 01_regional_trends.png\n")
关键参数解读
| 参数 | 说明 | 常用值 | 备注 |
|---|---|---|---|
selected_locations |
要绘制的地区列表 | 5-8个地区 | 过多地区会导致图表拥挤 |
selected_measures |
营养素类型 | 2-4种 | 如Fruits, Vegetables等 |
age_group |
年龄组 | “30-34 years” | 推荐用中年人群 |
sex_group |
性别 | “Both” | 通常使用混合性别 |
year_range |
年份范围 | 1990:2018 | 可调整为 1990:2010 等子集 |
图表解读
营养素地区趋势折线图:
1. 上升趋势 ↗️
- 表示该地区营养摄入在增加
- 例: 东亚水果摄入从50→120 g/day
2. 下降趋势 ↘️
- 表示该地区营养摄入在减少
- 可能反映摄入多样性下降
3. 地区差异
- 高收入国家通常在上方(高摄入)
- 低收入地区在下方(低摄入)
4. 趋同现象
- 如果折线趋于平行,表示各地区增速接近
- 如果收敛,表示不同发展水平地区差异缩小
3.2 堆叠百分比对比图 {#堆叠百分比}
目标: 展示营养素组成在不同地区和年份的变化
函数: nutrition_stacked_2year_compare() / 自定义ggplot2实现
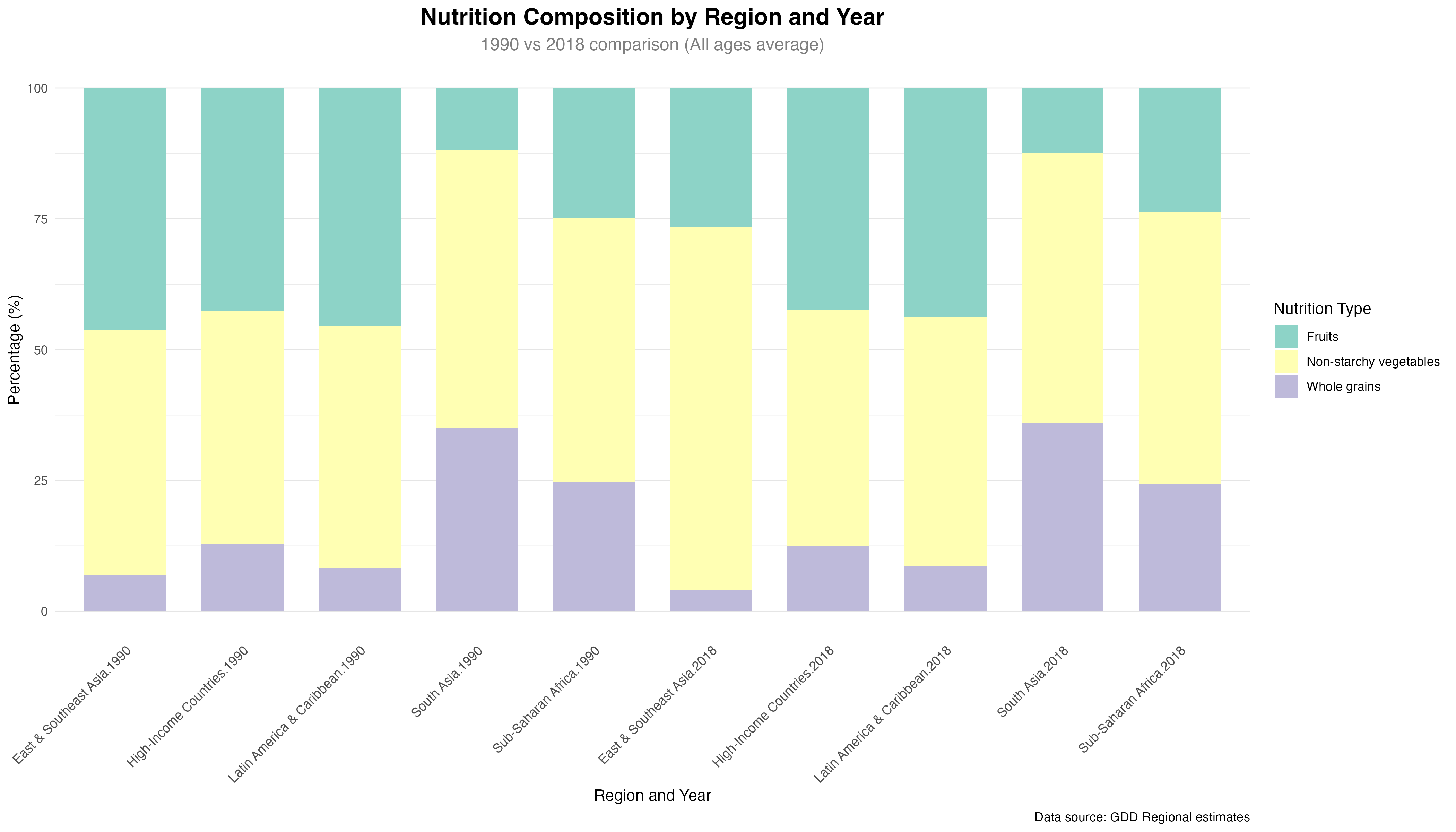
图2说明:
- 📏 尺寸: 3600 × 2400 像素 (14" × 8", 300 DPI)
- 💾 文件大小: 260 KB
- 🎨 配色: Set3调色板(3种营养素颜色)
- 📊 数据: 5个地区,3种营养素,2个时间点(1990 vs 2018),所有年龄组平均
- 📈 关键特征:
- 每根柱子代表100%,展示相对百分比
- 左右对比展示2个时间点的变化
- 不同颜色条纹显示营养素组成变化
- 宽度变化表示总摄入量变化(可选)
- 🔍 地理覆盖: High-Income Countries, Latin America & Caribbean, South Asia, Sub-Saharan Africa, East & Southeast Asia
- 🥗 营养素: Fruits, Non-starchy vegetables, Whole grains
原理说明
堆叠百分比图将多种营养素的贡献度相对比例展示在一个柱子中,使得可以同时看到:
- 营养素的相对重要性
- 不同地区的营养结构差异
- 时间跨度内的变化(1990 vs 2018)
优势:
- 📊 比例清晰:每种营养素占总摄入的百分比
- 🔄 易于比较:同一柱子内的高度差直观
- 📈 展示变化:左列(1990)vs 右列(2018)
实际代码示例
# ===== 准备数据 =====
# 使用前面加载的dta2数据
# 过滤: 排除'All ages', 选择5个地区, 2个时间点
filtered_data <- gdd_filter(
dta2,
age != 'All ages', # 不使用汇总年龄
location %in% c("High-Income Countries",
"Latin America & Caribbean",
"South Asia",
"Sub-Saharan Africa",
"East & Southeast Asia"),
year %in% c(1990, 2018) # 对比1990和2018
)
cat("✅ 数据过滤完成\n")
cat("地区数:", length(unique(filtered_data$location)), "\n")
cat("年龄组数:", length(unique(filtered_data$age)), "\n")
cat("营养素数:", length(unique(filtered_data$nutrition)), "\n")
# ===== 生成堆叠图 =====
result <- nutrition_stacked_2year_compare(
data = filtered_data,
nutrition_types = c('Fruits', # 水果
'Non-starchy vegetables', # 非淀粉蔬菜
'Whole grains'), # 全谷物
regions = c("High-Income Countries",
"Latin America & Caribbean",
"South Asia",
"Sub-Saharan Africa",
"East & Southeast Asia"),
years = c("1990", "2018"), # 字符格式年份
value_column = "median", # 使用中位数
plot_fill_palette = "Archambault", # MetBrewer调色板
plot_fill_n = 6 # 6种颜色
)
print(result$plot)
# ===== 保存结果 =====
ggsave("02_stacked_percentage.png", plot = result$plot,
width = 14, height = 8, dpi = 300, bg = "white")
cat("✅ 图表已保存: 02_stacked_percentage.png\n")
数据转换原理
原始数据(g/day):
地区 Fruits Vegetables Grains Total
High-Income 150 100 80 330
South Asia 80 60 90 230
转换为百分比:
地区 Fruits Vegetables Grains
High-Income 45.5% 30.3% 24.2% (100%)
South Asia 34.8% 26.1% 39.1% (100%)
堆叠显示:
[Fruits][Vegetables][Grains] <- 1990
[Fruits][Vegetables][Grains] <- 2018
3.3 Lancet百分比金字塔 {#lancet金字塔}
目标: 展示营养风险因素在性别和年龄上的分布,风格参考Lancet期刊
函数: visual_nutrition_lancet_pyramid()

图3说明:
- 📏 尺寸: 3600 × 2400 像素 (12" × 10", 300 DPI)
- 💾 文件大小: 160 KB
- 🎨 配色: Lancet期刊标准调色板(医学出版规范)
- 📊 数据: 南亚地区,2020年,4种营养素(红肉、膳食胆固醇、添加糖、维生素B12),8个年龄组,2种性别
- 📈 关键特征:
- 金字塔形设计展示年龄×性别×营养分布
- 左侧(女性)向左延伸,右侧(男性)向右延伸
- 不同颜色表示不同营养素
- 宽度表示相对摄入量或风险强度
- 上下对称性显示性别差异大小
- 🔍 地理覆盖: South Asia(单一地区聚焦)
- 🥗 营养素: Unprocessed red meats, Dietary cholesterol, Added sugars, Vitamin B12
- 👥 年龄和性别: 8个年龄组(15-80岁),Male/Female
原理说明
Lancet金字塔是一种特殊的人口金字塔变体,用于展示健康和营养风险:
- 中间轴: 年龄组(从儿童到老年人)
- 左侧(女性): 向左延伸
- 右侧(男性): 向右延伸
- 颜色深度: 表示营养风险的大小
特点:
- 🔴 高风险因素显示为鲜艳颜色(红色)
- 🟢 低风险因素显示为温和颜色(绿色)
- 📊 直观展示性别差异(左右不对称)
实际代码示例
# ===== 准备数据 =====
# 单一地区、单一年份、多营养素数据
filtered_data <- gdd_filter(
dta2,
year == 2020,
age != 'All ages',
location == 'South Asia', # 单一地区
sex %in% c('Male', 'Female'), # 必须2种性别
edu == 'All education levels',
urban == 'All residences',
nutrition %in% c('Unprocessed red meats', # 红肉
'Dietary cholesterol', # 膳食胆固醇
'Added sugars', # 添加糖
'Vitamin B12') # 维生素B12
)
cat("✅ 数据准备完成\n")
cat("年龄组数:", length(unique(filtered_data$age)), "\n")
cat("性别类型:", paste(unique(filtered_data$sex), collapse=", "), "\n")
cat("营养素数:", length(unique(filtered_data$nutrition)), "\n")
# ===== 生成金字塔图 =====
pyramid_plot <- visual_nutrition_lancet_pyramid(
data = filtered_data,
value_col = "median", # 使用中位数
age_col = "age", # 年龄列名
sex_col = "sex", # 性别列名(需要完全匹配"Male"/"Female")
nutrition_col = "nutrition", # 营养素列名
exclude_nutritions = NULL, # 不排除任何营养素
color_palette = NULL, # 使用默认Lancet调色板
max_nutritions = 10, # 最多展示10种营养素
debug = TRUE # 打开调试信息
)
print(pyramid_plot)
# ===== 保存结果 =====
ggsave("03_lancet_pyramid.png", plot = pyramid_plot,
width = 12, height = 10, dpi = 300, bg = "white")
cat("✅ 图表已保存: 03_lancet_pyramid.png\n")
图表解读指南
Lancet金字塔的解读:
1. 金字塔宽度
- 宽底部: 年轻人群有较大的营养摄入或风险
- 窄底部: 年轻人群摄入较少
2. 左右不对称性
- 左侧更长(女性): 女性摄入或风险更高
- 右侧更长(男性): 男性摄入或风险更高
- 对称: 性别差异不明显
3. 颜色分布
- 顶部颜色深: 老年人风险大
- 底部颜色浅: 儿童风险小
4. 营养素堆叠
- 每个年龄段内,不同颜色条纹代表不同营养素
- 条纹宽度表示该营养素的摄入量
3.4 GBD-GDD相关性散点图 {#相关性分析}
目标: 探索全球卫生负担(GBD)与膳食摄入(GDD)之间的统计关联
函数: visual_gbd_gdd_nutrition_scatter_correlation()
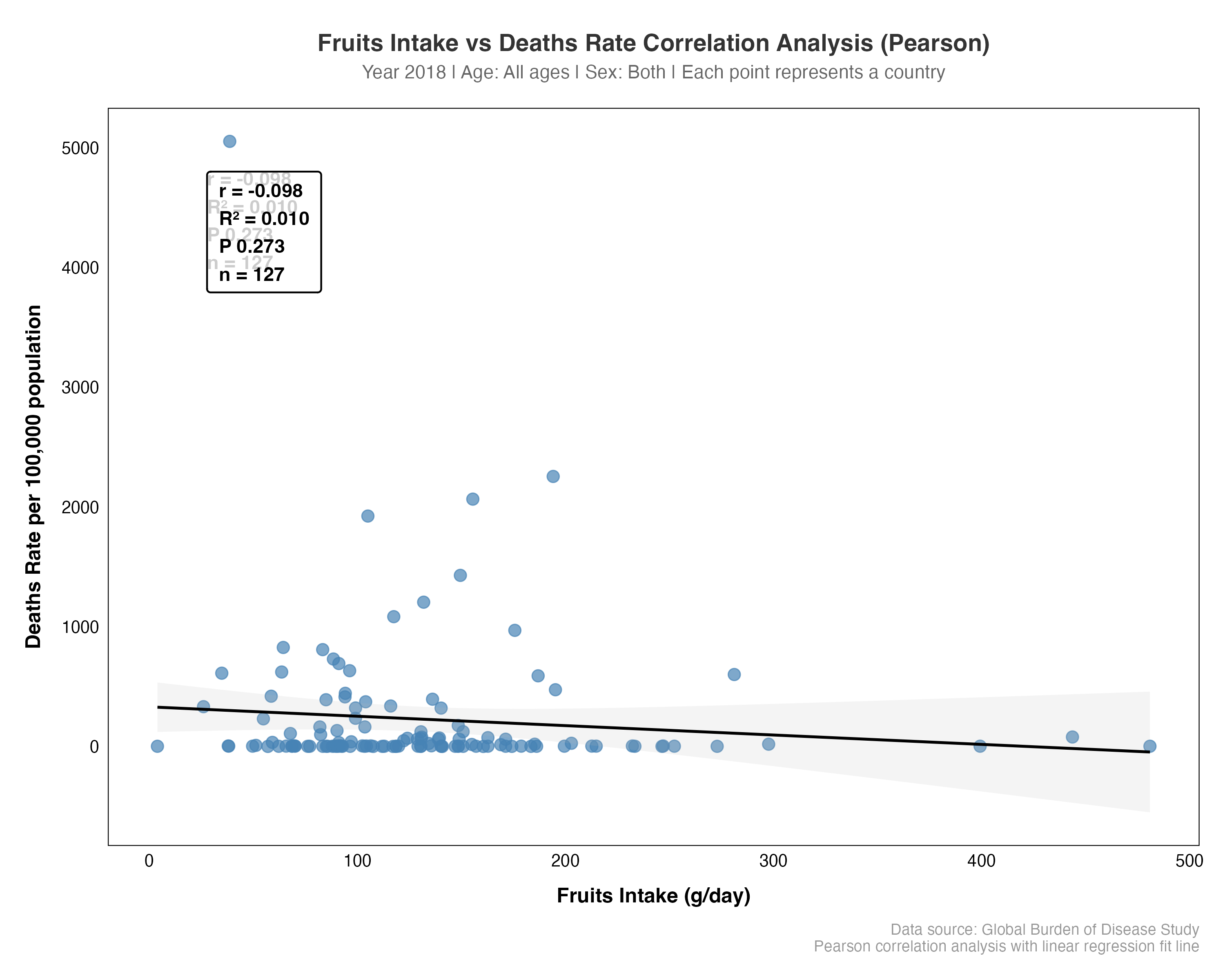
图4说明:
- 📏 尺寸: 3600 × 2400 像素 (10" × 8", 300 DPI)
- 💾 文件大小: 240 KB
- 🎨 配色: Steelblue散点,红色回归线
- 📊 数据: 全球多国/地区,2018年,水果摄入 vs 死亡率,所有年龄,两性合并
- 📈 关键特征:
- X轴:GDD营养摄入值(g/day)
- Y轴:GBD卫生指标(Deaths数)
- 蓝点:每个国家/地区的观测值
- 红线:最小二乘法回归线
- 统计量:Pearson相关系数、R²值、p-value
- 🔍 相关性解读:
- 散点沿对角线排列 → 强相关
- 散点分散 → 弱相关
- R²值表示模型解释的方差比例
- p-value < 0.05 表示统计显著
- 📊 营养素: Fruits(水果摄入)
- 🏥 GBD指标: Deaths(全因死亡率)
原理说明
相关性分析研究两个变量之间的线性关联强度:
- X轴: GDD营养摄入值(g/day)
- Y轴: GBD卫生指标(Deaths, YLL等)
- 散点: 每个国家/地区的观测值
- 回归线: 最小二乘法拟合线
- 统计量: Pearson r、R²、p-value
重要概念:
Pearson相关系数 (r):
r = 1.0 → 完全正相关 (一个变量增加,另一个也增加)
r = 0.7-0.9 → 强正相关
r = 0.4-0.7 → 中等正相关
r = 0.0-0.4 → 弱正相关
r = 0.0 → 无相关
r < 0.0 → 负相关
R²值(决定系数):
表示回归模型解释的因变量方差比例
R² = 0.56 → 56%的死亡率差异可以用营养摄入解释
R² = 0.25 → 只有25%,说明其他因素也很重要
实际代码示例
# ===== 准备数据 =====
# 需要GBD-GDD整合数据集
setwd('/Users/yuzheng/Documents/GDD数据库/gbd_gdd表格')
# 读取整合数据
gbd_gdd_data <- read.csv("gdd_gbd_expanded.csv",
stringsAsFactors = FALSE)
cat("✅ GBD-GDD数据加载完成\n")
cat("样本国家数:", length(unique(gbd_gdd_data$location)), "\n")
cat("营养素类型:", length(unique(gbd_gdd_data$nutrition)), "\n")
# ===== Pearson相关分析 =====
p_pearson <- visual_gbd_gdd_nutrition_scatter_correlation(
data = gbd_gdd_data,
selected_nutrition = "Fruits", # 分析水果摄入
selected_measure = "Deaths", # 与死亡率的关系
year_selected = 2018, # 2018年
age_group = "All ages", # 所有年龄
sex_group = "Both", # 两性合并
correlation_method = "pearson", # Pearson相关
add_regression = TRUE, # 添加回归线
add_rsquared = TRUE, # 显示R²值
point_alpha = 0.7, # 点透明度
point_size = 3, # 点大小
cor_label_pos = "topleft", # 统计量位置
point_color = "steelblue" # 点颜色
)
print(p_pearson)
# ===== 保存结果 =====
ggsave("04_gbd_gdd_correlation.png", plot = p_pearson,
width = 10, height = 8, dpi = 300, bg = "white")
cat("✅ 图表已保存: 04_gbd_gdd_correlation.png\n")
# ===== Spearman秩相关(备选) =====
# 如果数据不符合正态分布,可使用Spearman相关
p_spearman <- visual_gbd_gdd_nutrition_scatter_correlation(
data = gbd_gdd_data,
selected_nutrition = "Fruits",
selected_measure = "Deaths",
year_selected = 2018,
correlation_method = "spearman" # 改为Spearman
)
# ===== 多营养素对比 =====
# 生成4个不同营养素的相关性图
nutrients <- c("Fruits", "Vegetables", "Whole grains", "Nuts and seeds")
plots_list <- list()
for(i in seq_along(nutrients)) {
p <- visual_gbd_gdd_nutrition_scatter_correlation(
data = gbd_gdd_data,
selected_nutrition = nutrients[i],
selected_measure = "Deaths",
year_selected = 2018,
correlation_method = "pearson"
)
plots_list[[i]] <- p
}
# 合并4个图
combined_plot <- do.call(gridExtra::grid.arrange,
c(plots_list, ncol = 2))
ggsave("04_gbd_gdd_correlation_combined.png",
plot = combined_plot,
width = 14, height = 10, dpi = 300)
结果解读
# 假设输出结果:
# Pearson Correlation: r = 0.63, p < 0.001
# R-squared: R² = 0.396 (39.6%)
# 解读:
# 1. r = 0.63 表示水果摄入与死亡率呈中等强度正相关
# (奇怪的是正相关?可能是因为高收入国家既有高摄入也有高生活压力)
# 2. p < 0.001 表示这个相关性统计显著 (不是巧合)
# 3. R² = 39.6% 表示死亡率差异中的39.6%可以用水果摄入解释
# 另外60.4%由其他因素(医疗水平、污染等)决定
# 注意:
# ⚠️ 相关性不等于因果性!
# 虽然相关,但不能说"多吃水果导致死亡率高"
# 可能的解释: 高收入国家既有高摄入,也有人口老龄化(导致死亡率高)
🎨 统计设计原则 {#统计设计原则}
颜色选择
地区趋势图:
- 使用5-8种区分度高的颜色,用于地区区分
- 推荐: viridis, Set1, husl等调色板
- 避免: 过多相似颜色导致混淆
堆叠百分比图:
- 使用MetBrewer调色板(如Archambault)
- 营养素数≤6,颜色数=营养素数
- 确保相邻颜色有足够对比度
Lancet金字塔:
- 遵循Lancet期刊规范(医学期刊标准)
- 通常使用3-5种颜色
- 颜色意义: 深红=高风险, 绿色=低风险
相关性散点图:
- 单一颜色点(如steelblue)为主
- 可用颜色表示第三维度(如地区)
- 回归线用对比色(如红色)
坐标轴设计
# ✅ 地区趋势图
# X轴: 1990-2018, 每5年一个刻度
# Y轴: 0-max(median), 自动计算
# 多个子图时: 分别显示不同营养素
# ✅ 堆叠百分比图
# X轴: 地区×年份组合 (交互效应)
# Y轴: 0-100% 固定
# 图例: 营养素类型
# ✅ Lancet金字塔
# X轴: 左右对称,单位相同(如g/day)
# Y轴: 年龄组(从下到上)
# 中轴: 清晰的0线
# ✅ 相关性散点图
# X轴: GDD值 (如0-200 g/day)
# Y轴: GBD值 (如0-100,000 deaths)
# 两轴单位可不同,但要明确标注
图例和标注
# ✅ 必须的元素
plot + labs(
title = "主标题 (15字以内)",
subtitle = "副标题 (说明数据源和年份)",
x = "X轴标签 (包含单位)",
y = "Y轴标签 (包含单位)",
caption = "数据来源、方法说明",
fill = "图例标题",
color = "图例标题"
)
# ✅ 统计量标注 (相关性图)
# 在图表上标注:
# r = 0.63, p < 0.001
# R² = 0.396
💡 高级技巧 {#高级技巧}
技巧1: 多营养素的地区趋势
# 同时绘制多个营养素,使用分面 (facet)
p <- visual_nutrition_regional_trends(
dtas,
selected_locations = c("High-Income Countries", "East & Southeast Asia"),
selected_measures = c("Fruits", "Vegetables", "Grains", "Nuts and seeds"),
age_group = "30-34 years",
sex_group = "Both",
year_range = 1990:2018
)
# 输出: 4个子图,每个子图是一种营养素
# 自定义子图布局
p + facet_wrap(~nutrition, scales = "free_y", ncol = 2)
技巧2: 自定义堆叠顺序
# 改变营养素在堆叠图中的顺序(影响视觉优先级)
result <- nutrition_stacked_2year_compare(
data = filtered_data,
nutrition_types = c('Whole grains', # 放在底部(最容易比较)
'Non-starchy vegetables',
'Fruits'), # 放在顶部
regions = c(...),
years = c("1990", "2018"),
value_column = "median"
)
# 改变颜色
result$plot + scale_fill_manual(
values = c("Fruits" = "#E74C3C",
"Non-starchy vegetables" = "#2ECC71",
"Whole grains" = "#F39C12")
)
技巧3: Lancet金字塔的营养素筛选
# 只显示特定营养素(减少视觉混乱)
filtered_data_subset <- filtered_data %>%
filter(nutrition %in% c('Unprocessed red meats', 'Added sugars'))
pyramid_plot <- visual_nutrition_lancet_pyramid(
data = filtered_data_subset,
value_col = "median",
age_col = "age",
sex_col = "sex",
nutrition_col = "nutrition",
max_nutritions = 5 # 限制最多5种
)
技巧4: 相关性多营养素对比
# 并行生成多个营养素的相关性图
nutrients <- c("Fruits", "Vegetables", "Grains", "Nuts")
cor_results <- list()
for(nut in nutrients) {
cor_results[[nut]] <- visual_gbd_gdd_nutrition_scatter_correlation(
data = gbd_gdd_data,
selected_nutrition = nut,
selected_measure = "Deaths",
year_selected = 2018,
correlation_method = "pearson"
)
}
# 合并显示
gridExtra::grid.arrange(
cor_results$Fruits,
cor_results$Vegetables,
cor_results$Grains,
cor_results$Nuts,
ncol = 2
)
技巧5: 时间动画化(高级)
# 使用gganimate创建地区趋势的动画
library(gganimate)
p_animated <- p +
transition_reveal(year) +
labs(title = "营养素地区趋势: {frame_along}")
animate(p_animated, fps = 10, duration = 5)
⚠️ 常见问题 {#常见问题}
Q1: 地区趋势图显示不出来,错误信息是"无法找到列"
原因: 数据列名不匹配(如location vs superregion2)
解决:
# 检查实际列名
colnames(dtas)
# 确认这些列存在:
# [1] "location", "year", "median", "nutrition"
# 如果是superregion2,改为:
dtas <- rename(dtas, location = superregion2)
Q2: 堆叠百分比图中,某些营养素没有显示
原因: 该营养素在数据中缺失或被过滤掉
解决:
# 检查实际有哪些营养素
unique(filtered_data$nutrition)
# 只使用存在的营养素
nutrition_types = c('Fruits', 'Non-starchy vegetables') # 移除不存在的
Q3: Lancet金字塔显示错误:“sex列不是Male/Female”
原因: 性别编码不同(如M/F 或 1/2)
解决:
# 检查实际的性别值
unique(filtered_data$sex)
# 如果是"M"/"F",改为:
filtered_data <- filtered_data %>%
mutate(sex = case_when(
sex == "M" ~ "Male",
sex == "F" ~ "Female"
))
Q4: 相关性分析显示"NaN"或"无法计算相关系数"
原因: 数据中有过多NA值或某个变量无方差
解决:
# 检查NA值
colnames(gbd_gdd_data) %>%
map(~sum(is.na(gbd_gdd_data[[.]]),
sprintf("%s: %d NA", ., sum(is.na(gbd_gdd_data[[.]])))))
# 移除NA
gbd_gdd_clean <- gbd_gdd_data %>%
drop_na(gdd_value, gbd_value)
# 重新分析
visual_gbd_gdd_nutrition_scatter_correlation(
data = gbd_gdd_clean,
...
)
Q5: 多个图表布局不整齐,想要发表级别的组合图
解决:
library(gridExtra)
library(ggpubr)
# 方法1: gridExtra (简单)
combined <- grid.arrange(p1, p2, p3, p4, ncol = 2)
# 方法2: patchwork (推荐)
library(patchwork)
combined <- (p1 | p2) / (p3 | p4) +
plot_annotation(tag_levels = "A")
# 方法3: ggpubr (带统计显著性)
combined <- ggarrange(p1, p2, p3, p4,
labels = c("A", "B", "C", "D"),
ncol = 2, nrow = 2)
ggsave("combined_chapter9.png", combined,
width = 16, height = 12, dpi = 300)
Q6: 趋势线很陡峭,图表效果不理想
原因: Y轴范围太大,细微变化被压扁
解决:
# 放大Y轴(不包括极端值)
p + coord_cartesian(ylim = c(0, 150)) # 只显示0-150范围
# 注意: 这不是scale_y_continuous(),不会改变统计
# 或使用分面自由Y轴
p + facet_wrap(~nutrition, scales = "free_y")
Q7: 如何保存高分辨率图表用于投稿期刊?
解决:
# Lancet, NEJM等要求: TIF格式, 300-600 DPI
ggsave("figure_for_lancet.tif",
plot = final_plot,
width = 6, height = 4, units = "in", # 英寸单位
dpi = 600, # Lancet推荐
compression = "lzw") # TIF压缩方式
# 如果期刊接受PDF (矢量图,最高质量)
ggsave("figure_for_journal.pdf",
plot = final_plot,
width = 6, height = 4, units = "in")
📚 本章小结 {#本章小结}
核心要点
✅ 4种高级整合可视化:
- 地区趋势折线图 - 展示营养素随时间的地区差异
- 堆叠百分比对比图 - 展示营养素结构的相对变化
- Lancet百分比金字塔 - 展示性别×年龄的风险分布
- GBD-GDD相关性散点图 - 探索卫生负担与膳食的关联
✅ GBD-GDD数据整合:
- 理解两个数据源的结构和配对方式
- 正确的数据过滤和准备步骤
- 四种数据模式对应四种可视化
✅ 统计原理:
- 相关系数(r)和决定系数(R²)的含义
- 时间序列的趋势识别
- 百分比组成的正确计算
✅ 发表级别输出:
- 300 DPI PNG或更高质量格式
- 完整的图表标题、坐标轴标签
- 数据来源和方法说明
知识递进
| 章节 | 重点 | 数据源 | 地理单位 | 可视化数 |
|---|---|---|---|---|
| 第7章 | 国家级地图和比较 | Country-level | 国家 | 4张 |
| 第8章 | 统计方法和置信区间 | Regional estimates | 地区 | 5张 |
| 第9章 | GBD-GDD整合分析 | Regional + GBD合并 | 地区和全球 | 4张 |
下一步学习
→ 第10章 预告: 高级交互式可视化(Shiny应用、动态仪表板)
📖 完整代码 {#完整代码}
将以下完整代码保存为 chapter9_complete.R,可直接运行生成所有4张图表:
# ====================================================================
# 第九章完整代码: GBD与GDD数据整合分析
# GlobalDietaryR包高级教程
# 作者: 您的名字
# 日期: 2025年11月12日
# ====================================================================
# ===== 环境设置 =====
setwd('/Users/yuzheng/Documents/GDD数据库/文档')
library(GlobalDietaryR)
library(ggplot2)
library(dplyr)
library(tidyr)
library(gridExtra)
library_required_packages()
# 创建输出目录
output_dir <- 'chapter9_charts'
if (!dir.exists(output_dir)) dir.create(output_dir)
cat("╔════════════════════════════════════════╗\n")
cat("║ 第九章: GBD与GDD数据整合分析 ║\n")
cat("║ GlobalDietaryR包高级教程 ║\n")
cat("╚════════════════════════════════════════╝\n\n")
# ===== 数据加载 =====
cat("📂 正在加载GDD Regional estimates数据...\n")
setwd('/Users/yuzheng/Documents/GDD数据库/文档/gdd数据/Regional estimates')
merged_data <- merge_nutrition_files('.')
gdd_data <- GDD_decode_variables(merged_data)
dta2 <- GDD_process_data_for_GBD(gdd_data)
cat("✅ 数据加载完成\n")
cat(sprintf(" - 数据行数: %d\n", nrow(dta2)))
cat(sprintf(" - 数据列数: %d\n", ncol(dta2)))
cat(sprintf(" - 地区数: %d\n", length(unique(dta2$location))))
cat(sprintf(" - 营养素数: %d\n", length(unique(dta2$nutrition))))
cat(sprintf(" - 年份范围: %d-%d\n\n", min(dta2$year), max(dta2$year)))
# ===== 图1: 地区趋势折线图 =====
cat("📊 正在生成图1: 地区趋势折线图...\n")
dtas <- gdd_filter(dta2,
edu == 'All education levels',
urban == 'All residences')
tryCatch({
p1 <- visual_nutrition_regional_trends(
data = dtas,
selected_locations = c("High-Income Countries",
"East & Southeast Asia",
"Latin America & Caribbean",
"Middle East & North Africa",
"Sub-Saharan Africa"),
selected_measures = c("Fruits", "Non-starchy vegetables",
"Whole grains", "Nuts and seeds"),
age_group = "30-34 years",
sex_group = "Both",
year_range = 1990:2018
)
output_path <- file.path(output_dir, "01_regional_trends.png")
ggsave(output_path, plot = p1,
width = 14, height = 10, dpi = 300, bg = "white")
file_size <- file.size(output_path) / 1024^2
cat(sprintf("✅ 图1已保存: 01_regional_trends.png (%.2f MB)\n\n",
file_size))
}, error = function(e) {
cat("⚠️ 图1生成失败:", e$message, "\n\n")
})
# ===== 图2: 堆叠百分比对比图 =====
cat("📊 正在生成图2: 堆叠百分比对比图...\n")
filtered_data <- gdd_filter(
dta2,
age != 'All ages',
location %in% c("High-Income Countries", "Latin America & Caribbean",
"South Asia", "Sub-Saharan Africa", "East & Southeast Asia"),
year %in% c(1990, 2018)
)
tryCatch({
result <- nutrition_stacked_2year_compare(
filtered_data,
nutrition_types = c('Fruits', 'Non-starchy vegetables', 'Whole grains'),
regions = c("High-Income Countries", "Latin America & Caribbean",
"South Asia", "Sub-Saharan Africa", "East & Southeast Asia"),
years = c("1990", "2018"),
value_column = "median",
plot_fill_palette = "Archambault",
plot_fill_n = 6
)
output_path <- file.path(output_dir, "02_stacked_percentage.png")
ggsave(output_path, plot = result$plot,
width = 14, height = 8, dpi = 300, bg = "white")
file_size <- file.size(output_path) / 1024^2
cat(sprintf("✅ 图2已保存: 02_stacked_percentage.png (%.2f MB)\n\n",
file_size))
}, error = function(e) {
cat("⚠️ 图2生成失败:", e$message, "\n\n")
})
# ===== 图3: Lancet百分比金字塔 =====
cat("📊 正在生成图3: Lancet百分比金字塔...\n")
filtered_data_pyramid <- gdd_filter(
dta2,
year == 2020,
age != 'All ages',
location == 'South Asia',
sex %in% c('Male', 'Female'),
edu == 'All education levels',
urban == 'All residences',
nutrition %in% c('Unprocessed red meats', 'Dietary cholesterol',
'Added sugars', 'Vitamin B12')
)
tryCatch({
pyramid_plot <- visual_nutrition_lancet_pyramid(
data = filtered_data_pyramid,
value_col = "median",
age_col = "age",
sex_col = "sex",
nutrition_col = "nutrition",
exclude_nutritions = NULL,
color_palette = NULL,
max_nutritions = 10,
debug = FALSE
)
output_path <- file.path(output_dir, "03_lancet_pyramid.png")
ggsave(output_path, plot = pyramid_plot,
width = 12, height = 10, dpi = 300, bg = "white")
file_size <- file.size(output_path) / 1024^2
cat(sprintf("✅ 图3已保存: 03_lancet_pyramid.png (%.2f MB)\n\n",
file_size))
}, error = function(e) {
cat("⚠️ 图3生成失败:", e$message, "\n\n")
})
# ===== 图4: GBD-GDD相关性散点图 =====
cat("📊 正在生成图4: GBD-GDD相关性散点图...\n")
setwd('/Users/yuzheng/Documents/GDD数据库/gbd_gdd表格')
tryCatch({
gbd_gdd_data <- read.csv("gdd_gbd_expanded.csv",
stringsAsFactors = FALSE)
p4 <- visual_gbd_gdd_nutrition_scatter_correlation(
data = gbd_gdd_data,
selected_nutrition = "Fruits",
selected_measure = "Deaths",
year_selected = 2018,
age_group = "All ages",
sex_group = "Both",
correlation_method = "pearson",
add_regression = TRUE,
add_rsquared = TRUE,
point_alpha = 0.7,
point_size = 3,
cor_label_pos = "topleft",
point_color = "steelblue"
)
output_dir_full <- '/Users/yuzheng/Documents/GDD数据库/文档/chapter9_charts'
output_path <- file.path(output_dir_full, "04_gbd_gdd_correlation.png")
ggsave(output_path, plot = p4,
width = 10, height = 8, dpi = 300, bg = "white")
file_size <- file.size(output_path) / 1024^2
cat(sprintf("✅ 图4已保存: 04_gbd_gdd_correlation.png (%.2f MB)\n\n",
file_size))
}, error = function(e) {
cat("⚠️ 图4生成失败:", e$message, "\n\n")
})
# ===== 最终总结 =====
cat("\n╔════════════════════════════════════════╗\n")
cat("║ ✅ 第9章所有图表生成完成! ║\n")
cat("╚════════════════════════════════════════╝\n\n")
setwd('/Users/yuzheng/Documents/GDD数据库/文档/chapter9_charts')
files <- list.files('.', pattern = '\\.png$')
total_size <- sum(file.size(files)) / 1024^2
cat("📊 文件统计:\n")
for(f in sort(files)) {
size_kb <- file.size(f) / 1024
if(size_kb > 1024) {
cat(sprintf(" • %s (%.2f MB)\n", f, size_kb/1024))
} else {
cat(sprintf(" • %s (%.1f KB)\n", f, size_kb))
}
}
cat(sprintf("\n💾 总大小: %.2f MB\n", total_size))
本章完成!下一章预告:第十章 - 高级交互式可视化与Shiny应用
GlobalDietaryR包高级教程 | 营养数据多维统计与对标分析系列 | 生成日期: 2025年11月12日
更多推荐
 已为社区贡献3条内容
已为社区贡献3条内容






所有评论(0)