【医学影像 AI】用于可泛化疾病检测的视网膜图像基础模型
更多内容请关注【医学影像 AI by youcans@Xidian 专栏】
【医学影像 AI】用于可泛化疾病检测的视网膜图像基础模型
0. 论文简介
0.1 基本信息
2023年,Zhou, Y.等 在 Nature 发布论文 【用于可泛化疾病检测的视网膜图像基础模型】(A foundation model for generalizable disease detection from retinal images)。
本文提出基于自监督学习的 视网膜图像基础模型 RETFound,该模型通过在 160 万张未标注的视网膜图像(CFP 和 OCT)上训练,在眼部疾病诊断、眼部疾病预后及全身性疾病预测任务中以更少标签数据持续优于对比模型,具备良好标签效率、泛化性与可解释性,为医学 AI 临床应用提供新方案。
论文下载: Nature, rmaphoh
项目地址: Github
数据下载: RETFound_MAE
引用格式:
Zhou, Y., Chia, M.A., Wagner, S.K. et al. A foundation model for generalizable disease detection from retinal images. Nature 622, 156–163 (2023). https://doi.org/10.1038/s41586-023-06555-x

0.2 论文概览
本文于 2023 年发表在《Nature》,提出基于自监督学习(SSL)的 RETFound 视网膜图像基础模型。该模型在眼部疾病诊断(如糖尿病视网膜病变、青光眼)、眼部疾病预后(1 年内对侧眼发展为湿性 AMD)及全身性疾病预测(3 年内心力衰竭、心肌梗死等)任务中,以更少标签数据持续优于 SL-ImageNet、SSL-ImageNet 等对比模型,同时具备良好的标签效率、泛化性和可解释性,已公开供研究使用。
- 模型核心架构与训练流程
- 数据规模:
共 160 万张无标签视网膜图像:904,170 张 CFP(90.2% 来自 MEH-MIDAS,9.8% 来自 EyePACS)、736,442 张 OCT(85.2% 来自 MEH-MIDAS,14.8% 来自公开数据)。 - 基础架构:
基于掩码自编码器(MAE):编码器为 ViT-large(24 个 Transformer 块,嵌入维度 1024),解码器为 ViT-small(8 个 Transformer 块,嵌入维度 512) - 训练策略:
两阶段 SSL 训练:1. 先在 ImageNet-1k(140 万张自然图像)用 MAE 预训练;2. 再在视网膜图像上继续 SSL 训练(CFP 掩码率 75%,OCT 掩码率 85%) - 计算资源:
预训练:8 张 NVIDIA Tesla A100(40GB)GPU,耗时 14 天;微调:1 张 NVIDIA Tesla T4(16GB)GPU,1000 张图像约需 70 分钟。
-
模型性能验证
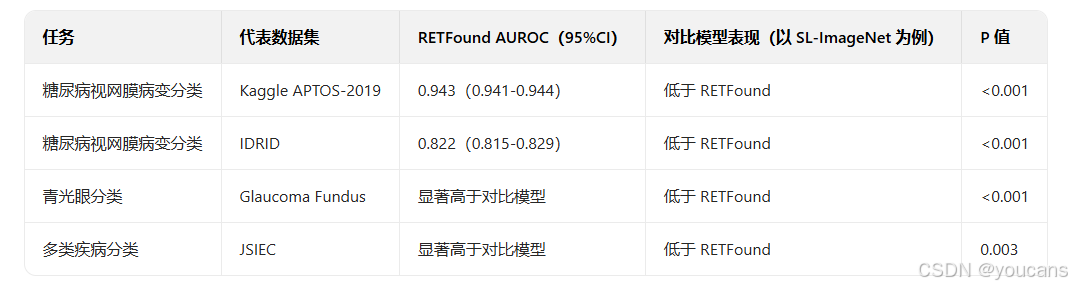
-
模型可解释性与鲁棒性
- 可解释性:
- 图像重建:RETFound 能从 75% 掩码的 CFP/OCT 图像中重建关键解剖结构(如 CFP 的视神经和大血管、OCT 的神经纤维层),证明其学习到视网膜特异性特征。
- 热图分析(RELPROP 工具):诊断眼部疾病时,可识别明确病变(如糖尿病视网膜病变的硬性渗出、青光眼的视盘周围萎缩);预测全身性疾病时,重点关注与疾病相关的结构(如视神经、神经纤维层)。
- 鲁棒性:
- 年龄混淆因素:在心肌梗死预测中,即使控制组与疾病组年龄差异缩小(从 5.3 岁降至 0),RETFound 仍保持稳定性能,而其他模型和单纯年龄逻辑回归性能下降。
- 跨种族泛化:在白人、亚裔、黑人等亚组中,RETFound 对心力衰竭、心肌梗死等疾病的预测 AUROC 均显著高于对比模型(P<0.001)。
- 数据与代码
PyTorch 版(https://github.com/rmaphoh/RETFound_MAE)
Keras 版(https://github.com/uw-biomedical-ml/RETFound_MAE)
0.3 摘要
医学人工智能(AI)在视网膜图像中识别健康状况迹象、加速眼部疾病及全身性疾病诊断方面具有巨大潜力 ¹。然而,AI 模型的开发需要大量标注数据,且这些模型通常具有任务特异性,在不同临床应用中的泛化能力有限 ²。
本文提出了一种用于视网膜图像的基础模型 RETFound,该模型通过无标签视网膜图像学习具有泛化性的特征表示,为多种应用场景下标签高效的模型适配提供基础。
具体而言,RETFound 采用自监督学习方法,在 160 万张无标签视网膜图像上完成训练,随后适配到带有明确标签的疾病检测任务中。
研究表明,经过适配的 RETFound 在威胁视力的眼部疾病诊断与预后评估,以及心力衰竭、心肌梗死等复杂全身性疾病的发病预测任务中,即便使用更少的标注数据,也能持续优于多个对比模型。
RETFound 为提升模型性能、减轻专家标注工作量提供了一种具有泛化性的解决方案,有望推动视网膜成像在临床 AI 领域的广泛应用。
1. 主要工作
近年来,随着深度学习技术的显著发展,医学人工智能(AI)已取得重大进展 ¹、³、⁴。例如,在多种应用场景中,深度神经网络的准确率已达到甚至超过临床专家水平⁵,如为威胁视力的视网膜疾病提供转诊建议⁶、检测胸部 X 光图像中的病变⁷等。这类模型的开发通常需要大量高质量的标注数据,而标注过程不仅需要专家参与评估,还涉及繁重的工作量 ¹、²。然而,具备领域知识的专家数量稀缺,无法满足这种高需求,导致大量医疗数据处于未标注、未利用的状态。
自监督学习(SSL)旨在通过直接从数据中提取监督信号来缓解数据效率低下的问题,而非依赖标签获取专家知识⁸、⁹、¹⁰、¹¹。自监督学习会训练模型完成 “pretext 任务”( pretext task,即预训练任务),这类任务无需人工标注标签,或可自动生成标签。该过程能利用海量未标注数据学习通用特征表示,且这些特征可轻松适配更具体的任务。完成预训练阶段后,模型会针对分类、分割等特定下游任务进行微调。在各类计算机视觉任务中,即便仅使用少量数据进行微调,自监督学习模型的性能仍优于基于监督学习的迁移学习模型(例如,利用 ImageNet¹² 数据集及分类标签对模型进行预训练)¹³、¹⁴。除标签效率优势外,在测试来自不同领域的新数据时,基于自监督学习的模型表现也优于监督学习模型 ¹⁵、¹⁶。自监督学习模型的特征表示具有强大泛化能力,且微调后在众多下游任务中能实现高性能,这些优势表明,在医疗 AI 领域 —— 该领域数据丰富、医疗任务多样但标签稀缺 ¹、⁸—— 自监督学习具备巨大应用潜力。
彩色眼底摄影(CFP)和光学相干断层扫描(OCT)是眼科最常用的成像方式,在常规临床实践中,这类视网膜图像的积累速度极快。除呈现眼部疾病相关的临床特征外,这些图像还能为研究全身性疾病提供宝贵线索,该领域近年来被称为 “眼组学(oculomics)”¹⁷、¹⁸。例如,视神经和视网膜内层可作为中枢神经系统组织的无创观察窗口 ¹⁹、²⁰、²¹,进而为研究神经退行性疾病提供途径;同样,视网膜血管形态能为心脏、肾脏等其他血管器官系统的研究提供参考 ²²、²³、²⁴、²⁵。尽管已有多项研究表明,自监督学习可提升单一眼部疾病检测任务的性能,如糖尿病黄斑水肿诊断 ²⁶、年龄相关性黄斑变性(AMD)诊断 ²⁷、可转诊糖尿病视网膜病变检测 ²⁸、²⁹、³⁰等,但目前鲜有研究证实,单一自监督预训练模型能够泛化到多种复杂任务中。这一领域的进展可能受到以下因素阻碍:难以构建大规模视网膜图像库,且该图像库需与多种相关疾病结局建立广泛关联。此外,不同自监督学习方法(对比式自监督学习与生成式自监督学习)的性能,以及自监督学习模型在视网膜成像中的可解释性,仍有待深入研究。理解自监督学习模型在训练过程中学习到的特定特征,是其安全、可靠地转化到临床实践中的重要一步。
在本研究中,我们提出了一种新的基于自监督学习的视网膜图像基础模型(RETFound),并系统评估了该模型在适配多种疾病检测任务时的性能与泛化能力。基础模型(foundation model)指通过大规模海量未标注数据训练得到的大型 AI 模型,这类模型可适配各类下游任务 ³¹、³²。本研究中,我们利用自监督学习技术,基于大规模未标注视网膜图像构建了 RETFound 模型,并将其用于促进多种疾病的检测。具体而言,我们采用先进的自监督学习技术(掩码自编码器 ¹⁵),先在自然图像(ImageNet-1k 数据集)上训练,再在 Moorfields 糖尿病图像数据集(MEH-MIDAS)和公开数据集中的视网膜图像上继续训练,最终开发出两个独立的 RETFound 模型,分别适用于彩色眼底摄影(CFP)和光学相干断层扫描(OCT)两种成像方式,训练所用图像总计包括 904,170 张 CFP 图像和 736,442 张 OCT 图像。我们通过特定任务标签对 RETFound 进行微调,使其适配一系列具有挑战性的检测与预测任务,并对其性能进行验证。研究涉及三类任务:一是眼部疾病诊断分类,包括糖尿病视网膜病变和青光眼;二是眼部疾病预后评估,具体为预测 1 年内对侧眼(“健眼”)向新生血管性(“湿性”)年龄相关性黄斑变性(AMD)的转化情况;三是眼组学相关任务,具体为预测 3 年内心血管疾病(缺血性中风、心肌梗死、心力衰竭)和一种神经退行性疾病(帕金森病)的发病风险。与当前最先进的竞争模型(包括通过传统迁移学习在 ImageNet-21k 数据集上预训练的模型)相比,RETFound 在适配这些任务时,始终展现出更优异的性能和标签效率。我们还通过定性结果和变量控制实验,探究了 RETFound 疾病检测性能的可解释性,结果表明,模型识别的图像关键区域与眼部疾病和眼组学领域文献中的已知知识一致。最后,我们将 RETFound 模型公开,以便其他研究者将其作为开展下游任务的基础,从而推动各类眼部疾病和眼组学相关研究的发展。
图 1 概述了 RETFound 模型的构建与应用流程。
在模型构建阶段,我们整理了 904,170 张 CFP 图像和 736,442 张 OCT 图像:其中 90.2% 的 CFP 图像来自 MEH-MIDAS 数据集,9.8% 来自 Kaggle EyePACS 数据集 ³³;85.2% 的 OCT 图像来自 MEH-MIDAS 数据集,14.8% 来自参考文献 34 提及的数据集。MEH-MIDAS 是一个回顾性数据集,包含 2000 年 1 月至 2022 年 3 月期间在 Moorfields 眼科医院就诊的 37,401 名糖尿病患者的完整眼部成像记录。
在这些视网膜图像上完成自监督预训练后,我们评估了 RETFound 在适配各类眼部疾病和眼组学任务时的性能与泛化能力。眼部疾病诊断任务采用公开可用的数据集,详细信息参见补充表 1。
眼部疾病预后评估和全身性疾病预测任务则采用 Moorfields AlzEye 研究(MEH-AlzEye)的队列数据,该数据集将 2008 年至 2018 年期间在 Moorfields 眼科医院就诊的 353,157 名患者的眼科数据,与英格兰全国范围内的医院入院全身性疾病数据相关联 ³⁵。在全身性疾病预测的外部验证中,我们还使用了英国生物样本库(UK Biobank)³⁶的数据。眼部疾病诊断的验证数据集来自多个国家,而由于此类纵向数据可获取性有限,全身性疾病预测仅基于英国的数据集进行验证。因此,我们对全身性疾病预测模型泛化能力的评估虽基于多种任务和数据集,但尚未扩展到地理分布差异极大的场景。临床数据集的详细信息参见补充表 2(数据选择方法详见 “方法” 部分)。
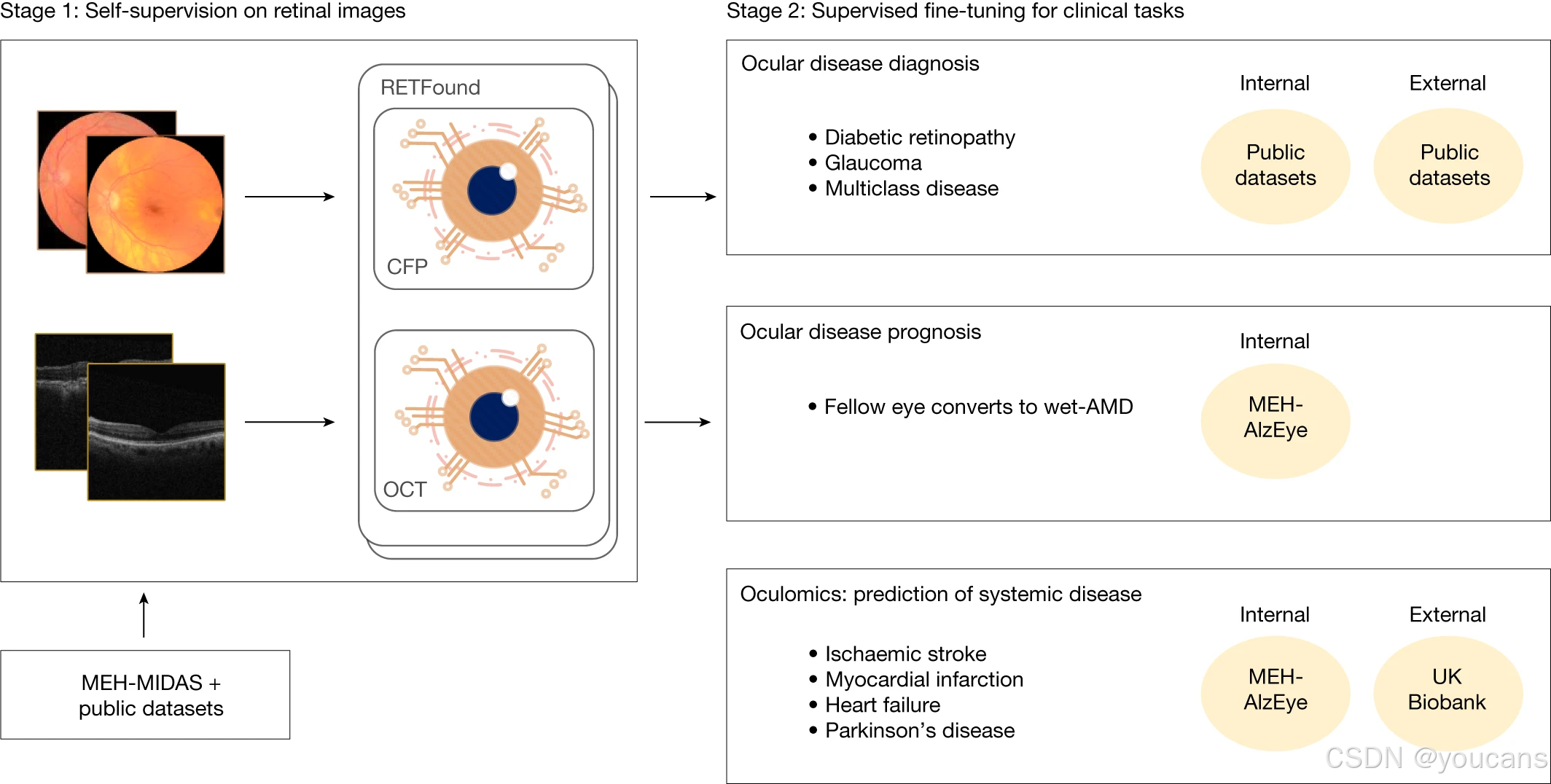
图 1:第一阶段利用MEH-MIDAS的彩色眼底照片(CFP)与光学相干断层扫描(OCT)图像以及公共数据集,通过自监督学习(SSL)构建RETFound;第二阶段通过监督学习将RETFound适配至下游任务,用于内部与外部评估。
我们将 RETFound 的性能和标签效率与三种预训练对比模型进行了比较,这三种模型分别是 SL-ImageNet、SSL-ImageNet 和 SSL-Retinal。所有模型采用的预训练策略不同,但具有相同的模型架构,且针对下游任务的微调流程一致(架构细节详见 “方法” 部分)。其中,SL-ImageNet 采用传统迁移学习方法,即通过在 ImageNet-21k 数据集(约 1400 万张含分类标签的自然图像)上进行监督学习来完成模型预训练;SSL-ImageNet 通过在 ImageNet-1k 数据集(约 140 万张自然图像)上进行自监督学习(SSL)完成预训练;SSL-Retinal 则是在视网膜图像上从零开始通过自监督学习进行预训练。RETFound 以 SSL-ImageNet 的权重为基础,再进一步扩展到视网膜图像上训练(相当于先在自然图像上进行自监督学习预训练,随后在视网膜图像上继续进行自监督学习预训练)。预训练流程示意图详见扩展数据图 1(Extended Data Fig. 1)。
此外,为探究不同自监督学习策略(即生成式自监督学习与对比式自监督学习方法)的性能,我们在 RETFound 框架内,分别用 SimCLR¹⁶、SwAV³⁷、DINO³⁸和 MoCo-v3(参考文献 14)替代了原有的核心自监督学习技术(即掩码自编码器)。我们还报告了这些模型的内部评估与外部评估结果:通过带标签的训练数据使模型适配各项任务后,分别在预留的内部测试集,以及与训练数据完全独立的外部数据集上对模型进行评估(详细信息详见 “方法” 部分)。
模型性能采用受试者工作特征曲线下面积(AUROC)和精确率 - 召回率曲线下面积(AUPR)进行评估。对于每项任务,我们通过双侧 t 检验计算 RETFound 与性能最优的对比模型之间的 P 值,以检验两者性能差异的统计学显著性。
2. 模型性能
2.1 眼部疾病诊断
我们纳入了 8 个公开可用的数据集,以验证模型在多种眼部疾病及不同成像条件下的性能(图 2)。
如图 2a 所示,在大多数数据集上,RETFound 的性能均表现最佳,SL-ImageNet 则位列第二。例如,在糖尿病视网膜病变分类任务中,RETFound 在 Kaggle APTOS-2019、IDRID³⁹和 MESSIDOR-2(参考文献 40、41)数据集上的受试者工作特征曲线下面积(AUROC)分别为 0.943(95% 置信区间(CI):0.941-0.944)、0.822(95% 置信区间:0.815-0.829)和 0.884(95% 置信区间:0.88-0.887),显著优于 SL-ImageNet(所有对比的 P 值均 < 0.001)。在青光眼诊断及多种疾病分类任务中,RETFound 同样展现出更优性能。此外,RETFound 的精确率 - 召回率曲线下面积(AUPR)结果也显著高于对照组(扩展数据图 2a)。
在外部评估中,我们在糖尿病视网膜病变数据集(Kaggle APTOS-2019、IDRID 和 MESSIDOR-2)上测试了 RETFound 的性能,这些数据集的标签均基于国际临床糖尿病视网膜病变严重程度五级分级标准制定。我们在这三个数据集间开展交叉评估,即在一个数据集上对模型进行微调后,在另外两个数据集上进行性能评估。如图 2b 所示,RETFound 在所有交叉评估中均表现最佳。例如,在 Kaggle APTOS-2019 数据集上微调后,RETFound 在 IDRID 数据集上的 AUROC 为 0.822(95% 置信区间:0.815-0.829),在 MESSIDOR-2 数据集上的 AUROC 为 0.738(95% 置信区间:0.729-0.747);其中,在 IDRID 数据集上的性能显著高于 SL-ImageNet(P<0.001),在 MESSIDOR-2 数据集上的性能显著高于 SSL-ImageNet(P<0.001)。所有组别的 AUPR 结果整体偏低,但 RETFound 仍取得了显著更高的性能(扩展数据图 2b)。所有定量结果均列于补充表 3 中。
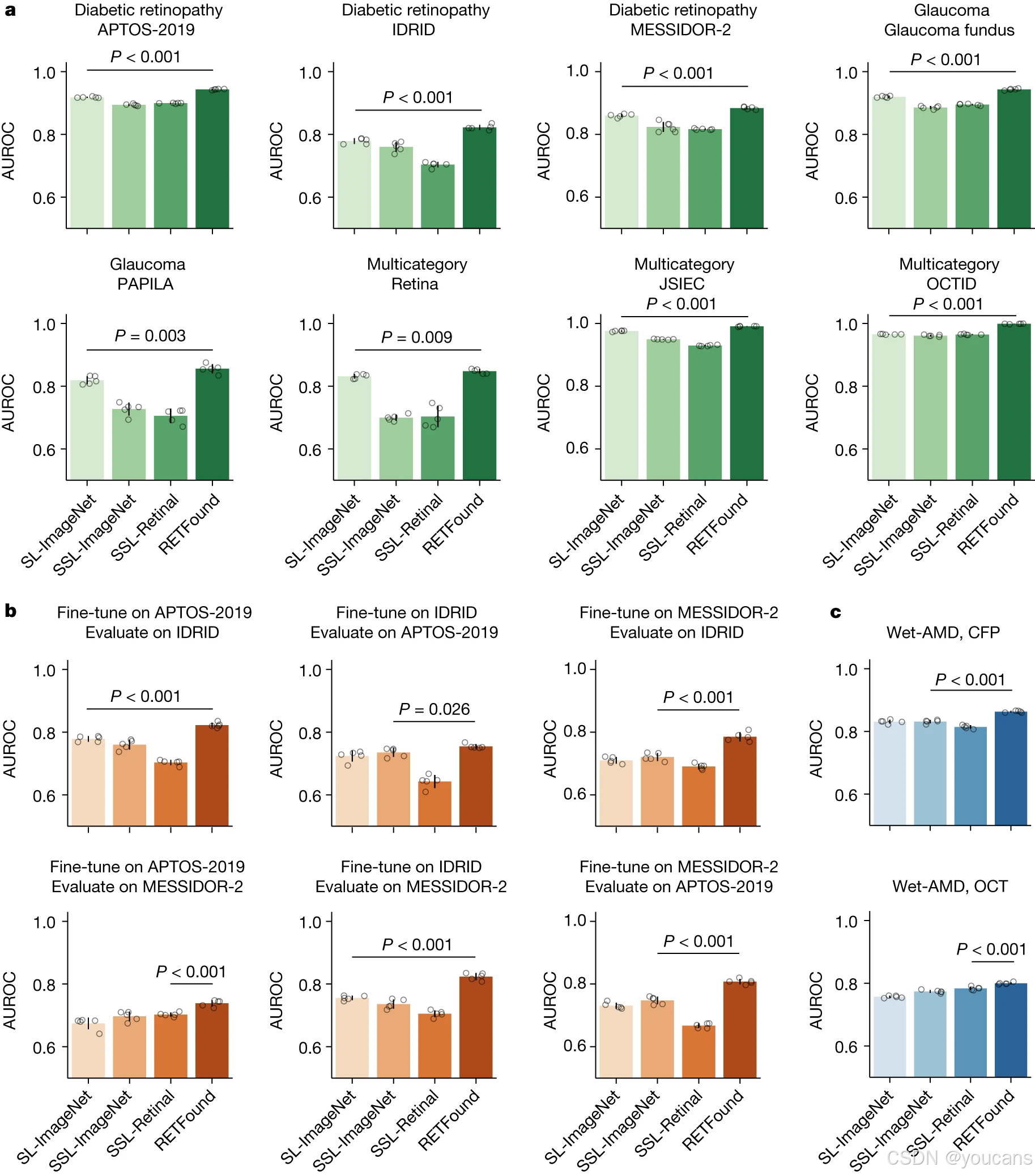
图2:眼科疾病诊断分类性能
a,内部评估。模型通过微调适配至各数据集,并在白内障疾病诊断任务(如糖尿病视网膜病变与青光眼)的留出测试数据上进行内部评估,疾病类别与数据集特征列于补充表1。
b,外部评估。模型在某一糖尿病视网膜病变数据集上微调,并在其余数据集上进行外部评估。
c,眼科疾病预后性能。模型经微调以预测对侧眼在1年内向湿性年龄相关性黄斑变性(wet-AMD)的转化,并进行内部评估。
RETFound在所有任务中表现最佳。对于每项任务,我们使用五个不同的随机种子训练模型,以确定训练数据的洗牌顺序,并在测试集上评估模型以获得五个副本。我们利用这五个副本计算统计量。误差条表示95%置信区间,条形中心代表AUROC的均值。我们将RETFound与最具竞争力的对比模型进行性能比较,以检验是否存在统计学显著差异。P值通过双侧t检验计算并列于图中。
2.2 眼部疾病预后
在对侧眼(“健眼”)1 年内转化为湿性年龄相关性黄斑变性(湿性 AMD)的预后评估中,我们基于 AlzEye 数据集的数据对模型进行了内部性能评估(图 2c)。以彩色眼底摄影(CFP)为输入模态时,RETFound 表现最佳,其受试者工作特征曲线下面积(AUROC)为 0.862(95% 置信区间(CI):0.86-0.865),显著优于所有对比模型组(P<0.001);排名第二的 SL-ImageNet 模型,AUROC 为 0.83(95% 置信区间:0.825-0.836)。以光学相干断层扫描(OCT)为输入模态时,RETFound 的 AUROC 仍为最高,达 0.799(95% 置信区间:0.796-0.802),且在统计学上显著高于 SSL-Retinal 模型(P<0.001)。在 AUPR(精确率 - 召回率曲线下面积)结果方面,RETFound 以 CFP 为输入时表现最佳,以 OCT 为输入时则与 SSL-Retinal 模型性能相当(扩展数据图 2c)。
2.3 全身性疾病预测
我们设计了四项眼组学任务,以评估模型通过视网膜图像预测全身性疾病发病风险的性能(图 3)。尽管这些任务难度较大,所有模型的整体性能均存在一定局限,但如图 3a 所示,在内部评估中,RETFound 无论以 CFP 还是 OCT 为输入模态,均展现出显著的性能提升。例如,以 CFP 为输入预测心肌梗死时,RETFound 的 AUROC 为 0.737(95% 置信区间:0.731-0.743);SSL-Retinal 模型虽排名第二,但性能显著低于 RETFound(P<0.001)。混淆矩阵(扩展数据表 1)显示,RETFound 在该任务中达到了 0.7 的最高敏感性和 0.67 的特异性。
同样,在心力衰竭、缺血性中风和帕金森病的预测任务中,RETFound 均位列第一:其 AUROC 分别为 0.794(95% 置信区间:0.792-0.797)、0.754(95% 置信区间:0.752-0.756)和 0.669(95% 置信区间:0.65-0.688)。即便以 OCT 为输入模态,RETFound 的性能仍显著优于其他模型,且在所有任务中的 AUPR 结果均显著更高(扩展数据图 3a)。
在英国生物样本库(UK Biobank)上进行的外部评估(图 3b)显示,RETFound 与 SSL-Retinal 在缺血性中风预测任务中的性能相近;而在心肌梗死、心力衰竭和帕金森病的预测任务中,RETFound 无论以 CFP 还是 OCT 为输入,均表现最佳。此外,在英国生物样本库的外部评估中,RETFound 在大多数任务中的 AUPR 也显著更高(扩展数据图 3b)。
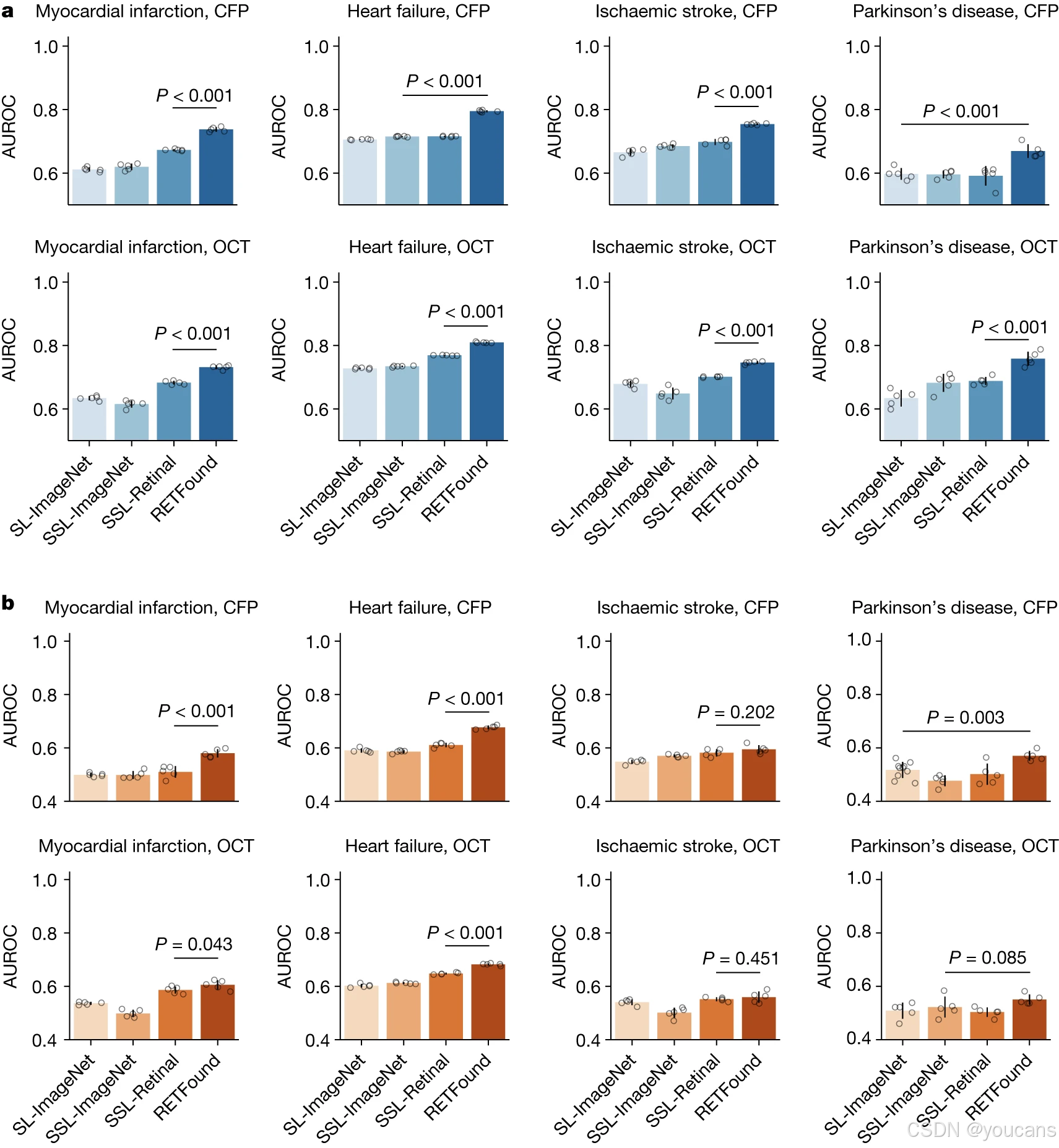
图3:基于视网膜图像的全身性疾病3年发病预测性能
a,内部评估。模型通过微调适配至来自 MEH-AlzEye 的精选数据集,并在留出测试数据上进行内部评估。
b,外部评估。模型在 MEH-AlzEye 上微调,并在 UK Biobank 上进行外部评估。内部与外部评估数据描述于补充表 2。
尽管由于任务难度导致整体性能不高,RETFound 在所有内部评估及大多数外部评估中均获得显著更高的 AUROC。对于每项任务,我们使用五个不同的随机种子训练模型,以确定训练数据的洗牌顺序,并在测试集上评估模型以获得五个副本。我们利用这五个副本计算统计量。误差条表示 95% 置信区间,条形中心代表 AUROC 的均值。我们将 RETFound 与最具竞争力的对比模型进行性能比较,以检验是否存在统计学显著差异。P 值通过双侧 t 检验计算并列于图中。
3. 模型训练与可解释性
3.1 疾病检测的标签效率
标签效率指在特定下游任务中,模型达到目标性能水平所需的训练数据量与标签量,该指标直接反映医学专家所需承担的标注工作量。RETFound 在各类任务中均展现出更优异的标签效率(图 4)。例如,在心力衰竭预测任务中,RETFound 仅使用 10% 的带标签训练数据,性能便超过了采用其他预训练策略的模型,这充分体现了该模型在缓解数据短缺问题上的潜力。
同样,在糖尿病视网膜病变分类和心肌梗死预测任务中,RETFound 的标签效率也显著优于其他模型。此外,RETFound 还始终保持着较高的适配效率(扩展数据图 4),这意味着该模型适配下游任务所需的时间更短。以心肌梗死预测任务为例,RETFound 有望节省约 80% 的模型收敛训练时间;若采用早停(early stopping)等合理机制,还能大幅降低计算成本(例如谷歌云平台的使用额度消耗)。
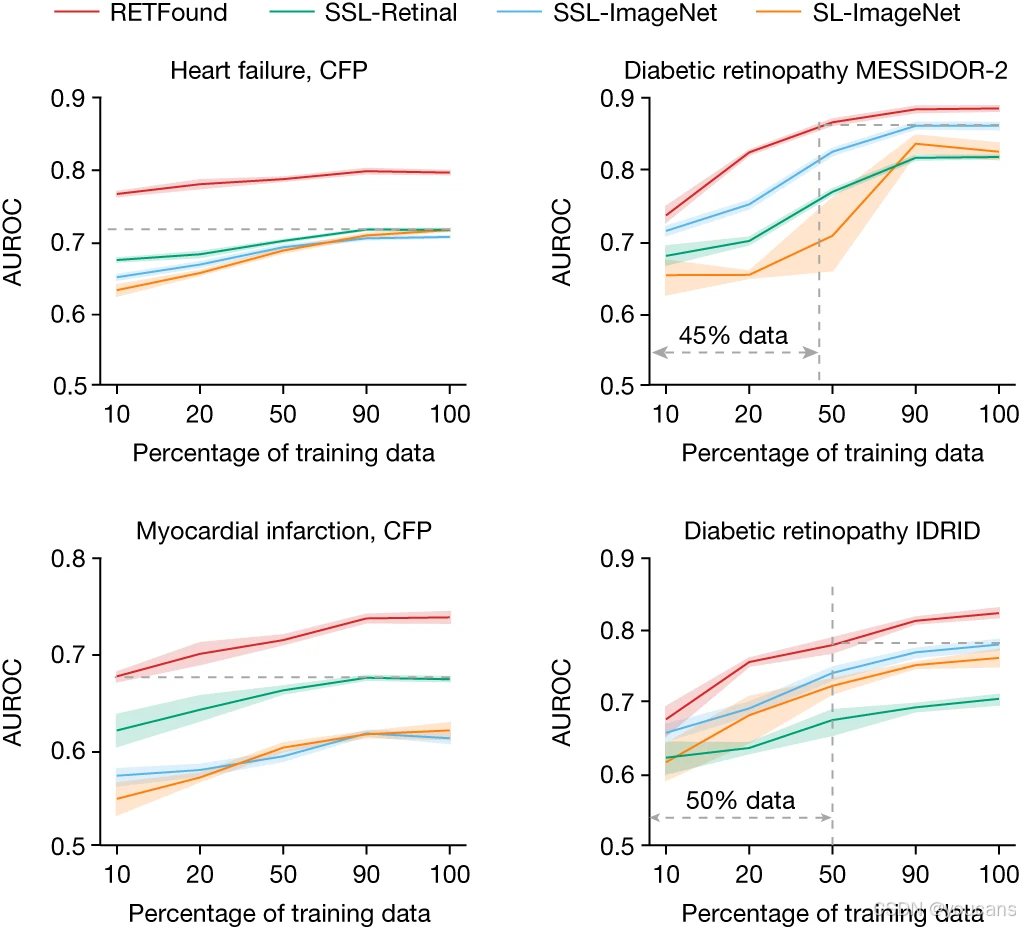
图4:示例应用中的标签效率
标签效率通过不同比例的训练数据衡量性能,以理解达到目标性能水平所需的数据量。灰色虚线突出 RETFound 与最具竞争力的对比模型在训练数据量上的差异。在心力衰竭与心肌梗死的3年发病预测任务中,RETFound 仅使用10%的CFP模态训练数据即优于对照组;在糖尿病视网膜病变 MESSIDOR-2 任务中使用45%数据、在IDRID任务中使用50%数据时,性能与其他组相当。AUROC 的95%置信区间以彩色带状显示,带中心点表示 AUROC 的均值。
3.2 RETFound 的自监督学习(SSL)策略
我们在 RETFound 框架中,探究了不同自监督学习(SSL)策略的性能,具体包括生成式自监督学习(如掩码自编码器)与对比式自监督学习(如 SimCLR、SwAV、DINO 和 MoCo-v3)。如图 5 所示,采用不同对比式自监督学习策略的 RETFound 在下游任务中均展现出良好性能。例如,结合 DINO 策略的 RETFound 在湿性年龄相关性黄斑变性(湿性 AMD)预后评估(扩展数据图 5)和缺血性中风预测(图 5)任务中,受试者工作特征曲线下面积(AUROC)分别为 0.866(95% 置信区间(CI):0.864-0.869)和 0.728(95% 置信区间:0.725-0.731),性能均优于基准模型 SL-ImageNet(补充表 3 和表 4)。这一结果证实,RETFound 框架在适配多种自监督学习策略时均具备有效性。
在所有自监督学习策略中,作为 RETFound 核心策略的掩码自编码器,在大多数疾病检测任务中,性能均显著优于对比式学习方法(图 5 和扩展数据图 5)。所有定量结果均列于补充表 4 中。
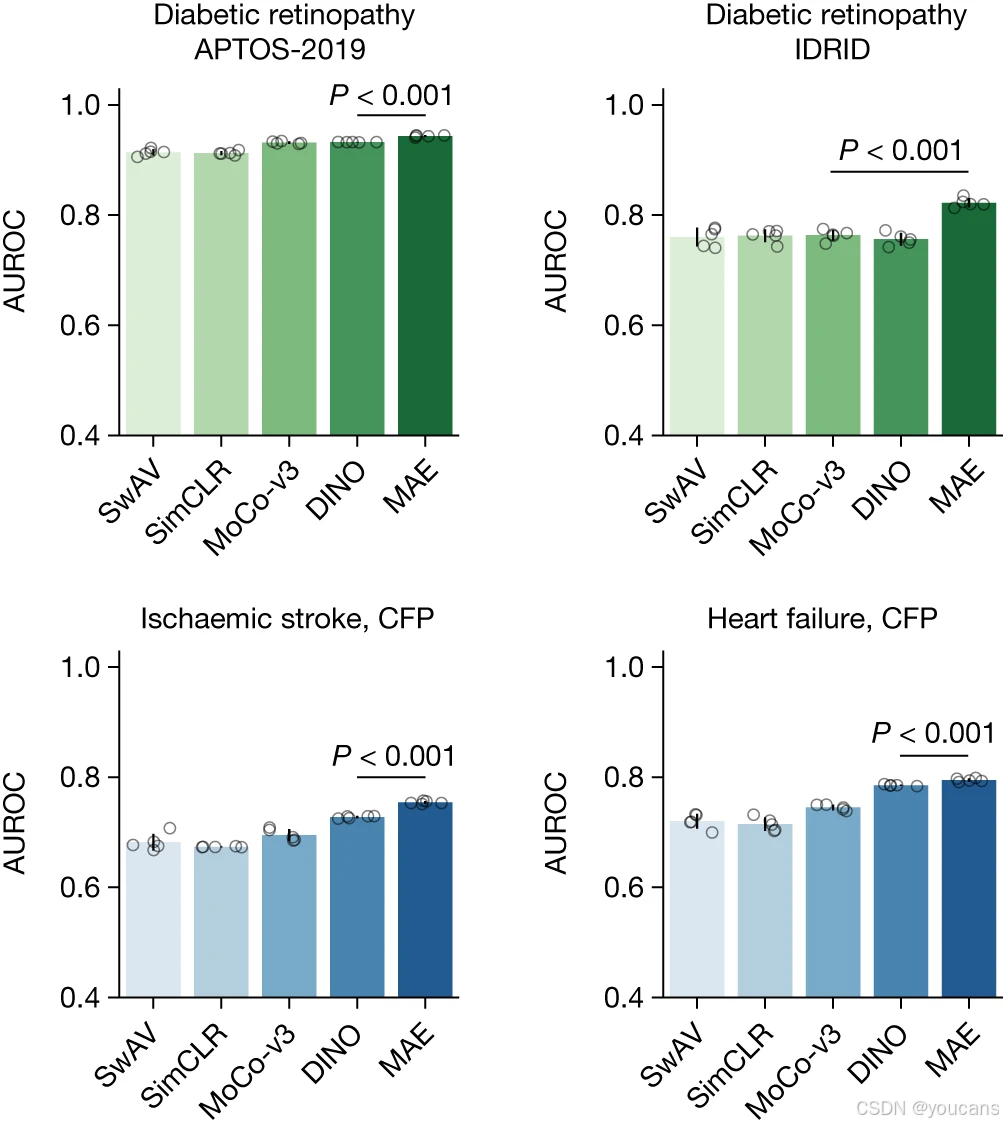
图5:在示例应用中对RETFound框架内不同SSL策略的比较
图5展示了通过不同自监督学习(SSL)策略预训练的模型在糖尿病视网膜病变、缺血性卒中及心力衰竭预测中的AUROC表现,所采用的策略包括掩码自编码器(MAE)、SwAV、SimCLR、MoCo-v3与DINO。系统性疾病任务的数据来源于MEH-AlzEye数据集。采用MAE的RETFound在大多数任务中取得了显著更高的AUROC。对比式SSL方法的相应定量结果列于补充表4。对于每项任务,我们使用五个不同的随机种子训练模型(用于确定训练数据的随机重排),并在测试集上评估模型以获得五个重复样本。我们基于这五个重复样本计算统计量。误差条表示95%置信区间,柱状图中心代表AUPR的均值。我们将RETFound的性能与最具竞争力的对比模型进行比较,以检验是否存在统计学显著差异。P值通过双侧t检验计算,并列于图中。
3.3 模型的可解释性
为深入了解 RETFound 在下游任务中实现优异性能与标签效率的内在机制,我们对其自监督预训练所用的预训练任务(pretext task)及任务特异性决策过程进行了定性分析(扩展数据图 6)。RETFound 的预训练任务能够让模型学习视网膜特异性背景信息,包括解剖结构与疾病病变。如扩展数据图 6a 所示,即便视网膜图像 75% 的区域被掩码,RETFound 仍能重建主要解剖结构 —— 在彩色眼底摄影(CFP)图像中可重建视神经和大血管,在光学相干断层扫描(OCT)图像中可重建神经纤维层和视网膜色素上皮。这一结果表明,RETFound 已通过自监督学习(SSL)掌握了识别并推断疾病相关区域特征表示的能力,而这正是其在下游任务中具备出色性能与标签效率的关键原因。
在基于图像重建的可解释性分析基础上,我们进一步采用先进的解释工具(RELPROP⁴²),对微调后模型在下游任务中实现分类决策所依赖的图像关键区域进行了可视化(扩展数据图 6b)。在眼部疾病诊断任务中,模型能识别出明确的病变特征并用于分类,例如将硬性渗出物和出血作为糖尿病视网膜病变的判断依据,将视盘周围萎缩作为青光眼的判断依据。在眼组学任务中,我们观察到与全身性疾病相关的解剖结构被标记为关键贡献区域:在 CFP 图像中是视神经,在 OCT 图像中是神经纤维层和神经节细胞层,这些区域对全身性疾病的发病预测具有重要作用(扩展数据图 6b)。
3.4 对年龄分布差异的稳健性
对于与衰老相关的全身性疾病,其临床相关解剖结构会随年龄增长⁴³、⁴⁴及疾病进展 ¹⁹、²⁰、²² 而发生变化。RETFound 在训练过程中,已学习识别用于检测全身性疾病的一般性结构变化(扩展数据图 6b)。为进一步验证模型对衰老相关解剖结构变化与疾病进展相关解剖结构变化的区分学习能力,我们在心肌梗死预测任务中,设置了四个年龄不同的对照组(平均年龄分别为 66.8 岁、68.5 岁、70.4 岁和 71.9 岁),并与一个年龄固定的疾病组(平均年龄 72.1 岁)进行对比,以此评估模型性能。
如扩展数据图 7 所示,年龄差异越大,模型性能越好,这表明年龄确实是研究衰老相关疾病时的混杂因素。年龄的影响可通过极端案例体现:当队列间年龄差异最大时(本研究中为 5.3 岁),仅以年龄为输入的简单逻辑回归模型,其受试者工作特征曲线下面积(AUROC)便达到 0.63,性能超过 SSL-ImageNet 和 SL-ImageNet。而当年龄差异缩小时,所有模型的性能均明显优于逻辑回归模型。值得注意的是,即便年龄差异缩小,RETFound 仍能保持稳定性能,这表明该模型能够有效识别与疾病相关的解剖结构变化,并利用这些信息进行全身性疾病预测。
4. 讨论
本研究提出了一种全新的基于自监督学习(SSL)的基础模型 RETFound,并评估了其在适配各类下游任务时的泛化能力。RETFound 采用先进的自监督学习技术(掩码自编码器),在大规模无标签视网膜图像上完成训练后,可高效适配各类疾病检测任务,显著提升眼部疾病检测性能,以及心血管疾病和神经退行性疾病的预测准确性。作为一款经过开发与评估的医学基础模型,RETFound 在利用多维度数据时无需依赖海量高质量标签,展现出巨大应用潜力。
RETFound 通过学习识别疾病相关病变,提升了眼部疾病的检测性能。眼部疾病的诊断依赖明确的病理特征,例如糖尿病视网膜病变的硬性渗出物与出血症状。这些特征在颜色或结构上存在异常变化,与周围视网膜组织存在明显视觉差异。RETFound 能够识别此类疾病相关特征,从而实现眼部疾病的准确诊断(例如扩展数据图 6b 中所示的近视与糖尿病视网膜病变案例)。如图 2 所示,RETFound 在各类任务中均排名第一,SL-ImageNet 紧随其后。SL-ImageNet 通过在 ImageNet-21k 数据集上进行监督学习完成预训练,该数据集包含 1400 万张图像,涵盖 21000 类自然物体(如斑马、橙子等),具有丰富的形状与纹理多样性。这种多样性使模型能够学习大量低级特征(如线条、曲线、边缘),进而识别异常特征的边界,因此在适配医学任务时可提升疾病诊断性能。本研究证实,通过在自然图像和无标签视网膜图像上依次进行自监督学习,能够开发出具备泛化能力的基础模型(RETFound),该模型不仅可进一步改善眼部疾病的诊断与预后评估效果,甚至能超越性能强劲的 SL-ImageNet。
RETFound 通过在无标签视网膜数据上进行自监督学习,掌握了视网膜特异性背景信息,从而提升了全身性健康状态的预测能力。在利用无标签视网膜图像进行自监督学习以预测全身性疾病的任务中,RETFound 与 SSL-Retinal 在内部和外部评估中均位列前两名(图 3)。在预训练阶段,RETFound 通过完成 “从高度掩码图像中重建完整图像” 这一预训练任务来学习特征表示,该任务要求模型仅依靠有限的可见图像块推断被掩码区域的信息。在视网膜图像上完成此类预训练任务后,模型能够学习到视网膜特异性背景信息,包括视神经、视网膜神经纤维层等解剖结构(扩展数据图 6a)—— 这些结构正是视网膜图像中神经退行性疾病与心血管疾病的潜在标志物 ¹⁷、¹⁹、²¹、⁴⁵。混淆矩阵显示,RETFound 的敏感性最高(扩展数据表 1),这意味着该模型能识别出更多全身性疾病高风险人群。眼组学任务评估结果表明,RETFound 的应用显著推动了视网膜图像在全身性疾病发病预测与风险分层中的应用。
与 SSL-Retinal 和 SSL-ImageNet 相比,RETFound 在疾病检测任务中始终表现更优(图 2、图 3 及补充表 3),这表明在视网膜图像和自然图像上分别进行自监督学习,对于开发高性能基础模型具有互补作用。在模型开发中结合自然图像与医学数据的策略,已在胸部 X 光成像⁶、皮肤科成像⁴⁶等其他医学领域得到验证。我们还对眼组学任务中的预测模型进行了校准分析,以检验预测概率与实际发病率的一致性。校准效果良好的模型能够提供有意义且可靠的疾病预测结果(预测概率可反映疾病实际发生的可能性),进而实现疾病风险分层⁴⁷、⁴⁸。结果显示,与其他模型相比,RETFound 的校准效果更优,在可靠性图中展现出最低的期望校准误差(扩展数据图 8)。这一结果证实,RETFound 生成的预测概率具有可靠性,不会出现过度自信的预测情况。
实验结果表明,彩色眼底摄影(CFP)和光学相干断层扫描(OCT)两种成像模态均包含独特的眼部及全身性信息,对预测未来健康状态具有重要价值。在眼部疾病诊断中,特定成像模态因能清晰呈现病变特征而被广泛应用,例如 OCT 常用于湿性年龄相关性黄斑变性(湿性 AMD)的诊断。然而,在眼组学任务中,此类应用认知相对模糊,原因包括:(1)不同成像模态下眼组学研究的标志物仍在探索阶段;(2)需在统一评估条件下对多种成像模态进行公平比较。本研究在完全一致的训练与评估条件下(例如根据匿名患者 ID 划分训练集、验证集和 / 或测试集),探究并比较了 CFP 与 OCT 在眼组学任务中的效能。结果发现,以 CFP 和 OCT 为输入的模型在全身性疾病预测任务中性能存在差异(图 3 及补充表 3),这表明两种模态为眼组学任务提供的信息丰富度不同。例如,在 3 年缺血性中风发病预测任务中,无论在 MEH-AlzEye 内部评估还是英国生物样本库(UK Biobank)外部评估中,以 CFP 为输入的 RETFound 性能均优于以 OCT 为输入的版本;而在帕金森病预测任务的内部评估中,以 OCT 为输入的 RETFound 表现更优。这些发现可能表明,不同衰老相关疾病(如中风、帕金森病)在视网膜图像上呈现的早期标志物存在差异。这一结论对医疗服务提供者和成像设备制造商具有实际指导意义:应认识到 CFP 仍具有重要价值,需将其保留为眼科健康检查中的标准视网膜评估项目之一。同时,该发现也为眼组学研究提供了方向 —— 需进一步探究全身性健康状况与多种成像模态所包含信息之间的关联强度。
当适配后的模型在人口统计学特征不同的新队列(甚至使用不同成像设备获取的数据)上进行测试时(外部评估阶段),性能会出现显著下降。这种现象在眼部疾病诊断(图 2b)和全身性疾病预测(图 3b)的外部评估中均有体现。例如,在缺血性中风预测任务中,模型性能明显下降(RETFound 以 CFP 为输入时,受试者工作特征曲线下面积(AUROC)下降 0.16;以 OCT 为输入时,AUROC 下降 0.19)。在具有挑战性的眼组学任务中,内部验证队列(MEH-AlzEye)与外部验证队列(英国生物样本库)在年龄、种族构成及所用成像设备方面均存在显著差异(补充表 2),这种差异可能是导致模型在英国生物样本库队列上进行外部评估时性能下降的原因。但与其他模型相比,RETFound 在大多数外部评估任务中(图 3b)及不同种族亚组中(扩展数据图 9-11)均表现出显著更优的性能,展现出良好的泛化能力。
我们观察到,即便在 RETFound 框架中替换为各类对比式自监督学习方法,模型在疾病检测任务中仍能保持具有竞争力的性能(图 5 及扩展数据图 5)。总体而言,采用掩码自编码器的生成式方法,性能优于 SwAV、SimCLR、MoCo-v3、DINO 等对比式方法。但需注意的是,由于所有模型存在多项变量差异(例如网络架构:SwAV 和 SimCLR 采用 ResNet-50,其他方法采用 Transformer;超参数:如学习率调度器),因此断言掩码自编码器的优越性时需谨慎。我们的对比实验证实,将强大的网络架构与复杂的预训练任务相结合,能够开发出高效且通用的医学基础模型,这与从医疗领域大型语言模型中得出的见解一致⁴⁹、⁵⁰。此外,该对比实验进一步支持了以下观点:通过掩码自编码器预训练任务学习到的视网膜特异性背景信息(包括视神经乳头、视网膜神经纤维层等解剖结构,如扩展数据图 6a 所示),确实为眼部疾病和全身性疾病的检测提供了具有区分度的信息。
我们认为,以 RETFound 为代表的医学基础模型研究,有望推动医疗人工智能的普及应用,并加速其向广泛临床实践转化的进程。要实现这一目标,基础模型必须从海量医学数据(本研究中为 160 万张视网膜图像)中学习强大的特征表示,而这类海量数据通常仅能被拥有高效数据集整理流程的大型机构获取。此外,基础模型的自监督学习预训练需大量计算资源才能实现训练收敛 —— 本研究使用谷歌云平台上的 8 张 NVIDIA Tesla A100(40GB)图形处理器(GPU),耗时 2 周完成预训练。相比之下,将 RETFound 微调到下游任务所需的数据量与计算资源显著更少,因此大多数机构均有能力实现:仅需 1 张 NVIDIA Tesla T4(16GB)GPU,处理 1000 张图像的微调任务约需 1.2 小时。此外,基础模型还有望提升医疗人工智能模型的整体质量,其应用可避免开发那些 “表面性能亮眼但对临床诊疗影响甚微” 的模型 —— 这类泛化能力差的模型不仅消耗大量资源,还可能加剧人们对医疗人工智能应用价值的质疑。通过公开 RETFound,我们希望为研究者提供支持:利用我们的大型数据集设计适用于自身机构的模型,或探索更多下游应用场景,从而推动人工智能在医学领域的发展进程。
尽管本研究已系统评估了 RETFound 在多种疾病检测与预测任务中的性能,但仍存在一些局限性与挑战,需在未来研究中进一步探索。首先,开发 RETFound 所用的数据大多来自英国队列,因此有必要纳入全球范围内的视网膜图像,构建规模更大、分布更均衡的多样化数据集,以探究其对模型性能的影响。其次,尽管本研究探究了 CFP 与 OCT 两种模态的模型性能,但尚未研究两种模态的信息融合 —— 这一方向或可进一步提升模型性能。最后,目前的自监督学习模型未纳入人口统计学特征、视力等临床相关信息,而这些信息可能是眼部疾病与眼组学研究中的重要协变量。基于以上分析,我们计划在后续研究中通过以下方式进一步增强 RETFound 的性能:纳入更大规模的图像数据、探索更多成像模态、实现多模态数据间的动态交互。尽管我们对 RETFound 在各类人工智能任务中的广泛应用前景持乐观态度,但也认识到:加强人机协同是实现医疗人工智能应用真正多样化的关键所在。
综上,本研究证实了 RETFound 在适配各类医疗应用场景中的有效性与高效性 —— 该模型在眼部疾病检测任务中表现出优异的性能与泛化能力,在全身性疾病预测任务中也实现了显著性能提升。基于自监督学习的基础模型突破了当前临床人工智能应用面临的关键障碍(尤其是对标注数据的依赖、有限的性能与泛化能力),为开发高效数据利用、加速临床转化的设备打开了大门,有望为眼部疾病及全身性疾病患者的诊疗带来变革。
5. 方法
5.1 用于开发 RETFound 的数据集
我们整理了大规模无标注视网膜图像集用于自监督学习(SSL),总计包含 904,170 张彩色眼底摄影(CFP)图像和 736,442 张光学相干断层扫描(OCT)图像。其中,815,468 张(90.2%)CFP 图像和 627,133 张(85.2%)OCT 图像来自 Moorfields 糖尿病图像数据集(MEH-MIDAS);88,702 张(9.8%)CFP 图像来自 Kaggle EyePACS 数据集,109,309 张(14.8%)OCT 图像来自参考文献 34 提及的数据集。
MEH-MIDAS 是一个回顾性数据集,包含 2000 年至 2022 年期间在英国伦敦 Moorfields 眼科医院就诊的 37,401 名糖尿病患者的完整眼部成像记录,其中女性 16,429 人、男性 20,966 人、性别未知 6 人。该数据集患者年龄分布的平均值为 64.5 岁,标准差为 13.3 岁;种族构成具有多样性,具体为英国裔(13.7%)、印度裔(14.9%)、加勒比裔(5.2%)、非洲裔(3.9%)、其他种族(37.9%)及未申报种族(24.4%)。MEH-MIDAS 包含多种成像设备拍摄的图像,如拓普康 3DOCT-2000SA(Topcon)、蔡司 CLARUS(ZEISS)和拓普康 Triton(Topcon)。EyePACS 数据集的成像设备包括 Centervue DRS(Centervue)、Optovue iCam(Optovue)、佳能 CR1/DGi/CR2(Canon)和拓普康 NW(Topcon);参考文献 34 的数据集则包含海德堡 SPECTRALIS 设备拍摄的图像。
5.2 用于眼部疾病诊断的数据
我们在三类不同的疾病检测任务中评估模型性能。第一类任务为眼部疾病诊断分类,采用公开可用的眼科数据集。其中,糖尿病视网膜病变诊断使用 Kaggle APTOS-2019(印度)、IDRID(印度)和 MESSIDOR-2(法国)数据集,其标签基于国际临床糖尿病视网膜病变严重程度五级分级标准制定,涵盖从无糖尿病视网膜病变到增殖性糖尿病视网膜病变的五个阶段。
青光眼诊断任务纳入了 PAPILA⁵¹(西班牙)和 Glaucoma Fundus⁵²(韩国)数据集,这两个数据集均包含三类标签:非青光眼、早期青光眼(疑似青光眼)和晚期青光眼。
多疾病分类任务则使用 JSIEC⁵³(中国)、Retina 和 OCTID⁵⁴(印度)数据集。其中,JSIEC 包含 1000 张图像,涵盖 39 类常见可转诊眼底疾病及病症;Retina 数据集的标签分为正常、青光眼、白内障和视网膜疾病四类;OCTID 包含 470 张 OCT 图像,标签分为正常、黄斑裂孔、年龄相关性黄斑变性(AMD)、中心性浆液性视网膜病变和糖尿病视网膜病变五类。
公开数据集的分级流程总结如下:IDRID 数据集由两名医学专家共同判定,达成共识后确定分级结果;MESSIDOR-2 数据集由三名视网膜专科医生组成的专家组依据已发表的流程⁵⁵进行判定;APTOS-2019 为 Kaggle 数据集,相关信息有限,推测可能由单一临床医生完成标注;PAPILA 数据集由两名专家在全面临床检查、测试(包括回顾性临床记录审查)后完成标注与分割;Glaucoma Fundus 数据集的标签由两名专家根据视野检查结果和大量成像数据共同确定;JSIEC 数据集由眼科医生标注,并经资深视网膜专家确认。对于分歧解决方式:Retina 数据集未提供详细信息;OCTID 数据集标注基于视网膜临床专家的诊断,但未明确说明是否通过重复判定解决分歧。各数据集的成像设备、来源国家、标签类别等详细信息详见补充表 1。
5.3 用于疾病预后与预测的数据
在对侧眼(“健眼”)1 年内转化为湿性年龄相关性黄斑变性(湿性 AMD)的预后任务中,我们使用了 Moorfields AlzEye 研究(MEH-AlzEye)的数据集。MEH-AlzEye 是一项回顾性队列研究,将 2008 年至 2018 年期间在 Moorfields 眼科医院就诊的 353,157 名患者的眼科数据,与英格兰全国范围内的医院入院全身性疾病数据相关联。其中,全身性疾病数据来源于医院事件统计(HES)中的住院患者护理数据,重点关注心血管疾病和全因痴呆;HES 住院患者护理数据中的诊断编码依据《国际疾病分类》(ICD)第十次修订版⁵⁶记录。
参照以往研究报告,我们通过 ICD 编码筛选研究队列,涉及疾病包括中风(I23-I24)、心肌梗死(I21-I22)、心力衰竭(I50)和帕金森病(G20)。在 186,651 名有 HES 数据的患者中,6,504 名患者至少一只眼被诊断为湿性 AMD;排除其他眼部疾病后,819 名患者在对侧眼转化为湿性 AMD 前 1 年内有视网膜成像记录,747 名患者的对侧眼未转化为湿性 AMD。
最后一类任务为全身性疾病 3 年预测,重点关注心血管疾病和神经退行性疾病,采用 MEH-AlzEye 和英国生物样本库(UK Biobank)的数据集。英国生物样本库包含 502,665 名年龄在 40-69 岁之间、在英国国家医疗服务体系(NHS)注册的英国居民数据;在所有参与者中,82,885 人接受了 CFP 和 OCT 检查,共收集 171,500 张视网膜图像。为避免因个体就诊次数不一致可能产生的偏差,每位患者仅纳入一次就诊中的左眼视网膜图像。
内部评估时,患者群体按 55:15:30 的比例划分为训练集、验证集和测试集:训练集用于调整模型参数以实现目标函数;验证集用于监测训练收敛情况并选择模型检查点;测试集用于测试保存的模型检查点,评估内部性能。外部验证时,所有患者数据均用于评估保存的模型检查点。详细的数据流程图详见补充图 1-5。
5.4 自监督学习(SSL)的数据处理与增强
在彩色眼底摄影(CFP)图像预处理中,我们使用自动化视网膜图像分析工具 AutoMorph⁵⁷剔除背景区域,保留视网膜有效区域。所有图像均通过三次插值法调整为 256×256 像素大小。对于光学相干断层扫描(OCT)图像,我们提取其中间切片,并将其调整为 256×256 像素大小。
模型训练过程中,我们采用与掩码自编码器一致的数据增强策略,具体包括:随机裁剪(裁剪范围下限为图像整体的 20%、上限为 100%),并将裁剪后的图像块调整为 224×224 像素;随机水平翻转;以及图像归一化处理。
RETFound 的架构与实现:我们采用掩码自编码器 ¹⁵的特定配置构建 RETFound,该架构由编码器(encoder)和解码器(decoder)两部分组成,详细架构见图补充图 6(Supplementary Fig. 6)。
- 编码器:采用大型视觉 Transformer(ViT-large),包含 24 个 Transformer 块,嵌入向量维度为 1024。编码器以未掩码的图像块(块大小为 16×16 像素)为输入,将其投影为维度为 1024 的特征向量;24 个 Transformer 块(由多头自注意力机制和多层感知机构成)接收该特征向量后,生成高级特征。
- 解码器:采用小型视觉 Transformer(ViT-small),包含 8 个 Transformer 块,嵌入向量维度为 512。解码器将掩码虚拟块插入编码器提取的高级特征中作为模型输入,经线性投影后重建图像块。
模型训练的目标是从高度掩码的图像中重建完整视网膜图像,其中 CFP 图像的掩码率为 0.75,OCT 图像的掩码率为 0.85。训练批次大小(batch size)设为 1792(8 张 GPU,每张 GPU 处理 224 个样本);总训练轮次(epoch)为 800,前 15 轮用于学习率预热(从 0 逐步提升至 1×10⁻³)。训练结束后,保存最后一轮的模型权重作为适配下游任务的检查点(checkpoint)。
5.5 下游任务适配
在适配下游任务时,仅需保留基础模型的编码器(ViT-large),舍弃解码器。编码器从视网膜图像中生成高级特征,多层感知机(multilayer perceptron)以这些特征为输入,输出各类疾病的预测概率,概率最高的类别即为最终分类结果。疾病类别的数量决定了多层感知机最后一层的神经元数量。
为防止模型过拟合,我们引入标签平滑(label smoothing)技术:通过软化训练数据中的真实标签(ground-truth labels)来规范输出分布。下游任务的训练目标是使模型输出与标签一致的分类结果,具体训练参数如下:批次大小为 16;总训练轮次为 50,前 10 轮用于学习率预热(从 0 逐步提升至 5×10⁻⁴),剩余 40 轮采用余弦退火调度(学习率从 5×10⁻⁴逐步降至 1×10⁻⁶)。
每轮训练结束后,均在验证集上评估模型性能,保存验证集上受试者工作特征曲线下面积(AUROC)最高的模型权重,作为内部评估与外部评估的模型检查点。
5.6 对比式自监督学习(SSL)的实现
在 RETFound 框架中,我们将核心自监督学习方法(即掩码自编码器)替换为 SimCLR¹⁶、SwAV³⁷、DINO³⁸和 MoCo-v3(参考文献 14),生成多种预训练模型变体以进行性能对比。对于每种对比式学习方法的自监督训练,为确保最优性能,我们均遵循已发表文献中推荐的网络架构与超参数设置。首先,我们将 ImageNet-1k 数据集上的预训练权重加载到模型中,随后使用每种对比式学习方法,在 160 万张视网膜图像上进一步训练模型,得到最终的预训练模型。之后,我们采用与掩码自编码器完全一致的迁移流程,对这些预训练模型进行微调,以适配下游疾病检测任务。
5.7 微调后模型的可解释性分析
我们采用专为 Transformer 类网络设计的 RELPROP⁴² 方法进行模型解释。该方法通过逐层相关性传播,计算每一层中每个注意力头的相关性得分,随后结合相关性信息与梯度信息,在整个注意力图中整合这些得分。最终,该方法能可视化输入图像中对特定分类结果起关键作用的区域。已有研究表明,RELPROP 的性能优于 GradCam⁵⁹等其他知名解释技术。
5.8 计算资源
自监督学习的训练过程通常受益于较大的批次大小(batch size),这有助于从数据中提取上下文信息,但同时也需要高性能图形处理器(GPU)支持计算。本研究中,我们在谷歌云平台(Google Cloud Platform)上使用 8 张 NVIDIA Tesla A100(40GB)GPU 开发 RETFound,整个开发过程耗时约 14 天。对于每种自监督学习方法的预训练,我们均分配了同等的计算资源。
在将 RETFound 微调到下游任务时,我们使用 NVIDIA Tesla T4(16GB)GPU,每处理 1000 张图像的微调任务约需 70 分钟。
5.9 评估与统计分析
所有任务的性能均采用分类指标受试者工作特征曲线下面积(AUROC)和精确率 - 召回率曲线下面积(AUPR)进行评估,这两个指标分别通过分类器的受试者工作特征(ROC)曲线和精确率 - 召回率(PR)曲线计算得出。
对于眼部疾病预后评估和眼组学预测任务,AUROC 和 AUPR 均在二分类场景下计算。
对于多分类任务(如糖尿病视网膜病变五级分级、多类别疾病诊断),我们先计算每个疾病类别的 AUROC 和 AUPR,再取平均值作为整体的 AUROC 和 AUPR。
为确保结果可靠性,每个任务均使用 5 个不同的随机种子训练模型(随机种子决定训练数据的打乱方式)。我们计算 5 次迭代训练的性能均值与标准差,并通过 “标准差 /√5” 计算标准误(standard error);95% 置信区间(CI)则通过 “1.96× 标准误” 计算得出。此外,我们通过双侧 t 检验比较 RETFound 与性能最优的对比模型之间的性能差异,以验证差异是否具有统计学显著性。
5.10 伦理声明
本研究涉及人类受试者,已获得以下机构批准:伦敦中央研究伦理委员会(批件号:18/LO/1163,批准日期:2018 年 8 月 1 日)、遗传性与获得性视网膜疾病多模态数据高级统计建模项目伦理委员会(批件号:20/HRA/2158,批准日期:2020 年 5 月 5 日),以及第 251 条保密咨询小组(批件号:18/CAG/0111,批准日期:2018 年 9 月 13 日)。英国国家医疗服务体系(NHS)健康研究管理局于 2018 年 9 月 13 日给予最终批准。Moorfields 眼科医院 NHS 基金会信托基金对数据的去标识化处理进行了验证。本研究仅使用去标识化的回顾性数据,未涉及患者主动参与。
6. 其它
报告摘要
有关本研究设计的更多信息,可查阅与本文相关联的《自然》系列期刊报告摘要(Nature Portfolio Reporting Summary)。
在线内容
所有方法、补充参考文献、《自然》系列期刊报告摘要、源数据、扩展数据、补充信息、致谢、同行评审信息,以及作者贡献详情、利益冲突说明和数据与代码可用性声明,均可通过以下链接获取:https://doi.org/10.1038/s41586-023-06555-x。
数据可用性
MIDAS 数据集包含常规收集的医疗健康数据。由于该数据集具有敏感性且存在身份再识别风险,其访问需通过结构化申请流程进行管控。如需咨询数据访问事宜,可发送邮件至 enquiries@insight.hdrhub.org,我们将在 2 周内予以回复。有关数据申请流程的更多详情,可访问 INSIGHT 健康数据研究中心网站(https://www.insight.hdrhub.org)查询。
AlzEye 数据集受英国国家医疗服务体系数字部门(National Health Service Digital)、Moorfields 眼科医院与伦敦大学学院之间数据共享协议的合同限制,仅 AlzEye 研究团队可访问。我们欢迎国内外研究机构开展合作,但受队列数据访问权限限制,仅 AlzEye 研究人员可直接分析个体层面的全身性健康数据。更多详情可访问以下链接:https://readingcentre.org/studies/artificial_intelligence/alzeye。
英国生物样本库(UK Biobank)数据可通过其官方网站(https://www.ukbiobank.ac.uk/)获取。眼部疾病实验所用数据均已在线公开,可通过以下链接访问:
- IDRID 数据集:https://ieee-dataport.org/open-access/indian-diabetic-retinopathy-image-dataset-idrid
- MESSIDOR-2 数据集:https://www.adcis.net/en/third-party/messidor2/
- APTOS-2019 数据集:https://www.kaggle.com/competitions/aptos2019-blindness-detection/data
- PAPILA 数据集:https://figshare.com/articles/dataset/PAPILA/14798004/1
- Glaucoma Fundus 数据集:https://dataverse.harvard.edu/dataset.xhtml?persistentId=doi:10.7910/DVN/1YRRAC
- JSIEC 数据集:https://zenodo.org/record/3477553
- Retina 数据集:https://www.kaggle.com/datasets/jr2ngb/cataractdataset
- OCTID 数据集:https://borealisdata.ca/dataverse/OCTID
代码可用性
由 Y.Z.(周宇坤)开发的 RETFound 训练、微调及评估代码基于 PyTorch 框架,可通过以下链接获取:https://github.com/rmaphoh/RETFound_MAE。此外,由 Y.K.(木原由香)实现的 Keras 版本代码可访问:https://github.com/uw-biomedical-ml/RETFound_MAE。请注意,本文报告的实验结果均来自 PyTorch 版本模型。
图像处理使用自动化视网膜图像分析工具 AutoMorph v.1.0(https://github.com/rmaphoh/AutoMorph);图像数据从 Dicom 文件中提取时使用 Pydicom v.2.3.0。
7. Github 项目使用
7.1 RETFound 项目介绍
项目地址: Github
本官方代码仓库包含一系列针对视网膜图像的基础模型及应用。
- [RETFound-MAE]:RETFound—— 一款可泛化检测视网膜图像疾病的基础模型。
- [RETFound-DINOv2]:《揭示预训练数据对医学基础模型的影响》(相关模型)。
- [DINOv2]:Meta 公司推出的通用视觉基础模型 DINOv2。
- [DINOv3]:Meta 公司推出的通用视觉基础模型 DINOv3。
📝核心特点
- RETFound 通过自监督学习,在 160 万张视网膜图像上完成预训练
- RETFound 已在多项疾病检测任务中完成有效性验证
- RETFound 可高效适配定制化任务
🎉最新动态
- 🐉2025 年 9 月:对比 DINOv3、DINOv2 与 RETFound 性能的预印本报告已发布!
- 🐉2025 年 9 月:已将当前最先进的 DINOv3 纳入视网膜应用的微调流程!
- 🐉2025 年 2 月:已在 HuggingFace 平台整理模型权重,无需再手动下载!
- 🐉2025 年 2 月:新增多种预训练权重,包括基于 MAE(掩码自编码器)和基于 DINOv2 的权重!
- 🐉2025 年 2 月:已更新软件包版本,支持 CUDA12+、PyTorch 2.3 + 等!
- 🐉2024 年 1 月:特征向量相关笔记本(notebook)已上线!
- 🐉2024 年 1 月:公开数据集的数据划分方式及模型检查点已上线!
- 🎄2023 年 12 月:Colab 笔记本已上线 —— 提供免费 GPU 资源,操作简便!
7.2 RETFound 快速入门
7.2.1 安装环境
- 创建虚拟环境:
conda create -n retfound python=3.11.0 -y
conda activate retfound
- 安装 项目依赖:
pip install torch==2.5.1 torchvision==0.20.1 --index-url https://download.pytorch.org/whl/cu121
git clone https://github.com/rmaphoh/RETFound/
cd RETFound
pip install -r requirements.txt
7.2.2 使用 RETFound 权重进行微调
- 首先在 HuggingFace 平台获取预训练模型(需注册账号并填写表单),然后进入步骤 2::
- 登录您的 HuggingFace 账户,在该账户中可创建并复制 HuggingFace token。
huggingface-cli login --token YOUR_HUGGINGFACE_TOKEN
可选:若您的机器或服务器因网络防火墙无法访问HuggingFace,请执行以下命令(如能正常访问则无需执行):
export HF_ENDPOINT=https://hf-mirror.com
-
若需对DINOv2与DINOv3进行微调,请访问其GitHub仓库下载模型权重,并将其置于RETFound文件夹中。
-
请将数据整理为如下目录结构(本研究所用的公开数据集可于此处下载:公开数据集)。
├── data folder
├──train
├──class_a
├──class_b
├──class_c
├──val
├──class_a
├──class_b
├──class_c
├──test
├──class_a
├──class_b
├──class_c
- 通过运行
sh train.sh开始微调。
在 train.sh 中,可通过修改超参数 MODEL、MODEL_ARCH、FINETUNE 来选择模型:

注意:将 DATA_PATH 更改为您的数据集目录。
# ==== Model settings ====
# adaptation {finetune,lp}
ADAPTATION="finetune"
MODEL="RETFound_dinov2"
MODEL_ARCH="retfound_dinov2"
FINETUNE="RETFound_dinov2_meh"
# ==== Data settings ====
# change the dataset name and corresponding class number
DATASET="MESSIDOR2"
NUM_CLASS=5
# =======================
DATA_PATH="PATH TO THE DATASET"
TASK="${MODEL_ARCH}_${DATASET}_${ADAPTATION}"
torchrun --nproc_per_node=1 --master_port=48766 main_finetune.py \
--model "${MODEL}" \
--model_arch "${MODEL_ARCH}" \
--finetune "${FINETUNE}" \
--savemodel \
--global_pool \
--batch_size 24 \
--world_size 1 \
--epochs 50 \
--nb_classes "${NUM_CLASS}" \
--data_path "${DATA_PATH}" \
--input_size 224 \
--task "${TASK}" \
--adaptation "${ADAPTATION}"
- 仅用于评估(请在此下载数据与模型检查点;将下方的 DATA_PATH 进行修改)
# ==== Model/settings (match training) ====
ADAPTATION="finetune"
MODEL="RETFound_dinov2"
MODEL_ARCH="retfound_dinov2"
FINETUNE="RETFound_dinov2_meh"
# ==== Data/settings (match training) ====
DATASET="MESSIDOR2"
NUM_CLASS=5
# =======================
DATA_PATH="PATH TO THE DATASET"
TASK="${MODEL_ARCH}_${DATASET}_${ADAPTATION}"
# Path to the trained checkpoint (adjust if you saved elsewhere)
CKPT="./output_dir/${TASK}/checkpoint-best.pth"
# ==== Evaluation only ====
torchrun --nproc_per_node=1 --master_port=48766 main_finetune.py \
--model "${MODEL}" \
--model_arch "${MODEL_ARCH}" \
--savemodel \
--global_pool \
--batch_size 128 \
--world_size 1 \
--nb_classes "${NUM_CLASS}" \
--data_path "${DATA_PATH}" \
--input_size 224 \
--task "${TASK}" \
--adaptation "${ADAPTATION}" \
--eval \
--resume "${CKPT}"
8. 参考文献
1. Rajpurkar, P., Chen, E., Banerjee, O. & Topol, E. J. AI in health and medicine. Nat. Med. https://doi.org/10.1038/s41591-021-01614-0 (2022).
2. Willemink, M. J. et al. Preparing medical imaging data for machine learning. Radiology295, 4–15 (2020).
3. Topol, E. J. High-performance medicine: the convergence of human and artificialintelligence. Nat. Med. 25, 44–56 (2019).
4. Yu, K.-H., Beam, A. L. & Kohane, I. S. Artificial intelligence in healthcare. Nat. Biomed. Eng. 2, 719–731 (2018).
5. Liu, X. et al. A comparison of deep learning performance against health-careprofessionals in detecting diseases from medical imaging: a systematic review andmeta-analysis. Lancet Digit. Health 1, e271–e297 (2019).
6. De Fauw, J. et al. Clinically applicable deep learning for diagnosis and referral in retinaldisease. Nat. Med. 24, 1342–1350 (2018).
7. Tiu, E. et al. Expert-level detection of pathologies from unannotated chest X-ray imagesvia self-supervised learning. Nat. Biomed. Eng. https://doi.org/10.1038/s41551-022-00936-9(2022).
8. Krishnan, R., Rajpurkar, P. & Topol, E. J. Self-supervised learning in medicine andhealthcare. Nat. Biomed. Eng. https://doi.org/10.1038/s41551-022-00914-1 (2022).
9. Doersch, C., Gupta, A. & Efros, A. A. Unsupervised visual representation learning bycontext prediction. In Proc. 2015 IEEE International Conference on Computer Vision (edsIkeuchi, K. et al.) 1422–1430 (IEEE, 2015).
10. Moor, M. et al. Foundation models for generalist medical artificial intelligence. Nature616, 259–265 (2023).
11. Jing, L. & Tian, Y. Self-supervised visual feature learning with deep neural networks:a survey. IEEE Trans. Pattern Anal. Mach. Intell. 43, 4037–4058 (2021).
12. Deng, J. et al. ImageNet: a large-scale hierarchical image database. In Proc. 2009 IEEEConference on Computer Vision and Pattern Recognition (eds Essa, I., Kang, S. B. &Pollefeys, M.) 248–255 (IEEE, 2009).
13. Chen, T., Kornblith, S., Swersky, K., Norouzi, M. & Hinton, G. Big self-supervised modelsare strong semi-supervised learners. In Proc. 34th International Conference on NeuralInformation Processing Systems (ed. Ranzato, M.) 22243–22255 (Neurips, 2020).
14. Chen, X., Xie, S. & He, K. An empirical study of training self-supervised vision transformers. In Proc. 2021 IEEE/CVF International Conference on Computer Vision (eds Hassner, T. et al.)9640–9649 (IEEE, 2021).
15. He, K. et al. Masked autoencoders are scalable vision learners. In Proc. 2022 IEEE/CVFConference on Computer Vision and Pattern Recognition (eds Dana, K. et al.) 16000–16009 (IEEE, 2022).
16. Chen, T., Kornblith, S., Norouzi, M. & Hinton, G. A simple framework for contrastivelearning of visual representations. In Proc. 37th International Conference on MachineLearning (eds Iii, H. D. & Singh, A.) 1597–1607 (PMLR, 2020).
17. Wagner, S. K. et al. Insights into systemic disease through retinal imaging-basedoculomics. Transl. Vis. Sci. Technol. 9, 6 (2020).
18. Cheung, C. Y. et al. A deep-learning system for the assessment of cardiovascular diseaserisk via the measurement of retinal-vessel calibre. Nat. Biomed. Eng. 5, 498–508 (2021).
19. Mutlu, U. et al. Association of retinal neurodegeneration on optical coherence tomographywith dementia: a population-based study. JAMA Neurol. 75, 1256–1263 (2018).
20. Thomson, K. L., Yeo, J. M., Waddell, B., Cameron, J. R. & Pal, S. A systematic review andmeta-analysis of retinal nerve fiber layer change in dementia, using optical coherencetomography. Alzheimers Dement. 1, 136–143 (2015).
21. Ko, F. et al. Association of retinal nerve fiber layer thinning with current and futurecognitive decline: a study using optical coherence tomography. JAMA Neurol. 75,1198–1205 (2018).
22. McGeechan, K. et al. Meta-analysis: retinal vessel caliber and risk for coronary heartdisease. Ann. Intern. Med. 151, 404–413 (2009).
23. Wong, T. Y. & Mitchell, P. Hypertensive retinopathy. N. Engl. J. Med. 351, 2310–2317 (2004).
24. Günthner, R. et al. Impaired retinal vessel dilation predicts mortality in end-stage renaldisease. Circ. Res. https://doi.org/10.1161/CIRCRESAHA.118.314318 (2019).
25. Diaz-Pinto, A. et al. Predicting myocardial infarction through retinal scans and minimalpersonal information. Nat. Mach. Intell. 4, 55–61 (2022).
26. Azizi, S. et al. Robust and efficient medical imaging with self-supervision. Nat. Biomed. Eng. 7, 756–779 (2023)27. Li, X., Jia, M., Islam, M. T., Yu, L. & Xing, L. Self-supervised feature learning via exploitingmulti-modal data for retinal disease diagnosis. IEEE Trans. Med. Imaging 39, 4023–4033(2020).
28. Burlina, P., Paul, W., Liu, T. Y. A. & Bressler, N. M. Detecting anomalies in retinal diseasesusing generative, discriminative, and self-supervised deep learning. JAMA Ophthalmol. 140, 185–189 (2022).
29. Holmberg, O. G. et al. Self-supervised retinal thickness prediction enables deep learningfrom unlabelled data to boost classification of diabetic retinopathy. Nat. Mach. Intell. 2,719–726 (2020).
30. Truong, T., Mohammadi, S. & Lenga, M. How transferable are self-supervised features inmedical image classification tasks? In Proc. 2021 Machine Learning for Health (eds Roy, S. et al.) 54–74 (PMLR, 2021)31. Bommasani, R. et al. On the opportunities and risks of foundation models. Preprint athttps://arxiv.org/abs/2108.07258 (2021).
32. Wiggins, W. F. & Tejani, A. S. On the opportunities and risks of foundation models fornatural language processing in radiology. Radiol. Artif. Intell. 4, e220119 (2022).
33. Gulshan, V. et al. Development and validation of a deep learning algorithm for detectionof diabetic retinopathy in retinal fundus photographs. J. Am. Med. Assoc. 316, 2402–2410(2016).
34. Kermany, D. S. et al. Identifying medical diagnoses and treatable diseases byimage-based deep learning. Cell 172, 1122–1131.e9 (2018).
35. Wagner, S. K. et al. AlzEye: longitudinal record-level linkage of ophthalmic imaging andhospital admissions of 353 157 patients in London, UK. BMJ Open 12, e058552 (2022).
36. Bycroft, C. et al. The UK Biobank resource with deep phenotyping and genomic data. Nature 562, 203–209 (2018).
37. Caron, M. et al. Unsupervised learning of visual features by contrasting clusterassignments. In Proc. 34th International Conference on Neural Information ProcessingSystems (ed. Ranzato, M.) 9912–9924 (Neurips, 2020).
38. Caron, M. et al. Emerging properties in self-supervised vision transformers. In Proc. 2021IEEE/CVF International Conference on Computer Vision (eds Hassner, T. et al.) 9650–9660(IEEE, 2021).
39. Porwal, P. et al. IDRiD: diabetic retinopathy - segmentation and grading challenge. Med. Image Anal. 59, 101561 (2020).
40. Abràmoff, M. D. et al. Automated analysis of retinal images for detection of referablediabetic retinopathy. JAMA Ophthalmol. 131, 351–357 (2013).
41. Decencière, E. et al. Feedback on a publicly distributed image database: the Messidordatabase. Image Anal. Stereol. 33, 231–234 (2014).
42. Chefer, H., Gur, S. & Wolf, L. Transformer interpretability beyond attention visualization. In Proc. 2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition (edsForsyth, D. et al.) 782–791 (IEEE, 2021).
43. Sung, K. R. et al. Effects of age on optical coherence tomography measurements of healthyretinal nerve fiber layer, macula, and optic nerve head. Ophthalmology 116, 1119–1124 (2009).
44. Wong, T. Y., Klein, R., Klein, B. E. K., Meuer, S. M. & Hubbard, L. D. Retinal vessel diametersand their associations with age and blood pressure. Invest. Ophthalmol. Vis. Sci. 44,4644–4650 (2003).
45. Hanssen, H., Streese, L. & Vilser, W. Retinal vessel diameters and function incardiovascular risk and disease. Prog. Retin. Eye Res. 91, 101095 (2022).
46. Esteva, A. et al. Dermatologist-level classification of skin cancer with deep neuralnetworks. Nature 542, 115–118 (2017).
47. Guo, C., Pleiss, G., Sun, Y. & Weinberger, K. Q. On calibration of modern neural networks. In Proc. 34th International Conference on Machine Learning (eds Precup, D. & Teh, Y. W.)Vol. 70, 1321–1330 (PMLR, 2017).
48. Ayhan, M. S. et al. Expert-validated estimation of diagnostic uncertainty for deep neuralnetworks in diabetic retinopathy detection. Med. Image Anal. 64, 101724 (2020).
49. Singhal, K. et al. Large language models encode clinical knowledge. Nature 620, 172–180(2023).
50. Singhal, K. et al. Towards expert-level medical question answering with large languagemodels. Preprint at https://arxiv.org/abs/2305.09617 (2023).
引用说明: Zhou, Y., Chia, M.A., Wagner, S.K. et al. A foundation model for generalizable disease detection from retinal images. Nature 622, 156–163 (2023). https://doi.org/10.1038/s41586-023-06555-x
版权说明:
youcans@xidian 作品,转载必须标注原文链接:
【医学影像 AI】用于可泛化疾病检测的视网膜图像基础模型(https://youcans.blog.csdn.net/article/details/154059838)
Crated:2025-10
更多推荐
 已为社区贡献207条内容
已为社区贡献207条内容








所有评论(0)