论文阅读 2025-9-13 论文阅读随心记
大模型自我反思被定义为两阶段的问同一个问题,但是思考多次。
·
随便记录一下最近阅读的几篇论文
1. Does DINOv3 Set a New Medical Vision Standard?
第一章 动机 (Motivation)
自然图像领域的成功范式:大型语言模型(LLMs)和视觉基础模型(如 DINO 系列)证明,通过自监督学习(SSL)在海量无标注数据上进行预训练,可以学习到极其强大且通用的表征能力,并遵循显著的“缩放定律”(模型越大、数据越多,性能越好)。
医学影像领域的独特挑战:
- 数据稀缺与获取困难:医学数据标注成本极高,且受隐私、法规限制,难以构建大规模数据集。
- 模态极度多样化:包括 2D(X光、病理切片)、3D(CT、MRI)、灰度图、RGB图像等多种成像技术,每种都需要不同的视觉理解能力。
- 对细微特征敏感:诊断依赖于捕捉图像中微小、细微的异常模式,因此对视觉表征的质量要求极高。
核心矛盾与问题: 医学领域亟需一个强大的、现成的(off-the-shelf)视觉特征提取器,但受限于上述挑战,很难从头训练一个大规模医学视觉基础模型。那么,一个直接的问题是:在自然图像上训练出的顶尖模型,其能力能否直接迁移到医学领域? 这就引出了论文要探究的几个根本性问题。
第二章 论文基准测试设置 (Benchmark Setup)
1. 2D医学图像分类 (2D Medical Image Classification)
任务描述:处理二维平面图像进行诊断分类。
方法:直接将2D图像输入DINOv3编码器,获取图像级特征后进行线性分类或更复杂的下游训练。
使用的数据集与评估协议:
| 数据集 | 模态 | 内容与规模 | 任务 | 评估协议 |
|---|---|---|---|---|
| NIH-14 | 胸部X光 | 112,120张图像,30,805名患者 | 14种胸廓疾病的多标签分类 | 严格遵守官方提供的患者划分 |
| RSNA-Pneumonia | 胸部X光 | 29,700张图像 | 肺炎分类(二分类) | 遵循MGCA论文提出的标准化数据划分方法 |
| Camelyon16 | 病理WSI (RGB) | 399张淋巴结切片 | 乳腺癌转移检测(肿瘤 vs. 正常) | 1. 官方划分(270训练/129测试) 2. 多折交叉验证 on test set 3. 在Camelyon17上测试泛化能力(跨中心) |
| Camelyon17 | 病理WSI (RGB) | 多中心数据集,500张切片(100名患者) | 作为域外泛化测试集 | 使用其官方训练集作为测试集(因官方测试标注未公开),评估从Camelyon16训好的模型在Unseen子集上的表现 |
| BCNB | 病理WSI (RGB) | 1,058名患者的早期乳腺癌活检切片 | 5项预测任务: - 腋窝淋巴结(ALN)转移状态 - 分子状态(ER, PR, HER2, Ki67) |
5折交叉验证,每折内按 7:1:2 划分训练/验证/测试集。使用CLAM方法提取图像块(~968 patches/WSI) |
2. 3D医学图像分类 (3D Medical Image Classification)
- 任务描述:对三维体积数据(如CT、MRI)进行整体分类。 核心方法:采用 “切片级特征提取 + 聚合” 策略:
- 独立处理:将3D体积的每一个2D切片分别输入DINOv3 backbone,得到每个切片的特征嵌入(embedding)。
- 特征聚合:将所有切片的特征通过平均池化(Mean Pooling) 聚合为一个代表整个体积的特征向量。
- 下游分类:用聚合后的特征训练分类器。
使用的数据集与评估协议:
| 数据集 | 模态 | 内容与规模 | 任务 | 评估协议 |
|---|---|---|---|---|
| CT-RATE | 非增强CT | 47,000个CT体积,20,000名患者 | 18种临床异常的多标签二分类 | 使用官方数据划分。采用两种下游评估方法: 1. 零样本 k-NN 2. 线性探测(Linear Probing) |
3. 3D医学图像分割 (3D Medical Image Segmentation)
- 任务描述:在三维体积数据上进行体素级的预测,以分割解剖结构或病变。
- 核心方法:采用 “逐片特征提取 + 伪3D重构” 策略
- 逐片处理:将3D体积的每一个2D切片分别输入DINOv3,并保留完整的2D特征图(而非图像级嵌入)。
- 构建伪3D特征体积:将所有2D特征图沿切片方向堆叠,形成一个3D的特征体积
- 轻量级分割头:将这个伪3D特征体积输入一个轻量的3D分割头(如UNet式 decoder),最终生成体素级预测。
使用的数据集与评估协议:
| 数据集 | 模态 | 内容与任务 | 评估协议 |
|---|---|---|---|
| Medical Segmentation Decathlon (MSD) | CT, MRI | 10个不同的3D分割任务,涵盖脑肿瘤、心脏、肝脏、海马体、前列腺、肺、胰腺、肝血管、脾脏和结肠 | 官方评估平台已关闭,采用 5折交叉验证。遵循此前工作的标准协议(体积标准化、随机旋转/翻转等数据增强) |
| CREMI | 电子显微镜(EM) | 3个子集(A,B,C),难度递增,用于神经元分割 | 每个子集:前100张切片训练,后25张测试 |
| AC3/AC4 | 电子显微镜(EM) | 两个密集标注的EM体积,用于神经元分割 | 在AC4(前80切片)上训练,在AC3(前100切片)上测试 -> 测试域外泛化 |
| AutoPET-II | PET/CT | 1,014个全身PET/CT扫描,用于肿瘤病灶分割 | 使用官方划分的训练/验证集。强度标准化,使用随机旋转/翻转等数据增强 |
| HECKTOR 2022 | PET/CT | 882个头颈部PET/CT扫描,用于原发灶(GTVp)和淋巴结(GTVn)肿瘤分割 | 遵循官方挑战协议和预处理流程(图像配准、强度标准化) |
实验baseline model设置
| 模型 | 参数量 | 预训练数据 | 数据类型 | 数据模态 | 学习范式 |
|---|---|---|---|---|---|
| BiomedCLIP | 86M | 1500万图像-文本对 | 2D 图像-文本 | 多样生物医学图像 | 文本监督 |
| CT-CLIP | 86M | 5万体积+报告 | 3D 体积-文本 | 胸部CT | 文本监督 |
| UNI | 304M | 1亿图像块 | 2D 图像块 | 头部/腹部/胸部CT | 视觉自监督 |
| CONCH | 86M | 117万 2D 块-文本对 | 2D 块-文本 | 病理学 | 文本监督 |
| DINOv3-S/B/L | 22M/86M/304M | 17亿自然图像 | 2D 图像 | 自然图像 | 视觉自监督 |
第三章 实验结论分析
| 任务类别 | 具体任务与数据集 | 实验结论 | 原因分析 |
|---|---|---|---|
| 2D分类 | 胸部X光分类 (NIH-14, RSNA-Pneumonia) | 表现出色,确立新基线。DINOv3-L在NIH-14上AUC 0.7865,超越医学专用模型BiomedCLIP (0.7771)。在RSNA上与BiomedCLIP性能接近。 | DINOv3从自然图像中学到的物体结构和形状表征能力,与X光片中基于宏观结构异常进行诊断的模式高度吻合。 |
| 2D分类 | 病理切片(WSI)分类 (Camelyon16, Camelyon17, BCNB) | 性能显著落后。AUC (~0.84) 远低于病理专用模型UNI和CONCH (AUC >0.96),仅与ResNet50相当。泛化能力差。 | 病理分析依赖细粒度的纹理和细胞模式,与DINOv3关注的“物体”级特征不匹配。表明领域专业化预训练绝对必要。 |
| 3D分类 | 3D CT体积分类 (CT-RATE) | 性能卓越,显著超越基线。DINOv3-B (线性探测) AUC 0.798,显著高于专用基线CT-CLIP (0.731)。 | DINOv3的2D特征通过切片平均池化聚合后,能有效表征整个3D体积,捕捉与诊断相关的视觉模式。 |
| 3D分割 | 多器官分割 (MSD) | 都是物体级别特征,表现尚可但平庸。为自监督方法设立新SOTA基线,但整体性能(71% Dice)远低于全监督nnU-Net (81.4% Dice)。在个别任务(肺、脾)上达到最佳。 | 简单的“冻结主干+逐片处理”策略不足以击败端到端训练的复杂3D架构。但其强大的2D特征是一个“不错的起点”。 |
| 3D分割 | 神经元分割 (EM: CREMI, AC3/4) | 灾难性失败。误差指标(VOI, ARAND)比经典方法高出十倍以上。 | DINOv3特征缺乏高频纹理细节,无法描绘神经元间错综复杂的边界。自然图像与EM图像领域差距过大。 |
| 3D分割 | 肿瘤分割 (PET/CT: AutoPET-II, HECKTOR) | 性能极差。Dice分数极低(<15%),远低于现有模型(>50%)。 | DINOv3特征适用于解剖结构,但无法解读PET数据中的功能性代谢信息。这是与自然图像模式的根本性背离。 |
| 缩放定律 | 所有任务 | 缩放定律不一致且不可靠。性能不随模型变大(S->B->L)或分辨率提高而单调增加。最佳模型规模因任务而异。 | 自然图像与医学图像存在领域差距。简单地堆砌计算资源不能解决所有问题,必须为不同任务仔细选择模型规模和分辨率。 |
2. Training Language Models to Self-Correct via Reinforcement Learning(2024)
Google技术报告
任务定义: 大模型自我反思被定义为两阶段的问同一个问题,但是思考多次

主要方法以及实验可以在下面表示:
主要方法的训练过程: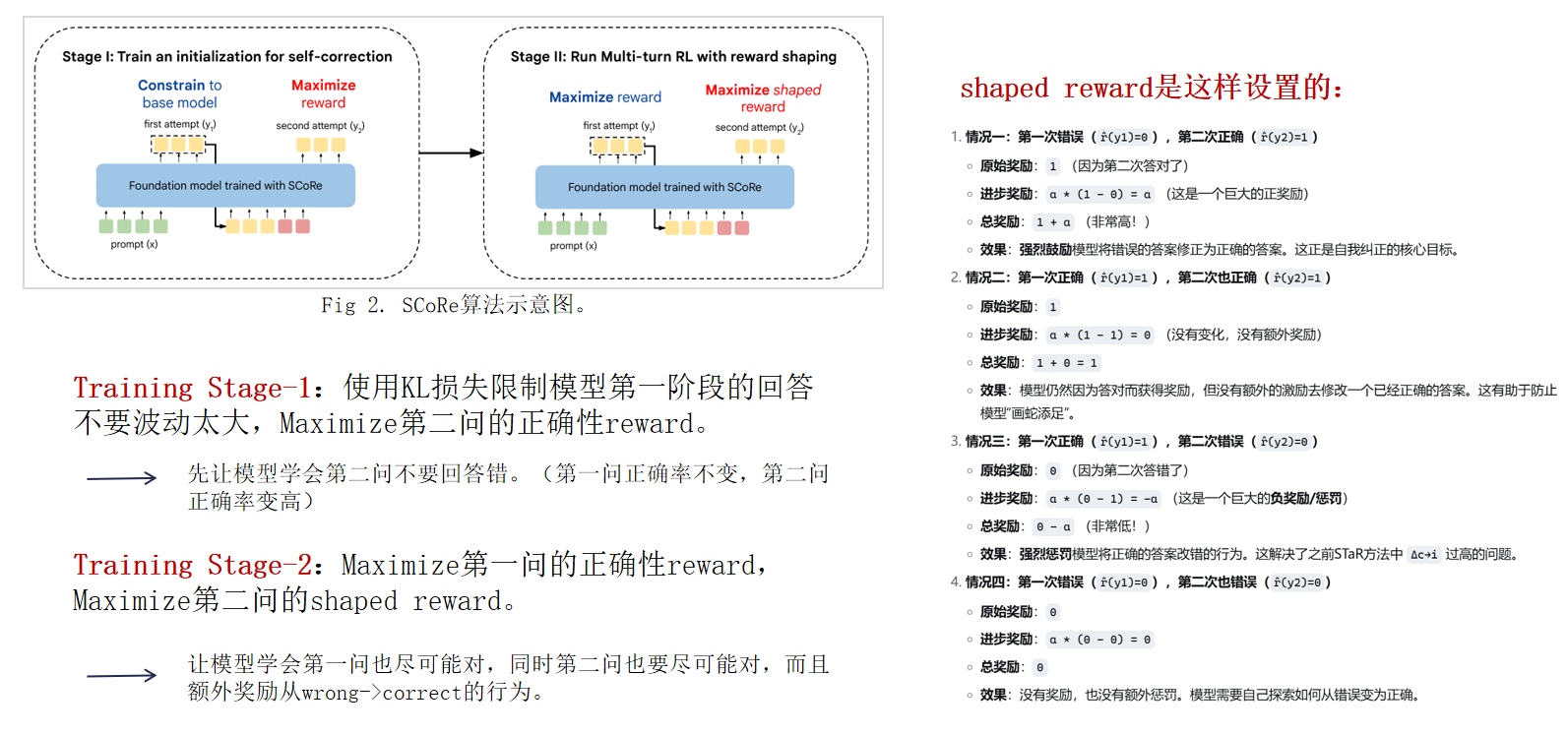
更多推荐
 已为社区贡献5条内容
已为社区贡献5条内容








所有评论(0)