(R语言)基于脑肿瘤数据集的生存分析
本文的生存分析基于R语言中ISLR2包里的Braincancer数据集,即脑肿瘤数据集。0:删失——患者在观察期内未发生事件(如失访,研究结束时仍存活;1:事件发生——患者在观察期内发生目标事件(如肿瘤相关死亡)
一、数据集介绍
本文的生存分析基于R语言中ISLR2包里的Braincancer数据集,即脑肿瘤数据集。
数据集中包含88位脑肿瘤患者的相关信息,具体变量如下所示:
| 变量名称 | 变量内容 | 变量类型 |
| sex:患者性别 | (female,male) | 类别变量 |
| diagnosis:肿瘤诊断类型 | (meningioma,lg glioma,hg glioma,other) | 类别变量 |
| loc:肿瘤位置 | (Infratentorial,Supratentorial) | 类别变量 |
| ki:ki-67指数 | 处于增值期的肿瘤细胞比例(单位:%) | 数值变量 |
| gtv:肿瘤实体体积 | 表示肿瘤大体体积(单位:立方厘米) | 数值变量 |
| stereo:放疗类型 | (SRS,SRT) | 类别变量 |
| status:生存状态 |
0:删失——患者在观察期内未发生事件(如失访,研究结束时仍存活; 1:事件发生——患者在观察期内发生目标事件(如肿瘤相关死亡) |
数值变量 |
| time:生存时间 | 从 “起始时间点”(如确诊日期、治疗开始日期)到 “事件发生时间” 或 “删失时间” 的间隔 | 数值变量 |
二、绘制Kaplan-Meier生存曲线,并添加±1个标准误的置信带
library(dplyr)
# 加载脑肿瘤数据
data(BrainCancer)
data <- BrainCancer
## 问题a: 绘制Kaplan-Meier生存曲线并添加置信带
fit_km <- survfit(Surv(time, status) ~ 1, data = data)
# 绘制Kaplan-Meier曲线
autoplot(fit_km, conf.int = TRUE, censor = TRUE) +
ggtitle("Kaplan-Meier Survival Curve with 95% CI") +
xlab("Time (days)") +
ylab("Survival Probability")这段代码用于对脑肿瘤患者数据进行生存分析,并通过可视化化方式展示患者群体的生存概率随时间的变化趋势,绘制 了Kaplan-Meier 生存曲线。
运用Surv(time,status):创建生存分析的核心对象,其中包含两个关键变量,即生存时间time和生存状态status。
最终绘制的生存曲线如下所示:
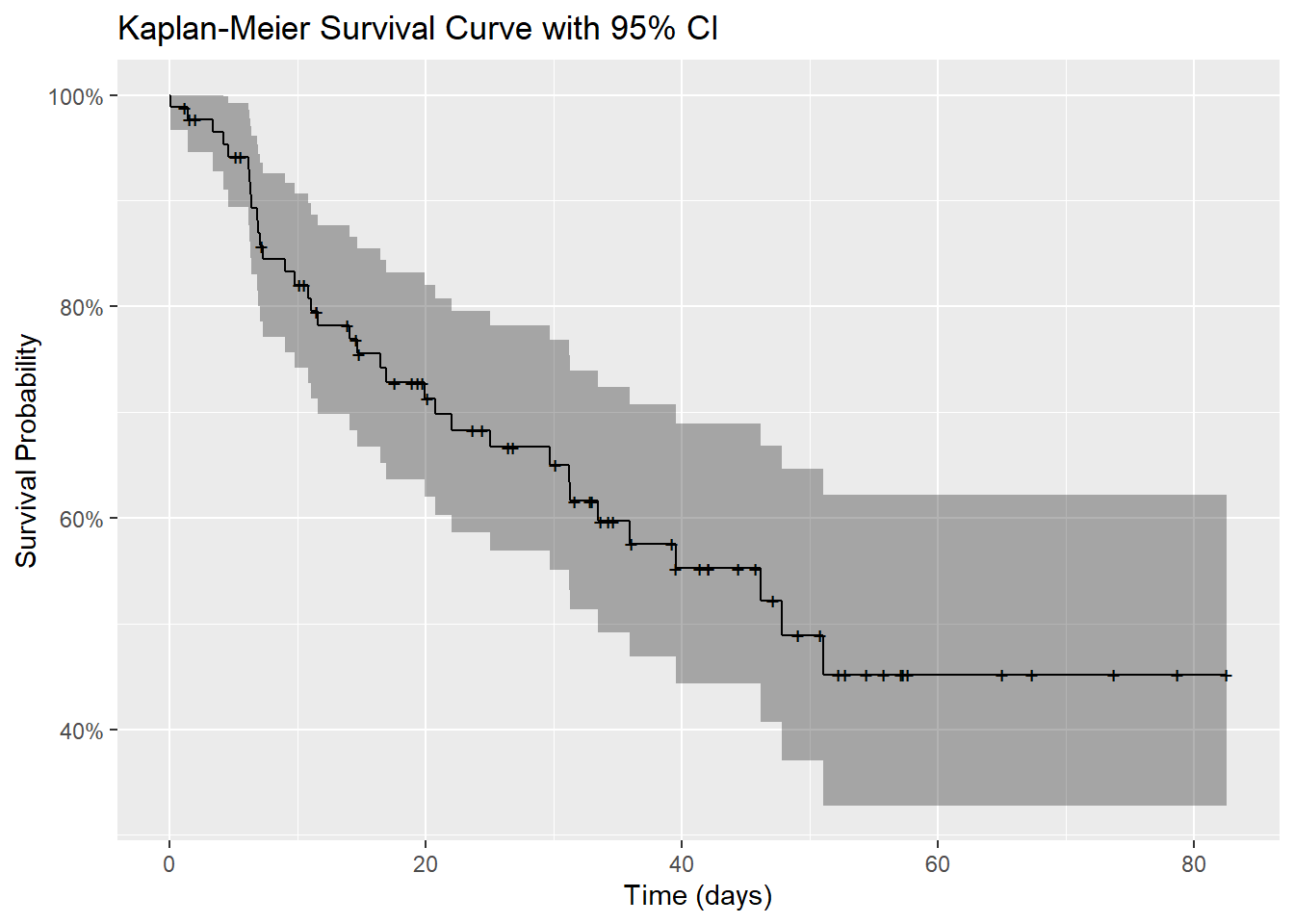
这张 Kaplan - Meier 生存曲线展示了脑肿瘤患者群体的生存概率随时间(以天为单位)的变化情况,结合曲线形态、置信区间和删失标记,可从以下几个方面分析:
1. 生存概率的整体趋势
-
曲线从左上方(时间为 0,生存概率接近 100%)开始,随着时间推移逐渐下降,这符合生存分析的一般规律,即随着时间增加,发生终点事件(如死亡等)的患者增多,生存概率降低。
-
在前 40 天左右,生存概率下降速度较快,之后下降速度逐渐变缓,到后期(约 60 天之后)生存概率基本趋于平稳,维持在 45% 左右。这表明在研究的早期阶段,患者的生存风险较高,而到了后期,生存概率相对稳定,剩余患者的生存情况变化不大。
2.置信区间(灰色阴影部分)
-
灰色阴影区域代表 95% 的置信区间,反映了生存概率估计的不确定性
3. 删失标记(“+” 号)
-
曲线上的 “+” 号表示删失数据点,即对应时间点有患者未发生终点事件(如失访、研究结束时仍存活等)。
-
从图中可以看到,在整个时间范围内都有 “+” 号分布,说明在研究过程中,不同时间点都存在删失情况。
二、使用Bootstrap方法估计每个时间点生存曲线的标准误
## 问题b: Bootstrap估计标准误
# 定义bootstrap函数
boot_km <- function(data, indices) {
d <- data[indices, ]
fit <- survfit(Surv(time, status) ~ 1, data = d)
# 获取原始fit的所有时间点
all_times <- summary(fit_km)$time
# 对当前bootstrap样本的生存概率进行插值
surv_prob <- approx(fit$time, fit$surv, xout = all_times,
yleft = 1, yright = min(fit$surv, na.rm = TRUE))$y
return(surv_prob)
}
# 进行bootstrap
set.seed(123)
boot_results <- boot(data = data, statistic = boot_km, R = 200)
# 计算bootstrap标准误
bootstrap_se <- apply(boot_results$t, 2, sd, na.rm = TRUE)
# 获取原始KM估计的标准误
original_se <- summary(fit_km)$std.err
# 比较两种标准误
comparison <- data.frame(
Time = summary(fit_km)$time,
Original_SE = original_se,
Bootstrap_SE = bootstrap_se[1:length(original_se)]
)
# 绘制比较图
ggplot(comparison, aes(x = Time)) +
geom_line(aes(y = Original_SE, color = "Original KM SE")) +
geom_line(aes(y = Bootstrap_SE, color = "Bootstrap SE")) +
labs(title = "Comparison of Standard Errors",
x = "Time (days)",
y = "Standard Error",
color = "Method") +
theme_minimal()这段代码的核心功能是通过Bootstrap(自助法) 估计 Kaplan-Meier 生存曲线的标准误,并将其与传统方法计算的标准误进行比较,以此评估生存概率估计的不确定性。以下是这段代码中一些关键点的说明:
1.Bootstrap方法的原理
Bootstrap 方法的核心思想是通过对原始样本进行有放回的重复抽样,生成多个与原始样本容量相同的 “自助样本”(bootstrap sample)。由于是有放回抽样,每个自助样本中可能会有重复的观测值,也可能会有部分原始样本中的观测值未被抽到。
基于这些自助样本,多次计算感兴趣的统计量(如均值、中位数、标准差、回归系数等),进而利用这些统计量的分布情况来评估原始样本统计量的不确定性,比如估计其标准误、构建置信区间等。从本质上讲,它是利用样本数据本身来模拟抽样分布,避免了对总体分布的强假设。
2.代码中的indices由boot函数自动生成的抽样索引,实现 “有放回抽样”
3.插值(approx函数):由于不同 bootstrap 样本的事件时间可能不同,通过插值将各样本的生存概率 “对齐” 到原始 KM 曲线的时间点上,确保后续计算可比
-
yleft = 1:当时间早于 bootstrap 样本的第一个事件时间时,生存概率默认为 1(所有患者存活)。 -
yright = min(fit$surv):当时间晚于 bootstrap 样本的最后一个事件时间时,生存概率取该样本的最小生存概率(保守估计)。
4.boot函数:来自boot包,核心参数:
data:原始数据集(BrainCancer)。
statistic = boot_km:指定每次抽样后计算的统计量(即boot_km函数返回的生存概率)。
R = 200:设置 bootstrap 抽样次数(生成 200 个模拟样本),次数越多估计越稳定(但计算时间更长)。
然后我们再计算Bootstrap方法下的标准误,并与传统方法下的标准误进行比较。比较的可视化结果如下所示:
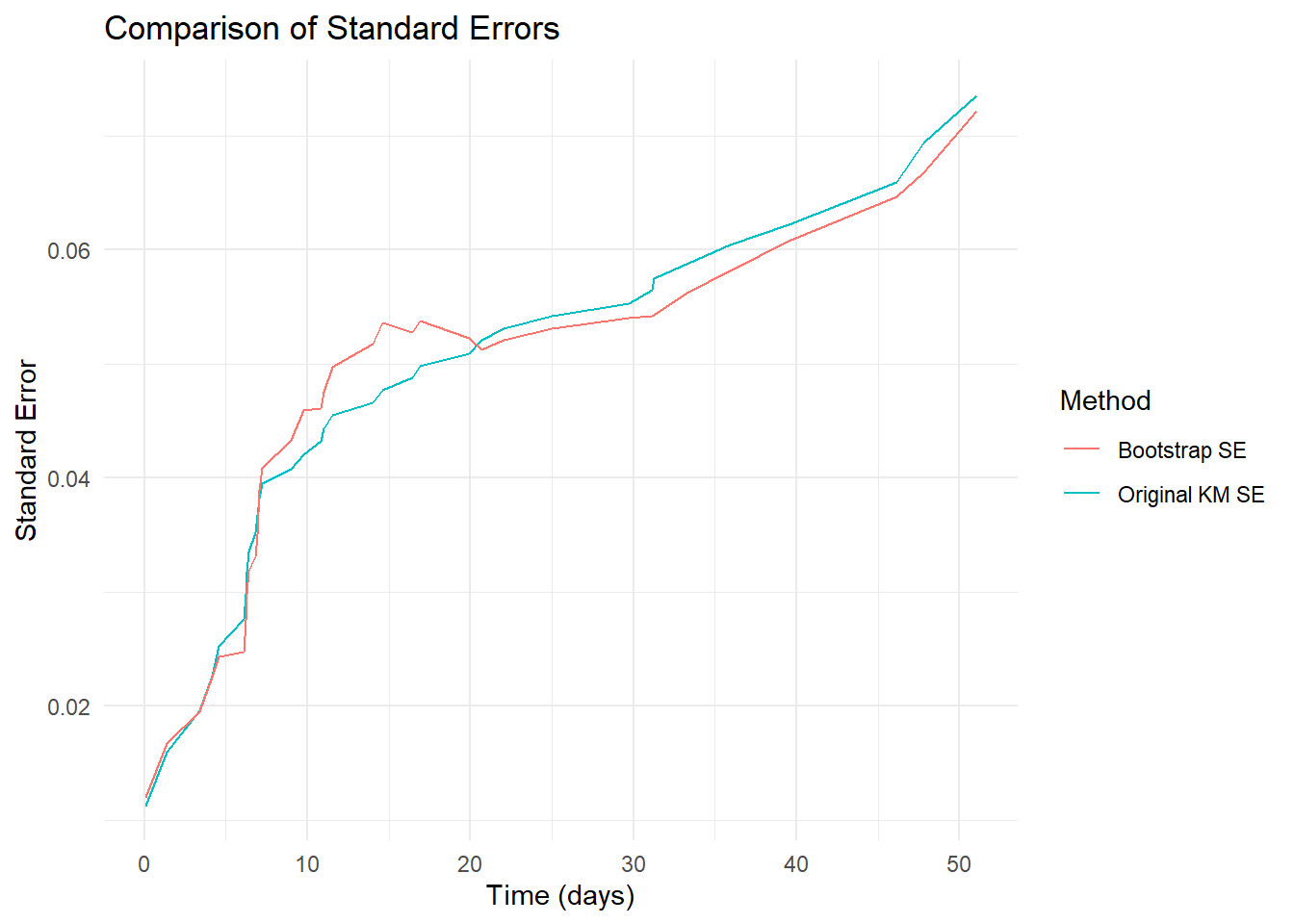
通过上图可以初步分析得到:
红色线表示Bootsrap方法得到的标准误,青色线表示传统方法得到的标准误
1.两种方法的标准误整体趋势一致:
两条曲线均随时间(横轴,单位:天)增加而上升,说明生存概率的估计不确定性随时间增大(时间越久,经历事件或删失的患者越多,剩余样本量越少,生存概率的估计越不稳定)。
2.Bootstrap与传统方法的差异:
从图中可以看到在初期几天时,两条曲线几乎重合;在7--20天这个时期,Bootsrap方法的标准误要高于传统方法;而在第20天开始,一直到后期,传统方法的标准误一直都要高于Bootstrap方法。这可以说明,在后期删失率更高时,Bootstrap方法比传统方法更有优势。
三、拟合包含所有预测变量的Cox比例风险模型以预测生存时间
## 问题c: 拟合Cox比例风险模型
cox_fit <- coxph(Surv(time, status) ~ sex + diagnosis + loc + ki + gtv + stereo,
data = data)
# 总结模型
summary(cox_fit)这段代码使用survival包中的coxph()函数拟合了一个Cox 比例风险模型,用于分析脑肿瘤患者的生存时间与性别、肿瘤诊断类型、肿瘤位置、ki指数、肿瘤实体体积、放疗方式之间的关系。模型结果如下所示:
## Call:
## coxph(formula = Surv(time, status) ~ sex + diagnosis + loc +
## ki + gtv + stereo, data = data)
##
## n= 87, number of events= 35
## (因为不存在,1个观察量被删除了)
##
## coef exp(coef) se(coef) z Pr(>|z|)
## sexMale 0.18375 1.20171 0.36036 0.510 0.61012
## diagnosisLG glioma 0.91502 2.49683 0.63816 1.434 0.15161
## diagnosisHG glioma 2.15457 8.62414 0.45052 4.782 1.73e-06 ***
## diagnosisOther 0.88570 2.42467 0.65787 1.346 0.17821
## locSupratentorial 0.44119 1.55456 0.70367 0.627 0.53066
## ki -0.05496 0.94653 0.01831 -3.001 0.00269 **
## gtv 0.03429 1.03489 0.02233 1.536 0.12466
## stereoSRT 0.17778 1.19456 0.60158 0.296 0.76760
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## exp(coef) exp(-coef) lower .95 upper .95
## sexMale 1.2017 0.8321 0.5930 2.4352
## diagnosisLG glioma 2.4968 0.4005 0.7148 8.7215
## diagnosisHG glioma 8.6241 0.1160 3.5664 20.8546
## diagnosisOther 2.4247 0.4124 0.6678 8.8031
## locSupratentorial 1.5546 0.6433 0.3914 6.1741
## ki 0.9465 1.0565 0.9132 0.9811
## gtv 1.0349 0.9663 0.9906 1.0812
## stereoSRT 1.1946 0.8371 0.3674 3.8839
##
## Concordance= 0.794 (se = 0.04 )
## Likelihood ratio test= 41.37 on 8 df, p=2e-06
## Wald test = 38.7 on 8 df, p=6e-06
## Score (logrank) test = 46.59 on 8 df, p=2e-07我们将模型结果可视化,结果如下所示:
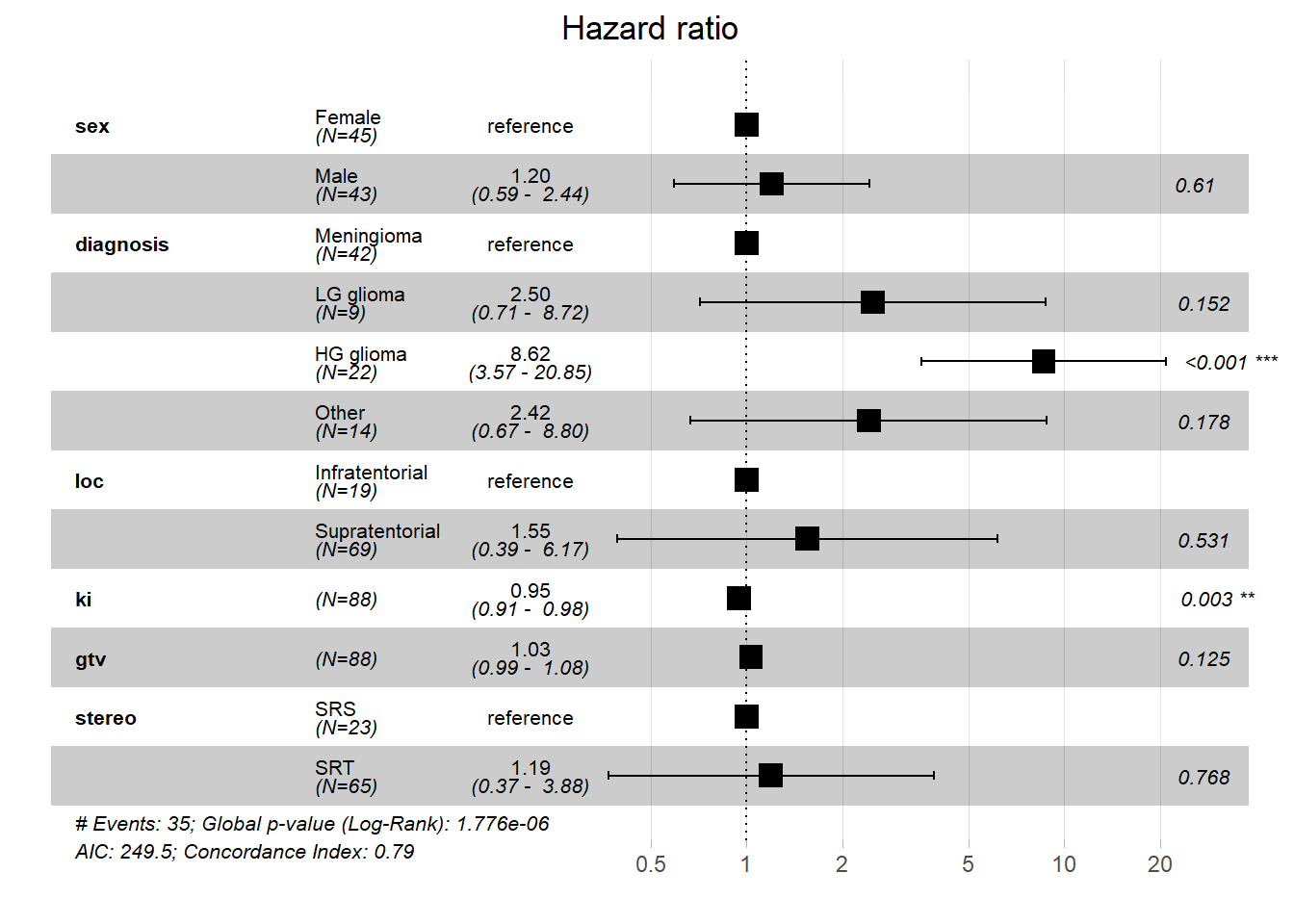
从结果中我们可分析得到:
1.对生存时间有显著影响的变量是diagnosis(诊断类型)和KI指数。
其中diagnosis HG glioma的风险比为8.62,风险最高,即患者的死亡风险是参照组的8.6倍;而diagnosis LG glioma和diagnosisOther的风险比也高于参照组,但它们的p值>0.05,并不显著。
而对于KI指数,它每增加1个单位,死亡风险就显著降低5.4%。
2.对生存时间无显著影响的变量是sex(性别)和loc(肿瘤位置)。
3.模型评估指标 模型的一致性检验Concordance=0.794,很接近于1,这说明模型的预测能力较强。 模型的似然比检验、Wald检验、Score检验p值均<0.05,说明检验均显著,即此模型整体显著优于空模型。
四、按KI值分层并绘制生存曲线
## 问题d: 按ki值分层并绘制KM曲线
# 合并ki=40和ki=60的观测
data <- data %>%
mutate(ki_group = case_when(
ki == 40 ~ "60",
ki == 60 ~ "60",
TRUE ~ as.character(ki)
))
# 确保ki_group是因子
data$ki_group <- factor(data$ki_group, levels = c("70", "80", "90", "100", "60"))
# 分层KM曲线
fit_strata <- survfit(Surv(time, status) ~ ki_group, data = data)
# 绘制分层KM曲线
autoplot(fit_strata, conf.int = TRUE, censor = TRUE) +
ggtitle("Kaplan-Meier Survival Curves by KI Group") +
xlab("Time (days)") +
ylab("Survival Probability") +
labs(color = "KI Group")这段代码的是根据 Ki-67 指数(ki值)对脑肿瘤患者进行分组,然后按分组绘制 Kaplan-Meier 生存曲线,以此分析不同 Ki-67 水平患者的生存差异。因为ki=40仅有1例,所以我们将ki=40合并到ki=60的样本中。
绘制的分层KM生存曲线如下所示:
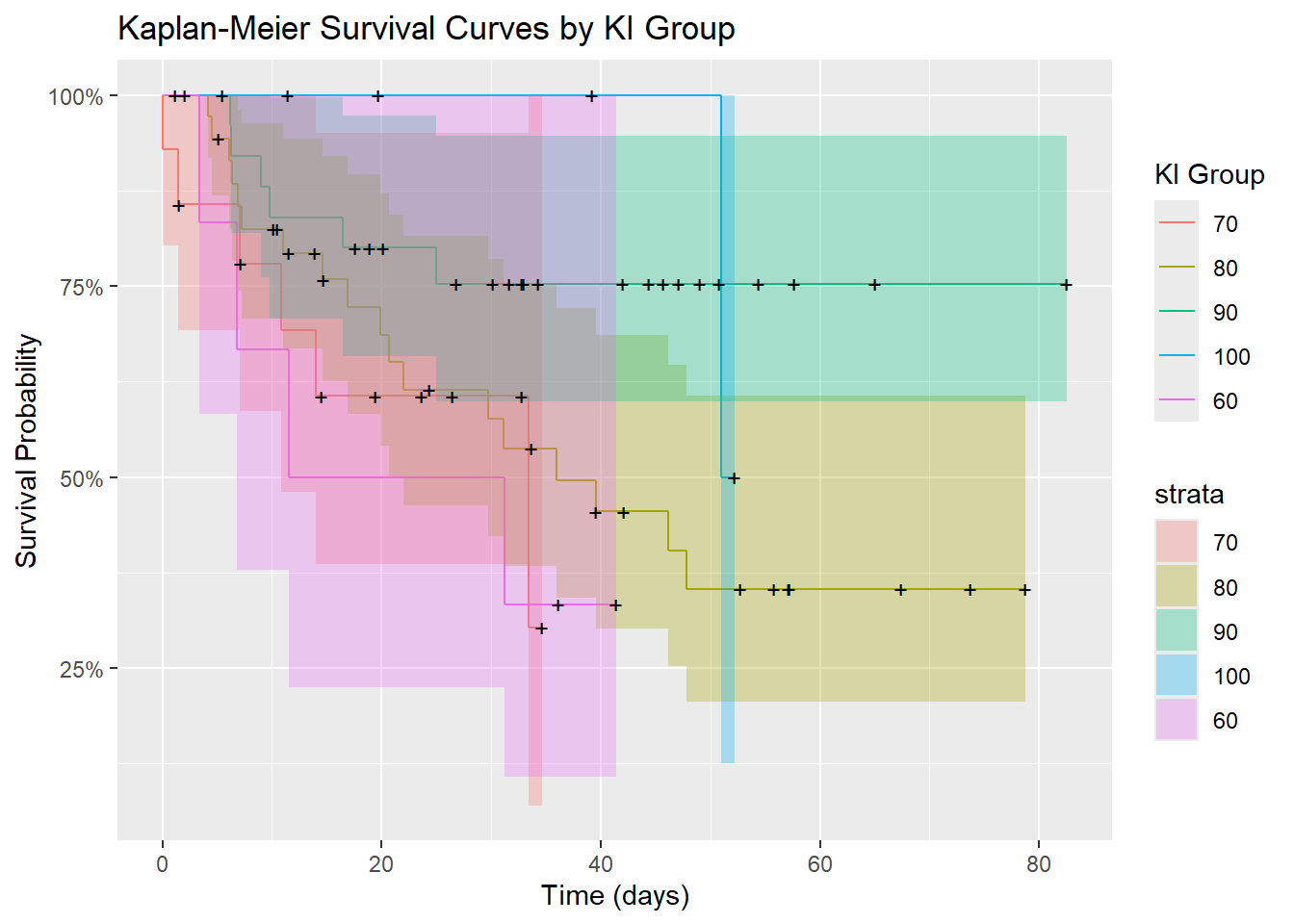
从图中可以直观地看出,Ki - 67 指数与脑肿瘤患者的生存预后存在一定关联。总体而言,Ki - 67 指数越高(如90、100组),患者的生存概率相对越高,预后越好;Ki - 67 指数较低(如60、70组),患者的生存概率相对较低,预后较差。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)