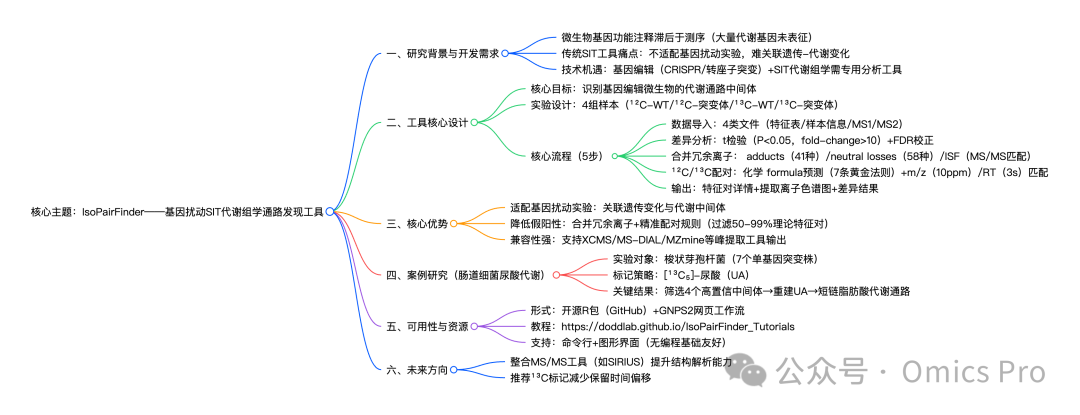
分析破局!IsoPairFinder解锁新通路
斯坦福大学等团队开发的IsoPairFinder,是一款专为基因编辑微生物的稳定同位素示踪(SIT)代谢组学数据设计的计算工具,通过分析配对的未标记(¹²C)和同位素标记(¹³C)数据,结合 “差异分析→合并冗余离子→¹²C/¹³C 特征配对” 核心流程,减少 50-99% 假阳性并优先筛选基因扰动相关的代谢通路中间体;
一段话总结
斯坦福大学等团队开发的IsoPairFinder,是一款专为基因编辑微生物的稳定同位素示踪(SIT)代谢组学数据设计的计算工具,通过分析配对的未标记(¹²C)和同位素标记(¹³C)数据,结合 “差异分析→合并冗余离子→¹²C/¹³C 特征配对” 核心流程,减少 50-99% 假阳性并优先筛选基因扰动相关的代谢通路中间体;该工具以开源 R 包(https://github.com/DoddLab/IsoPairFinder)和 GNPS2 网页工作流形式提供,支持无编程基础用户使用,在肠道细菌尿酸(UA)代谢研究中成功筛选出 4 个关键中间体并重建代谢通路,有效解决了传统 SIT 工具不适配基因扰动实验的痛点,加速微生物代谢通路的发现与验证。
思维导图
详细总结
一、研究背景
微生物代谢通路是细胞功能的核心,但超过 50% 的微生物代谢基因功能未被注释,限制了微生物代谢在健康、生物技术领域的应用。当前技术已实现基因编辑(如 CRISPR、转座子突变)的系统化扰动,但传统稳定同位素示踪(SIT)代谢组学工具(如 X13CMS、Miso)存在关键缺陷:
未针对基因扰动实验优化,无法有效关联 “基因变化 - 代谢中间体积累”;
非靶向代谢组学中全局代谢变化易掩盖通路特异性信号;
质谱数据中的加合物(adducts)、中性丢失(neutral losses)、源内碎片(ISF)易产生假阳性,干扰中间体筛选。
二、工具核心设计与实验框架
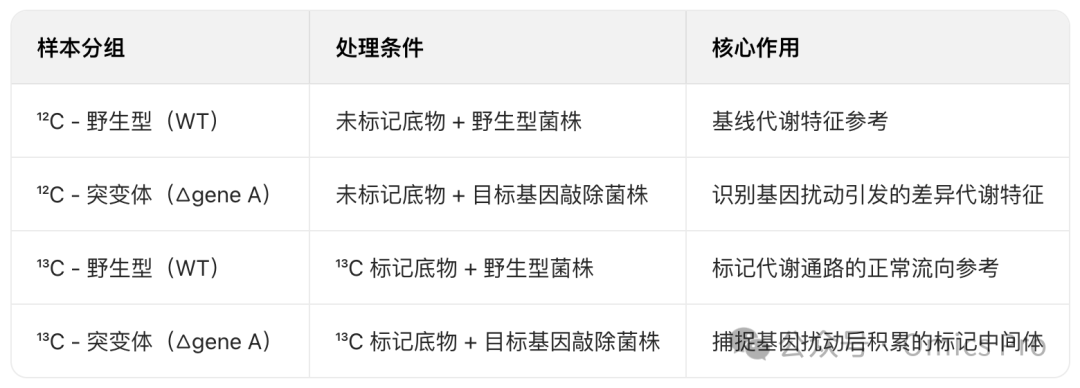
1. 实验设计原则
IsoPairFinder 需基于 4 组对照样本设计,确保精准捕捉基因扰动对代谢的影响,具体分组如下:
2. 核心工作流程(5 步)
IsoPairFinder 通过标准化流程实现 “从原始数据到通路中间体” 的精准筛选,关键参数与操作如下:
数据导入
必需输入:4 类文件(均支持 CSV/mzML/mzXML 格式)
-
特征表(来自 XCMS/MS-DIAL/MZmine 等峰提取工具,需轻微格式调整);
-
样本信息表(定义分组与标记类型);
-
原始 MS1 数据(用于离子合并);
-
原始 MS2 数据(用于 ISF 识别)。
差异分析
-
分析逻辑:分别对 “¹²C 组” 和 “¹³C 组” 进行 WT 与突变体的特征强度对比;
-
筛选标准:P 值 < 0.05(Student’s t 检验)且fold-change>10,结合 Benjamini-Hochberg FDR 校正;
-
目的:聚焦基因扰动直接影响的代谢特征,减少后续分析冗余。
合并冗余离子
-
处理对象:电喷雾电离产生的冗余离子(41 种加合物、58 种中性丢失、ISF);
-
操作逻辑:以 ¹²C 组差异特征为基准,在 ±3 秒保留时间窗口内匹配相关离子,通过m/z 公差 ±25 ppm(加合物 / 中性丢失)和MS/MS 光谱匹配(ISF)识别冗余;
-
效果:移除假阳性特征,提升后续配对准确性。
¹²C 与 ¹³C 特征配对
核心步骤:
① 基于精确质量和7 条黄金法则预测 ¹²C 特征的化学 formula(仅考虑 CHNOPS 生物常见元素);
② 计算对应 ¹³C 标记特征的理论 m/z;
③ 按m/z 公差 10 ppm和保留时间公差 3 秒匹配实测与理论特征;
目的:锁定基因扰动后积累的、具有同位素标记特征的通路中间体。
结果输出
核心输出:
-
特征对详情(m/z、保留时间、质量差、预测化学 formula、标记原子数);
-
差异分析报告(含冗余离子识别结果);
-
镜像提取离子色谱图(用于视觉验证特征对可靠性)。
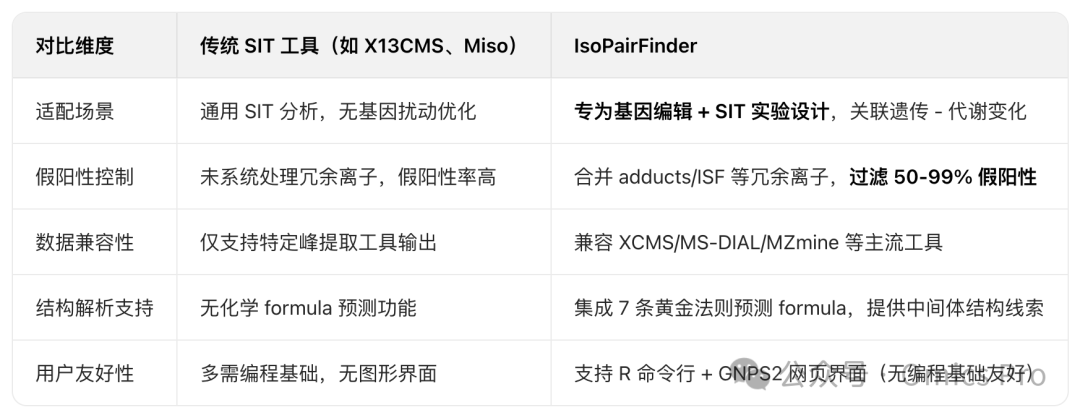
三、工具核心优势(与传统 SIT 工具对比)
IsoPairFinder 在功能设计上针对基因扰动实验做了专属优化,优势如下表所示:
四、案例研究:肠道细菌尿酸代谢通路发现
为验证工具有效性,研究团队以肠道细菌尿酸(UA)降解通路为对象开展实验,具体如下:
实验设置:
-
菌株:梭状芽孢杆菌(Clostridium sporogenes)的 7 个单基因突变株(CRISPR/ClosTron 构建);
-
底物:未标记 UA 和 [¹³C₅]- 标记 UA(培养 48 小时);
-
检测:HILIC 柱 LC-MS/MS(MS1+MS2),原始数据用 MS-DIAL 处理。
关键结果:
-
假阳性过滤:IsoPairFinder 将理论特征对从 725 个筛选至 4 个高置信候选(以 ygeW 突变株为例);
-
中间体鉴定:通过特征对(191.0747@5.417_196.0914@5.414)的质量差(提示 5 个 ¹³C 标记)和化学 formula(C₅H₁₀N₄O₄),确定中间体为 2,3 - 二脲基丙酸;
-
通路重建:最终通过化学标准品验证 4 个中间体结构,成功解析 UA→短链脂肪酸的代谢通路,解释了人类尿酸酶缺失后肠道菌的代偿机制。
五、可用性与资源支持

六、结论与未来展望
IsoPairFinder 填补了 “基因扰动 + SIT 代谢组学” 分析工具的空白,可广泛应用于微生物、哺乳动物细胞、模式生物等具备基因编辑工具的系统,助力新型代谢通路、天然产物的发现。未来将通过整合 SIRIUS 等 MS/MS 结构解析工具,进一步提升中间体注释的完整性;同时推荐优先使用 ¹³C 标记以减少保留时间偏移,优化分析精度。
关键问题
问题 1:IsoPairFinder 在设计上如何针对性解决传统 SIT 代谢组学工具不适配基因扰动实验的痛点?
传统 SIT 工具的核心痛点是 “无法关联基因扰动与代谢变化”“假阳性高”“兼容性差”,IsoPairFinder 通过3️⃣方面设计针对性解决:
-
专属实验框架:强制要求 4 组对照样本(¹²C-WT/¹²C - 突变体 /¹³C-WT/¹³C - 突变体),通过 “突变体 vs WT” 的差异分析直接捕捉基因扰动引发的代谢中间体积累,而非仅分析同位素流向;
-
假阳性精准控制:新增 “合并冗余离子” 步骤,系统识别并移除 41 种加合物、58 种中性丢失及 ISF,结合 “m/z 10 ppm+RT 3 秒” 的配对公差,可过滤 50-99% 的理论假阳性特征对;
-
遗传 - 代谢关联能力:通过 “差异分析(锁定基因相关特征)→同位素配对(确认通路中间体)” 的串联流程,直接建立 “基因敲除→中间体积累” 的因果关系,而传统工具仅能追踪同位素标记,无法关联基因变化。
问题 2:在 IsoPairFinder 的工作流程中,“差异分析” 和 “¹²C/¹³C 特征配对” 两个关键步骤的核心参数、操作逻辑及目的分别是什么?
两个步骤的核心细节如下:

问题 3:肠道细菌尿酸代谢的案例研究中,IsoPairFinder 的有效性通过哪些具体结果得到验证?这些结果对代谢通路研究有何价值?
有效性验证及价值如下:
-
假阳性过滤能力:以 ygeW 突变株为例,工具将理论上的 725 个特征对筛选至 4 个高置信候选,过滤效率达 99.4%,证明其能精准排除冗余信号;
-
中间体鉴定准确性:通过特征对(191.0747@5.417_196.0914@5.414)的质量差(提示 5 个 ¹³C 原子)和预测化学 formula(C₅H₁₀N₄O₄),成功鉴定出 2,3 - 二脲基丙酸,后续经化学标准品验证无误;
-
通路重建价值:最终验证 4 个关键中间体结构,完整解析 “尿酸→短链脂肪酸” 的代谢通路,解释了人类尿酸酶缺失后肠道菌的代偿机制,为微生物代谢通路的 “基因扰动→中间体筛选→通路验证” 提供了标准化流程,证明 IsoPairFinder 可加速未知代谢通路的发现效率。
参考
bioRxiv[Preprint]. 2025 Aug 22:2025.08.18.670916. doi: 10.1101/2025.08.18.670916
注:AI辅助翻译,如有错误欢迎指出。请以复制粘贴,附上本号名片的方式转载此文。
更多推荐
 已为社区贡献14条内容
已为社区贡献14条内容









所有评论(0)