Hallucination Is All You Need?从BindCraft谈谈蛋白“幻觉设计”
“ 我们希望AI蛋白模型能够学习到超越自然已解析蛋白质折叠规律的底层逻辑,并指导我们对尚未探索但具有生物合理性的蛋白质空间进行采样 ”
“ 我们希望AI蛋白模型能够学习到超越自然已解析蛋白质折叠规律的底层逻辑,并指导我们对尚未探索但具有生物合理性的蛋白质空间进行采样 ”
01
何为“幻觉设计”
Hallucination 设计是一种生成式蛋白质设计思路,通过将蛋白质结构预测模型反向使用来设计新序列。具体地,给定目标蛋白结构(或复合物),我们从随机序列开始,反复将序列输入结构预测模块(AlphaFold2/ RoseTTAFold),计算预测结构的置信度指标(如 pLDDT、ipTM),并对这些指标进行反向传播优化序列。这样序列和结构“Codesign”,逐步收敛到一个预测置信度高、符合设计目标的结合蛋白。
Hallucination的方法最初由Baker Lab提出,用于生成单体或对称多聚体结构(Science 2022 “对称体幻觉”设计),近年被推广到蛋白质结合物设计(binder)。与传统基于物理能量函数的设计方法不同,Hallucination设计无需预定义折叠骨架,而是利用训练好的深度模型学到的“能量景观”来探索序列空间。
“ 正如《Hallucinating symmetric protein assemblies》的第一句——Deep learning generative approaches provide an opportunity to broadly explore protein structure space beyond the sequences and structures of natural proteins, 我们希望AI蛋白模型能够学习到超越自然已解析蛋白质折叠规律的底层逻辑,并指导我们对尚未探索但具有生物合理性的蛋白质空间进行采样”
基于幻觉的设计背景源于深度学习结构预测的突破。AlphaFold2 和 RoseTTAFold 等模型具有极高的结构预测准确度,同时输出的 pLDDT、pAE 等置信度分布可以视为序列-结构匹配的代理目标。研究者发现,通过对这些置信度指标进行梯度优化,可“幻觉出”全新的蛋白质结构。Binder 设计方法以此为基础,将结构预测模块得到的指标组合为损失函数,通过反向传播直接生成与目标蛋白结合的蛋白质序列。例如,BindCraft 算法即利用 AlphaFold2模型的权重,将目标蛋白与随机序列共同输入网络,不断调整序列以最小化Loss。这种幻觉设计理念使得设计者能够“直接在神经网络里探索”结构空间,从而生成常规模板或对接方法难以覆盖的新型结合蛋白序列。
02
主流模型
在具体实现上,典型的幻觉式 binder 设计工具均采用预训练的深度结构预测网络作为评估和优化器。
ColabDesign 是 Sergey Ovchinnikov 实验室推出的一个开源pipeline合集,支持多种设计方法,其中核心模块AfDesign也是BindCraft设计的核心模块。ColabDesign 平台包含 P(structure|sequence) 的设计模块(如 TrRosetta-Design, AfDesign 等)以及 P(sequence|structure) 的模块(如 ProteinMPNN)。其中,AfDesign模块实现了基于 AlphaFold2 的 binder 幻觉设计功能:用户可自行或在 Google Colab 上提供目标结构和 binder 长度等设定,系统自动迭代优化序列以满足结构约束。其实现无需重新训练模型,基于Alpha-Multimer的权重进行梯度计算。
“ColabDesign作者Wang Jue为了使AlphaFold2支持反向传播做了不少interesting的操作,此处由于篇幅原因不过多介绍。由于AlphaFold2基于jax的架构,ColabDesign也采用jax,在代码阅读上对于习惯pytorch的读者有一定挑战性。如果感兴趣可以移步知乎Peldom学长写的三篇《AI+蛋白设计|AlphaFold2的梯度回传》,对于技术实现写得非常细节”
BindCraft(Pacesa et al, 2024)就是融合ColabDesign成功实现高质量设计的代表之一,声称设计成功率10%-100%。其流程如下:输入目标蛋白结构及一个随机初始化的 binder 序列,调用 AlphaFold-Multimer 预测复合物结构;输出 ipTM、pLDDT、pAE 等置信度值,加权求和作为损失函数并计算梯度;然后通过反向传播更新 binder 序列(one-hot 表示),优化 ipTM 和 pLDDT 等损失函数,以生成亲和力高的结合蛋白。BindCraft 不需任何实验数据或已知结合位点,完全依赖AlphaFold-Multimer的参数。同时结合了ProteinMPNN(结构到序列模型)等手段,在优化过程中提高序列的可合成性和多样性。整个管道自动化,一次“设计”流程即可产生上百个候选序列,然后通过实验验证。
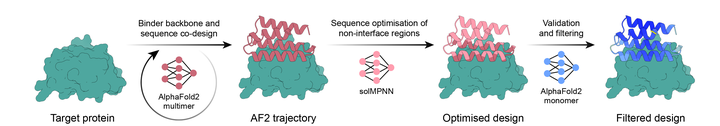
BindCraft flowchat
“BindCraft严格来说应该算一种混合设计模型(ColabDesign+ ProteinMPNN ),可以抽象理解为利用AlphaFold-Multimer反向传播对Binder interface部分进行结构序列的codesign,再用ProteinMPNN对Binder非interface的scaffold区域序列进行redesign。如果湿实验的高成功率(10%-100%)是真实的,说明interface和scaffold分开设计的hybrid design方法或许是Binder design一个不错的思路”
此外,BindEnergyCraft(2025 preprint)提出将 AF2 置信度解析为能量模型(pTMEnergy),仅仅修改了BindCraft的Loss function便显著提升了幻觉设计的性能,并在体外测试中超越了 BindCraft 和 RFdiffusion。 FoldCraft 引入额外约束(接触图相似度)进行聚合物条件设计,但只进行了基于AF3的insilico评估,有效性有待进一步评估。总体而言,这些方法大多共享模块化设计:以结构预测模型为核心,结合多种损失函数(预测准确度、界面亲和力等),通过梯度或采样等方式搜索序列空间。
除了以上基于AlphaFold2权重反向传播进行Binder design的工具,AlphaFold3的横空出世也让基于AlphaFold3进行设计的思路成为可能。BoltzDesign1(2025)构建于 Boltz-1( AlphaFold3开源实现)之上,扩展了设计目标范围,可设计结合小分子、核酸、金属和修饰蛋白的结合物。在无实验优化的条件下,使用 Boltz-1 结构预测进行幻觉优化,生成了对多种配体(包括药物分子、DNA、RNA、金属离子和带共价修饰的蛋白)有高预测亲和力的结合蛋白。虽然尚待实验验证,但 BoltzDesign1 在模拟测试中的成功率高达 90% 以上。
03
应用场景
抗体与纳米抗体设计是幻觉设计的重要场景之一。如Takafumi等研究中,通过 AfDesign 重新设计抗体的 CDR 环(补体决定区)序列,在无需结构先验的情况下优化抗原结合亲和力。该方法针对已知的抗原-抗体复合物,随机生成 CDR 序列并迭代优化,实现了替换后抗体结合能力的提升。

小肽与环状肽设计也是活跃领域。Kosugi 等(2022)利用 AfDesign 的 binder 幻觉,对抗体-蛋白相互作用界面设计小肽,并进一步引入溶解度损失控制肽链亲水性。他们的结果表明,通过调整损失函数权重,生成的肽序列保留高亲和力同时显著提高可溶性。另有工作利用幻觉设计环肽(cyclic peptide),靶向蛋白–蛋白相互作用界面,实现了高可溶性结合肽的定向生成。
过敏原、细胞受体等靶点的结合物设计是BindCraft文章内的湿实验成功案例。BindCraft 实验性验证了一系列不同目标,包括花粉过敏原(通过设计 binder 降低 IgE 结合)、Cas9(影响基因编辑活性)、细菌肠毒素(降低细胞毒性)等。此外,BindCraft 还开发了 AAV 载体重定向技术:通过设计特异性结合细胞表面受体的binder,将病毒载体定向至特定细胞类型。这说明结合物不仅可用作抑制剂,也能作为靶向递送元件。
04
主流方法对比
与对接(docking)或模板驱动(template-driven)的传统方法相比,幻觉式设计具有显著区别和优势。传统方法通常先筛选或准备一个蛋白骨架库(如基于模板库或随机蛋白库),然后使用分子对接算法(如 Rosetta Docking)来评估绑定模式及结合能。这需要大量的先验信息(如已知结合位点或类似结合物结构),且因为物理力场或打分函数的不完美,往往必须设计上千上万候选序列并进行筛选才能找到少数有效 binders。例如,Baker 实验室报道的 Rosetta 设计方法,对 13 个不同目标位点设计结合蛋白时,需要生成上千万个候选,最终只有极少数获得亚微摩尔亲和力。这种方法适应性较差:如果无法找到合适模板或库,就无法设计新结构;且对结合位点形状的匹配度要求很高。相较而言,幻觉法无需预设骨架模板,可直接在新序列空间中创造全新的折叠形状,使得设计更加灵活多样。
与传统序列为主(sequence-based)方法(如统计共进化模型、语言模型生成等)相比,幻觉方法显著不同。传统基于序列的方法主要学习序列统计规律,或根据给定骨架使用 ProteinMPNN 此类模型预测序列(P(sequence|structure)),但它们不涉及动态评估结构折叠。幻觉方法在设计过程中同时考虑序列和结构,通过多次结构预测确保生成的序列可稳定折叠且与目标界面匹配。例如,Binder Hallucination在每次迭代中预测复合体结构,并以结构置信度为损失进行优化。这使得幻觉设计在生成能力上更强:它能探索超出已知序列和结构范畴的新蛋白折叠样式;而传统序列方法往往受限于训练数据,不易跳出已知蛋白空间。
在结合亲和力预测和适应性方面,幻觉式方法与传统方法亦有差异。传统对接方法通常使用经验力场计算结合能,而幻觉式设计则依赖structure prediction model输出的置信度指标。尽管这些指标是近似的“预测结构质量”,但它们直接反映了网络对复合物结构可行性的信心。在适应性方面,幻觉设计能够轻松集成新损失项(如设计时加入可溶性优化),并可以针对不同靶点类型(例如蛋白、核酸、小分子)采用不同优化策略。传统方法往往需要为每种Target手工调整流程,而AI方法依托于模型的模块化,可更快速地迭代。
05
“幻觉设计”之困境
一个问题是设计精度与稳定性。当前方法往往依赖预测模型的置信度作为目标,但实际实验验证中仍有失配或结构不稳定的问题。例如,Wicky 等的对称体幻觉设计中,大量设计无法在实验中成功表达,表明“过拟合”风险,即网络优化可能生成在生物体系中不具备稳定性的序列。因此,提高生成序列的可表达性和可折叠性(如通过更多约束或额外损失)是未来工作重点。
另一个问题还是模型局限与泛化。AlphaFold2 等模型虽然强大,但依赖于蛋白质多序列比对(MSA)信息,对纯设计序列可能有所偏差。有研究使用单序列 AF2 或微调模型来减小依赖。另一个挑战是幻觉方法目前主要针对蛋白–蛋白界面,对于高度柔性靶点仍有难度。
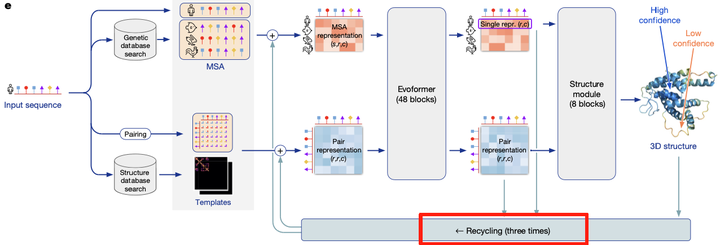
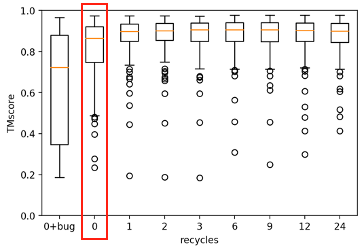
“为了使反向传播能够实现,AlphaFold2原架构中的3次recycles无法进行(相当于AlphaFold2的recycles=3 变为 0)对于结构预测的准确度会存在一定下降,可能会进一步影响设计质量”
未来来看,任何基于In silico的设计和优化最终还是要落回到湿实验验证。将幻觉设计与湿实验自动化相结合(如微流控高通量筛选、机器学习预测免疫原性等)可加速验证闭环优化。总之,随着更强结构预测模型和高效优化算法的出现,幻觉式 binder 设计有望逐步解决当前挑战,迈向更可靠的药物理性设计。
06
写在最后
在晶泰做AIDD实习生已经四个月啦,非常感谢师兄师姐们对我的照顾。在AIDD领域,Diffusion,LLM,GNN等一众大模型八仙过海,涉及架构之广,模型迭代之快,学习起来颇有蚍蜉撼树之感。创建这个公众号与大家一起分享探讨一下AI4Protein的新鲜事,也对自己阶段性的学习成果进行整理。才疏学浅,有不当之处恳请各位批评指正!也欢迎大家一起探讨AIDD的边界🥳
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容







所有评论(0)