MedCLIP-SAMv2:迈向通用文本驱动的医学图像分割|文献速递-深度学习人工智能医疗图像
Title
题目
MedCLIP-SAMv2: Towards universal text-driven medical imagesegmentation
MedCLIP-SAMv2:迈向通用文本驱动的医学图像分割
01
文献速递介绍
随着放射技术日益普及,对精准、高效的医学图像分割技术的需求不断增长,以支持各类疾病的研究、诊断与治疗(Siuly & Zhang, 2016)。深度学习(DL)技术已成为该领域的最先进(SOTA)方法;然而,这些技术面临三大关键挑战,阻碍了其在临床中的广泛应用。**首先**,大规模、标注完善的数据集稀缺,这是深度学习模型开发的主要障碍。**其次**,交互性与可解释性的缺失降低了人们对这些方法的信任度。**最后**,大多数医学深度学习模型是针对特定任务和成像对比度/模态训练的,灵活性受限。尽管已有多种自监督和弱监督方法(Baevski et al., 2023;Chen et al., 2020;Taleb et al., 2021)被提出以提高训练效率,且可解释人工智能(XAI)技术(包括不确定性估计(Loquercio et al., 2020;Liu et al., 2020)和显著图(Arun et al., 2021;Bae et al., 2020))也在积极研究中,但跨域泛化能力仍是一大挑战。尽管做出了这些努力,现有模型往往难以在不同模态/任务间实现泛化,且缺乏交互式临床部署所需的灵活性。要解决这些局限性,需要一个能够在零样本设置下运行、适配多种成像类型/任务并融入人工指导的框架。近年来,对比语言-图像预训练模型(CLIP)(Radford et al., 2021)、任意分割模型(SAM)(Kirillov et al., 2023)等基础模型的出现,为交互式、通用型医学图像分割开辟了道路。已有多个研究团队将CLIP和SAM适配于放射学任务,开发出BiomedCLIP(Zhang et al., 2023)和MedSAM(Ma et al., 2024)等模型,这些模型均在海量生物医学数据上进行了预训练。此类模型有望实现跨模态对齐与分割灵活性,但要充分释放其在临床成像任务中的潜力,仍需进一步适配与微调。具体而言,为减少SAM模型对专业人员精准绘制视觉提示(如点、边界框)的依赖,CLIP可提供一种替代机制——通过自然语言与用户交互来生成视觉提示,这种方式更灵活、直观且可扩展。尽管CLIP训练主要在全局层面实现图文映射,但相关研究(Fu et al., 2024)表明,这类模型能够编码丰富的图像特征表示。这使得我们能够建立全局文本信息与局部视觉特征之间的关联(Zhou et al., 2022;Rao et al., 2022),进而将其用于高效的零样本医学图像分割,即便在数据稀缺场景下也能实现更广泛的应用——这一点我们已在2024年国际医学图像计算与计算机辅助干预会议(MICCAI 2024)的论文(Koleilat et al., 2024b)中首次探索。然而,由于医学描述的复杂性和诊断成像特征的细微性,将CLIP在自然图像领域的成功迁移到放射学领域并非易事。尽管将CLIP适配到医学图像领域看似具有吸引力,但这一过程难度较大,且需要大量真值标签才能有效微调模型,尤其是针对分割这类下游任务(Poudel et al., 2024)。医学成像领域中大规模、高质量标注数据集的缺乏,进一步加剧了这一挑战。这促使研究人员采用BiomedCLIP(Zhang et al., 2023)等生物医学领域专用模型变体——这类模型更适合捕捉与疾病相关的放射学特征表示,同时也推动了高效微调损失函数的研发,以在病理定位、分割、诊断等放射学应用中实现更有效的跨模态学习。另一方面,随着对SAM关注度的提升,为减少其分割任务对视觉提示(如点和/或边界框)的依赖(这类提示需要专业临床知识),近年来出现了多种新方法:无需此类提示即可微调SAM(Chen et al., 2024;Hu et al., 2023)、通过分类任务的类激活图(CAM)生成提示(Li et al., 2025, 2023b;Liu & Huang, 2024)、利用弱监督优化其输出(Yang & Gong, 2024;Chen et al., 2023;Huang et al., 2023)。这些方法表明,研究人员对“构建融合视觉-语言理解与交互式分割的框架”的兴趣日益浓厚,并开辟了新的研究方向,摆脱了对人工绘制提示的依赖。近期,为解决上述挑战,我们在2024年MICCAI会议(Koleilat et al., 2024b)中提出了MedCLIP-SAM框架。该框架利用BiomedCLIP(Zhang et al., 2023)生成基于文本的边界框提示,为SAM(Kirillov et al., 2023)提供支持,以实现零样本和弱监督设置下的交互式、通用型医学图像分割。在初步取得成功后,有必要对该框架进行进一步改进与探索,以提升性能,并更深入地理解CLIP和SAM这两种基础模型在医学成像应用中的作用。因此,本文提出**MedCLIP-SAMv2**——这是一个显著增强的框架,在MedCLIP-SAM的基础上,通过优化CLIP与SAM的融合方式、基于显著图的提示生成以及含不确定性感知的弱监督,更好地发挥这些基础模型的协同作用,实现通用、可扩展的医学分割。具体而言,与原始方法相比,新提出的MedCLIP-SAMv2框架的主要升级包括:- 研究了适用于CLIP模型的多种显著图生成技术,用M2IB(Wang et al., 2024)替代了gScoreCAM(Chen et al., 2022);将M2IB与BiomedCLIP(Zhang et al., 2023)的微调相结合,显著提高了零样本分割精度。- 通过伪标签训练nnUNet(Isensee et al., 2021),同时借助检查点集成(Zhao et al., 2022)提供不确定性估计,从而改进了原有框架的弱监督分割结果与可解释性。- 扩展了验证范围,新增了肺部CT数据集,覆盖计算机断层扫描(CT)、磁共振成像(MRI)、超声、X射线四种关键放射学模态。这种全面的测试进一步证明了该框架在各类分割任务中的通用性与稳健性。- 利用大型语言模型(LLM)的推理能力和多种集成方法,研究并优化了先进的文本提示工程策略,结果表明这些策略能显著提升零样本分割性能。- 开展了更广泛的实验,以进一步验证框架设计组件的有效性,包括测试不同的SAM骨干网络和视觉提示类型。我们对框架中各组件的必要性进行了细致评估,并证明了每个组件对整体性能提升的单独贡献。所提出的MedCLIP-SAMv2框架结合了BiomedCLIP的跨模态对齐能力与SAM的灵活分割能力,解决了以往研究的根本性局限,推动放射学领域向实用、通用、交互式的分割工具迈进。新提出的MedCLIP-SAMv2框架精度更高,在实现通用文本驱动医学图像分割的道路上更进一步:零样本和弱监督范式下的Dice系数分别提升了13.07%和11.21%。本文的主要贡献包括三方面:**第一**,提出了一种新的CLIP训练/微调损失函数,即解耦硬负样本噪声对比估计(DHN-NCE)。**第二**,提出了一种基于文本驱动的零样本医学分割方法,将CLIP与SAM结合用于放射学任务。**第三**,探索了一种弱监督策略,通过不确定性估计进一步优化零样本分割结果。我们在四种不同的分割任务和模态(包括超声乳腺肿瘤分割、MRI脑肿瘤分割、胸部X射线肺部分割、CT肺部分割)上对所提框架进行了广泛验证。
Abatract
摘要
Segmentation of anatomical structures and pathologies in medical images is essential for modern diseasediagnosis, clinical research, and treatment planning. While significant advancements have been made in deeplearning-based segmentation techniques, many of these methods still suffer from limitations in data efficiency,generalizability, and interactivity. As a result, developing robust segmentation methods that require fewerlabeled datasets remains a critical challenge in medical image analysis. Recently, the introduction of foundationmodels like CLIP and Segment-Anything-Model (SAM), with robust cross-domain representations, has pavedthe way for interactive and universal image segmentation. However, further exploration of these models fordata-efficient segmentation in medical imaging is an active field of research. In this paper, we introduceMedCLIP-SAMv2, a novel framework that integrates the CLIP and SAM models to perform segmentation onclinical scans using text prompts, in both zero-shot and weakly supervised settings. Our approach includes finetuning the BiomedCLIP model with a new Decoupled Hard Negative Noise Contrastive Estimation (DHN-NCE)loss, and leveraging the Multi-modal Information Bottleneck (M2IB) to create visual prompts for generatingsegmentation masks with SAM in the zero-shot setting. We also investigate using zero-shot segmentation labelsin a weakly supervised paradigm to enhance segmentation quality further. Extensive validation across fourdiverse segmentation tasks and medical imaging modalities (breast tumor ultrasound, brain tumor MRI, lungX-ray, and lung CT) demonstrates the high accuracy of our proposed framework.
医学图像中解剖结构与病变的分割,对于现代疾病诊断、临床研究及治疗方案制定至关重要。尽管基于深度学习的分割技术已取得显著进展,但许多此类方法在数据效率、泛化能力和交互性方面仍存在局限。因此,开发所需标注数据集更少的稳健分割方法,仍是医学图像分析领域的核心挑战。 近年来,CLIP(对比语言-图像预训练模型)、SAM(任意分割模型)等基础模型应运而生,它们具备强大的跨域表征能力,为交互式、通用型图像分割开辟了新路径。然而,如何将这些模型应用于医学成像领域,实现数据高效的分割,仍是当前的活跃研究方向。 本文提出一种新型框架MedCLIP-SAMv2,该框架整合了CLIP与SAM模型,可在零样本和弱监督设置下,通过文本提示对临床影像进行分割。具体而言,我们的方法包括:采用全新的解耦硬负样本噪声对比估计(DHN-NCE)损失函数对BiomedCLIP模型进行微调;在零样本设置下,利用多模态信息瓶颈(M2IB)生成视觉提示,以辅助SAM生成分割掩码。此外,我们还探索在弱监督范式中使用零样本分割标签,进一步提升分割质量。 在四种不同分割任务及医学成像模态(乳腺肿瘤超声、脑肿瘤磁共振成像(MRI)、肺部X射线、肺部计算机断层扫描(CT))上开展的大量验证实验表明,所提框架具有较高的分割精度。
Method
方法
A full overview of the proposed MedCLIP-SAMv2 framework ispresented in Fig. 3, which is organized into three distinct stages: (1)BiomedCLIP fine-tuning employing our new DHN-NCE loss, (2) zeroshot segmentation guided by text-prompts, and (3) weakly supervisedsegmentation for potential label refinement. We additionally showcasea summary of the main components of the framework in Fig. 1 for thereaders’ easy reference.
所提出的MedCLIP-SAMv2框架的完整概览如图3所示,该框架分为三个不同阶段:(1)采用全新DHN-NCE损失函数对BiomedCLIP进行微调,(2)由文本提示引导的零样本分割,(3)用于潜在标签优化的弱监督分割。此外,为方便读者参考,我们在图1中还展示了该框架主要组件的汇总信息。
Conclusion
结论
We presented MedCLIP-SAMv2, an upgraded version of the originalMedCLIP-SAM framework, significantly improving segmentation performance with minimal supervision across CT, X-ray, Ultrasound, andMRI. By introducing the novel DHN-NCE loss for fine-tuning BiomedCLIP and leveraging SAM, our model achieved enhanced accuracy,particularly in complex tasks. MedCLIP-SAMv2 outperforms its predecessor through superior generalization and refined segmentation,demonstrating strong potential for clinical use in data-limited environments.
我们提出了MedCLIP-SAMv2,它是原始MedCLIP-SAM框架的升级版本。在计算机断层扫描(CT)、X射线、超声和磁共振成像(MRI)等模态下,该框架仅需极少监督即可显著提升分割性能。通过引入全新的DHN-NCE损失函数对BiomedCLIP进行微调,并结合SAM模型,我们的模型实现了精度提升,尤其在复杂任务中表现突出。相较于前代框架,MedCLIP-SAMv2具备更优的泛化能力和更精细的分割效果,在数据有限的环境中展现出强大的临床应用潜力。
Figure
图
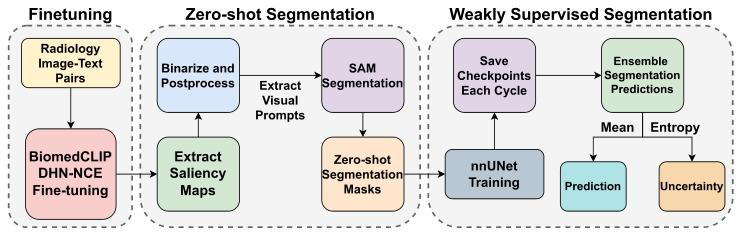
Fig. 1. A general overview of the essential components.
图 1 核心组件的整体概览
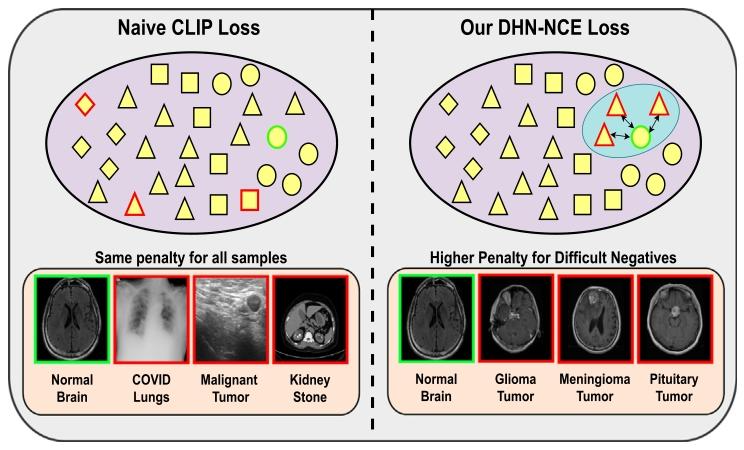
Fig. 2. Comparison of the standard CLIP loss, which applies uniform penaltiesto all examples regardless of difficulty, with our DHN-NCE loss, which prioritizesharder examples. The DHN-NCE loss enhances the differentiation of medical cases byappropriately penalizing close negatives through adaptive weighting formulas. Greenoutline represents the anchor example while the red outline represents the negativeexamples.
图 2 标准 CLIP 损失与 DHN-NCE 损失的对比
标准 CLIP 损失对所有样本施加统一惩罚,不区分样本难度;而本文提出的 DHN-NCE 损失(解耦硬负样本噪声对比估计损失)会优先关注难度更高的样本。通过自适应加权公式对 “相近负样本”(与锚点样本特征相似的负样本)施加合理惩罚,DHN-NCE 损失能够增强对医学案例的区分能力。图中绿色轮廓代表锚点样本,红色轮廓代表负样本。
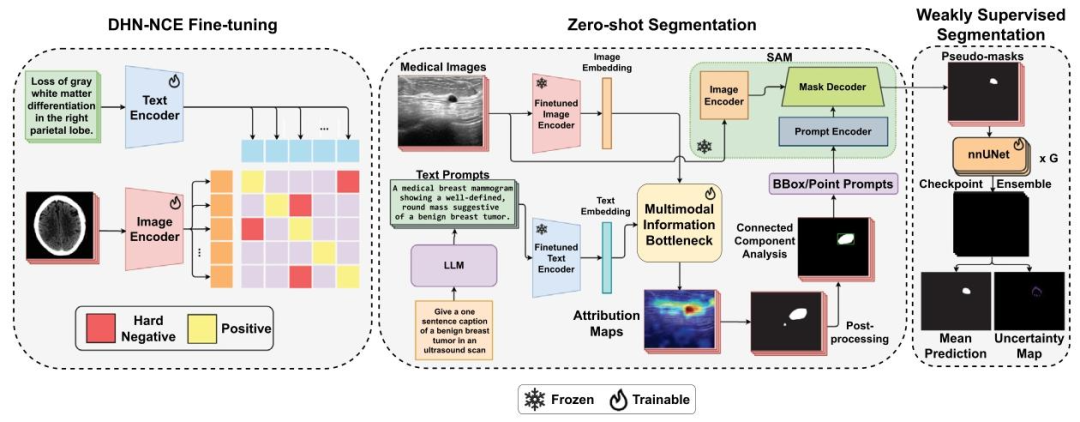
Fig. 3. An overview of the proposed MedCLIP-SAMv2 framework
图 3 所提 MedCLIP-SAMv2 框架的整体概览
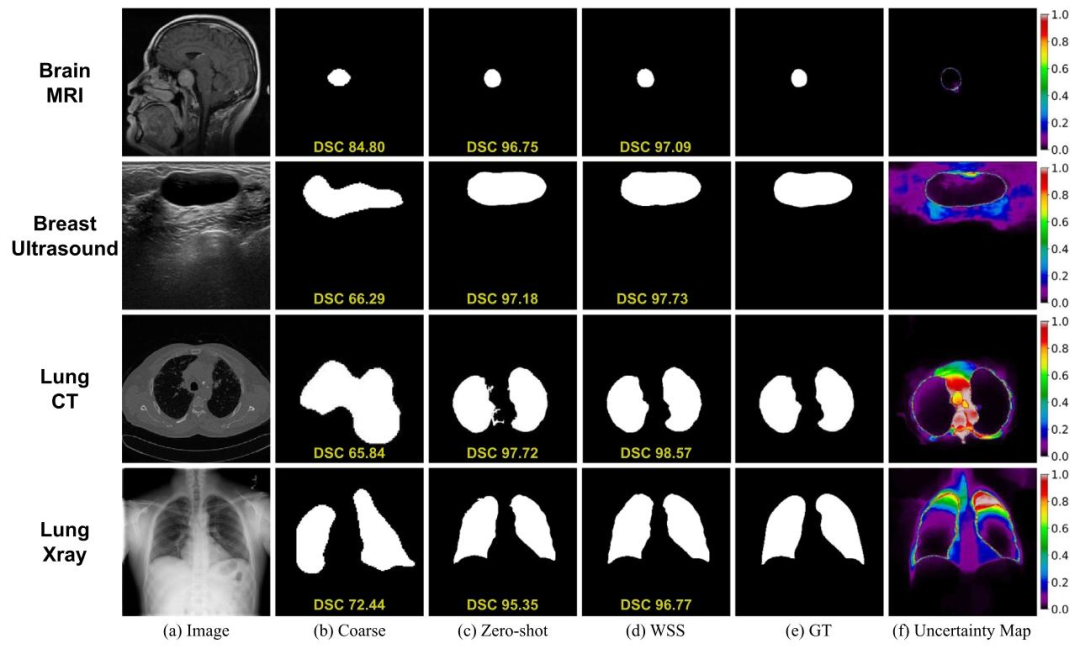
Fig. 4. Qualitative comparison of segmentation results. Coarse=post-processed saliency map, WSS=Weakly Supervised Segmentation and GT=Ground Truth. The uncertainty mapcorresponds to the weakly supervised segmentation
图 4 分割结果的定性对比其中,Coarse 代表经后处理的显著图,WSS 代表弱监督分割(结果),GT 代表真实标注(Ground Truth)。不确定性图对应弱监督分割(结果)。

Fig. 5. Diagram showing upsampled feature representations from the last transformerlayer of CLIP and BiomedCLIP. Feature Maps were upsampled using FeatUp (Fu et al.,for visualization purposes.
图 5 展示 CLIP 与 BiomedCLIP 模型最后一个 Transformer 层上采样特征表示的示意图为便于可视化,特征图采用 FeatUp 方法(Fu 等人,2024)进行上采样处理。
Table
表

Table 1Comparison of DSC and NSD values (%) with different few-shot and zero-shot medical image segmentation methods (mean𝑠𝑡𝑑 )
表 1 不同少样本与零样本医学图像分割方法的 DSC 和 NSD 数值对比(单位:%,均值 ± 标准差)

Table 2Top-K cross-modal retrieval accuracy (mean𝑠𝑡𝑑 ) for CLIP models.
表2 CLIP模型的Top-K跨模态检索准确率(均值±标准差)
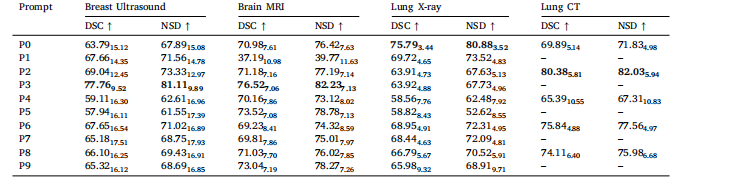
Table 3Effect of different text prompt templates on the segmentation performance (%, mean𝑠𝑡𝑑 )
表3 不同文本提示模板对分割性能的影响(单位:%,均值±标准差)
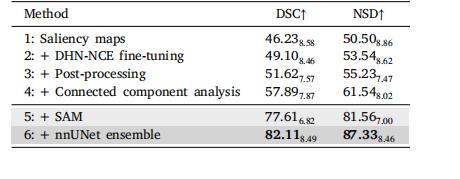
Table 4Effect of different components (%, mean𝑠𝑡𝑑 )
表4 不同组件的影响(单位:%,均值±标准差)
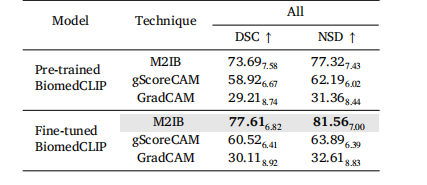
Table 5Comparison between different saliency map techniques aswell as the pre-trained and fine-tuned BiomedCLIP on theoverall performance (%, mean𝑠𝑡𝑑 )
表5 不同显著图技术以及预训练与微调后的BiomedCLIP模型在整体性能上的对比(单位:%,均值±标准差)
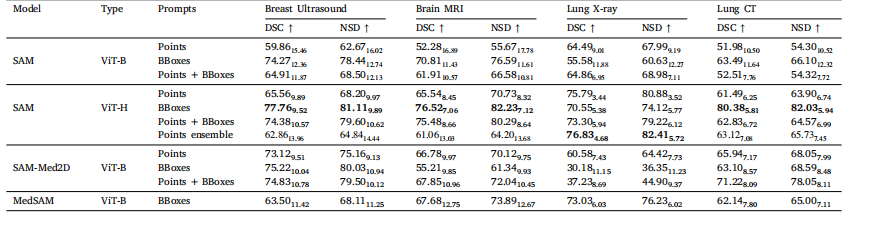
Table 6Comparison between different SAM pre-trained models and visual prompting techniques (%, mean𝑠𝑡𝑑 ).
表6 不同SAM预训练模型与视觉提示技术的对比(单位:%,均值±标准差)
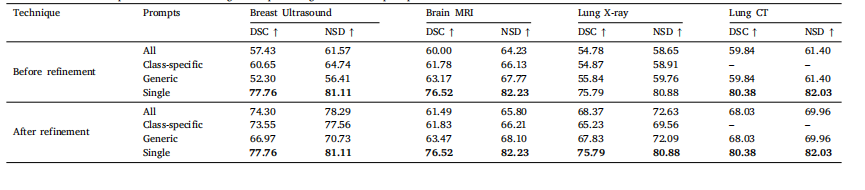
Table 7Zero-shot evaluation of pseudo-mask ensembling techniques using different text prompts.
表7 采用不同文本提示的伪掩码集成技术零样本评估结果

Table 8Comparison between different nnUNet ensembling techniques on weaklysupervised segmentation.
表8 不同nnUNet集成技术在弱监督分割上的对比
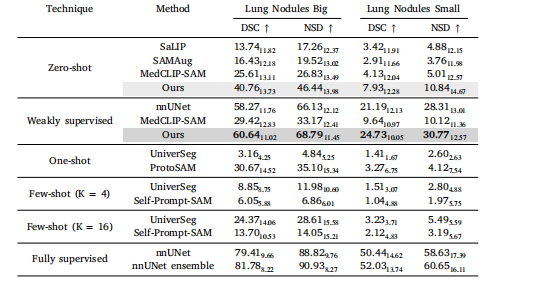
Table 9Comparison of DSC and NSD values (%) on Small and Big Lung Nodules (mean𝑠𝑡𝑑 ).
表9 小型与大型肺结节的DSC和NSD数值对比(单位:%,均值±标准差)
更多推荐
 已为社区贡献19条内容
已为社区贡献19条内容






所有评论(0)