组学数据自动化工具AutoXAI4Omics
机器学习(ML)方法为深入了解复杂生物系统的精妙运作提供了机会,在组学数据分析中的应用日益突出,有助于完成诸如新型生物标志物的识别和表型的预测建模等任务。对于科学家和领域专家而言,利用用户友好的ML流程极具价值,能在无需深入编程或算法优化专业知识的情况下运行复杂、稳健且可解释的模型。简化模型开发和训练的过程,研究人员可将时间和精力集中在生物解释和验证的关键任务上,从而最大化ML驱动洞察的科学影响力
摘要
机器学习(ML)方法为深入了解复杂生物系统的精妙运作提供了机会,在组学数据分析中的应用日益突出,有助于完成诸如新型生物标志物的识别和表型的预测建模等任务。对于科学家和领域专家而言,利用用户友好的ML流程极具价值,能在无需深入编程或算法优化专业知识的情况下运行复杂、稳健且可解释的模型。简化模型开发和训练的过程,研究人员可将时间和精力集中在生物解释和验证的关键任务上,从而最大化ML驱动洞察的科学影响力。介绍了完全自动化的开源可解释人工智能工具AutoXAI4Omics,能从组学和表格数值数据中执行分类和回归任务。AutoXAI4Omics通过自动化AI专家所做的过程和决策(如选择最佳特征集、不同ML算法的超参数调优以及为特定任务和数据集选择最佳ML模型)来加速科学研究。在ML分析前,AutoXAI4Omics还包含了针对特定组学数据类型定制的特征筛选选项。该工具通过可解释性分析提供的预测见解突出了组学特征值与研究目标(如预测的表型)之间的关联,有助于发现新的可操作洞见。
https://github.com/IBM/AutoXAI4Omics
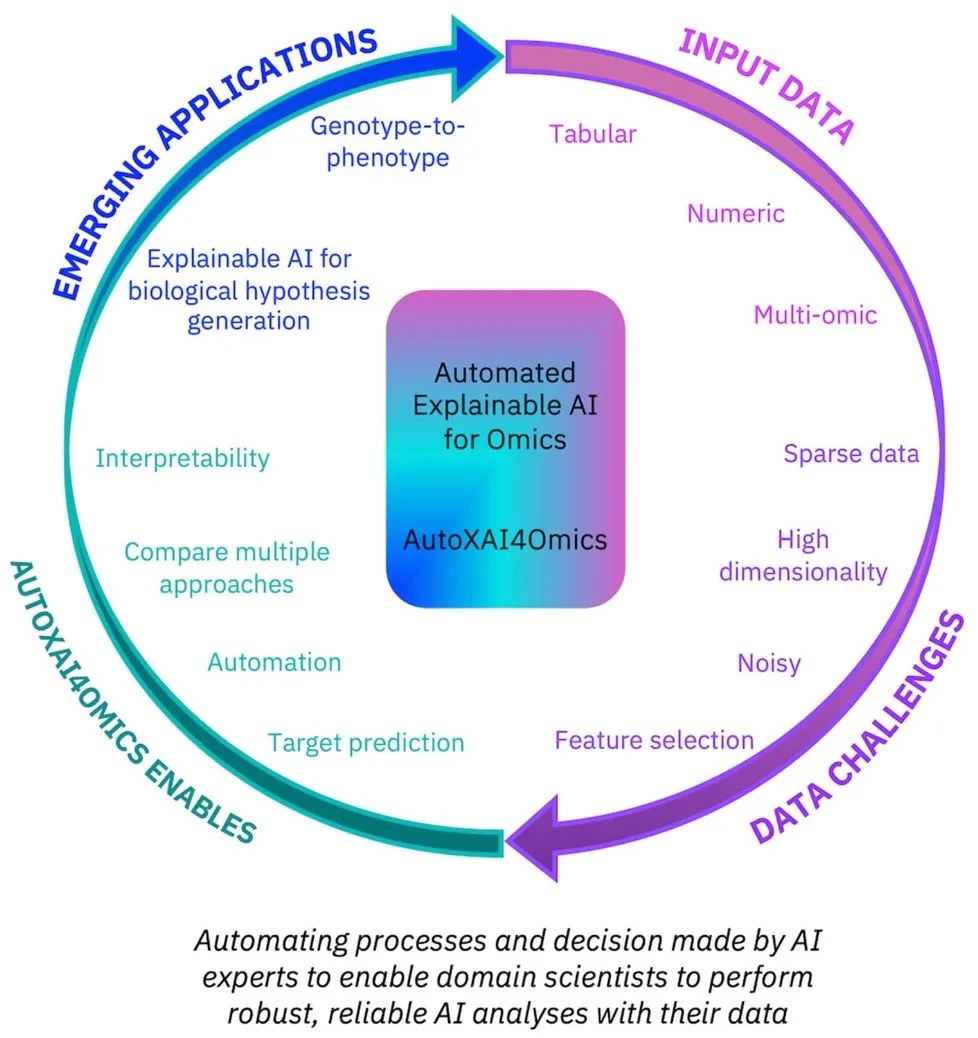
引言
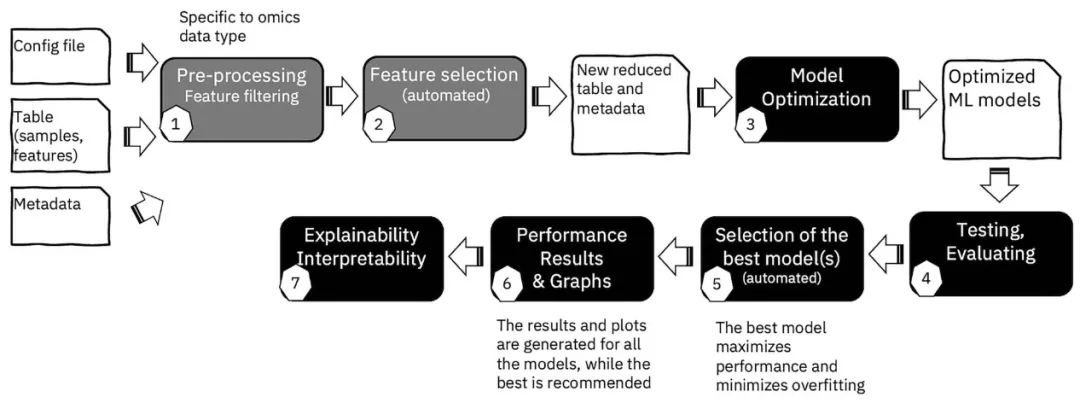
图1 AutoXAI4Omics XAI工作流程概述,从数据输入到结果和解释
结果和讨论
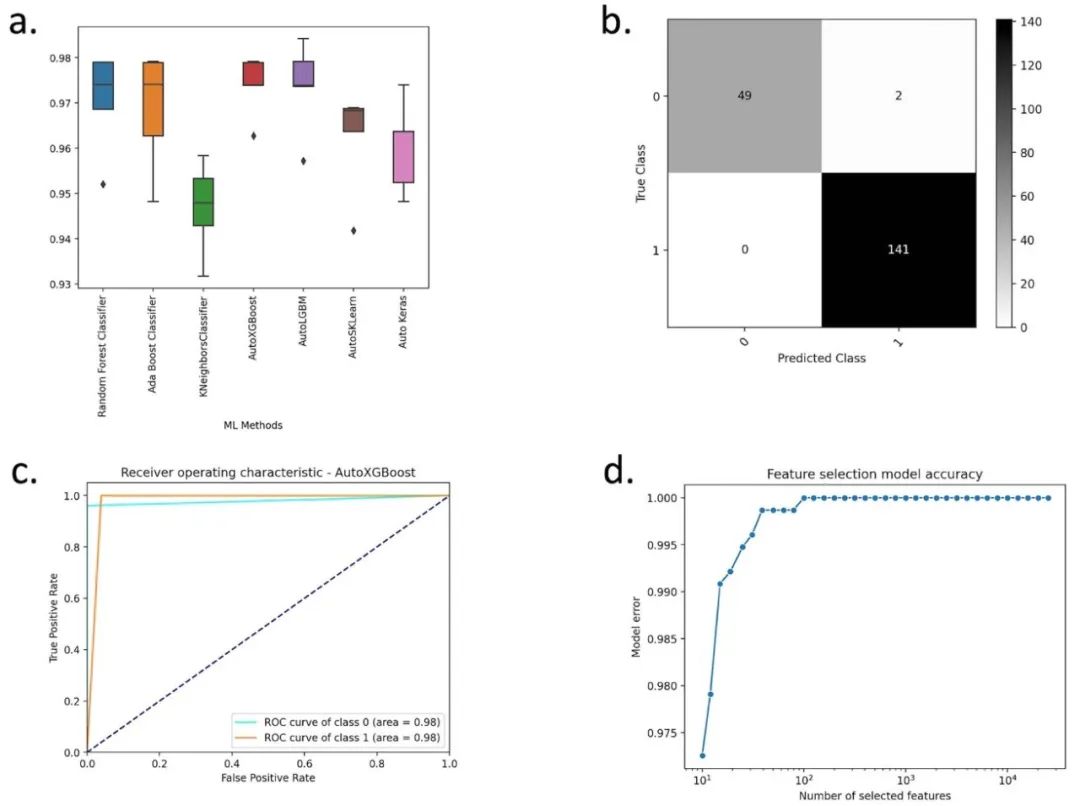
图2 AutoXAI4Omics二分类:植物基因组学的案例研究。总结了AutoXAI4Omics在预测2行(0)或6行大麦(1)时的预测性能。(a) 显示交叉验证过程中F1分数的箱线图,(b) 表现最佳的机器学习模型(XGBoost)的混淆矩阵,(c) 表现最佳模型的ROC曲线,以及(d) 特征选择准确率曲线。
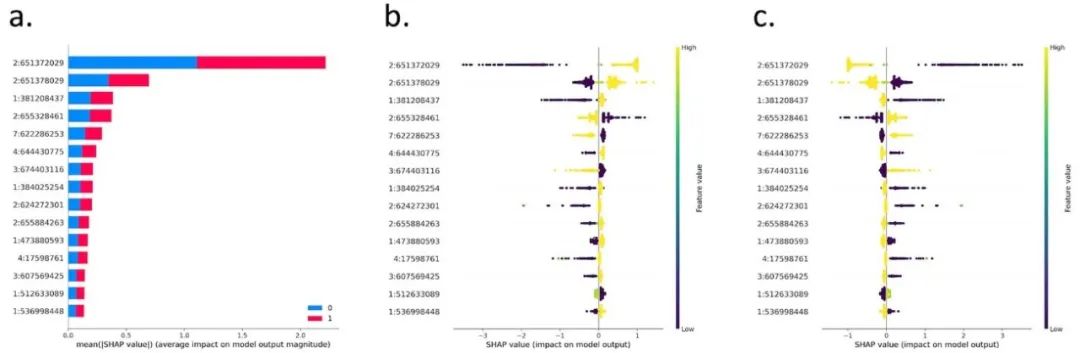
图3 AutoXAI4Omics二分类:植物基因组学的案例研究。总结了AutoXAI4Omics (XGBoost)在预测2行(0)或6行大麦(1)时表现最佳模型的可解释人工智能(XAI)输出。(a) 条形图总结了为最佳模型(XGBoost)选择的前15个特征值,(b) 6行以及(c) 2行大麦预测的SHAP全局解释视图。
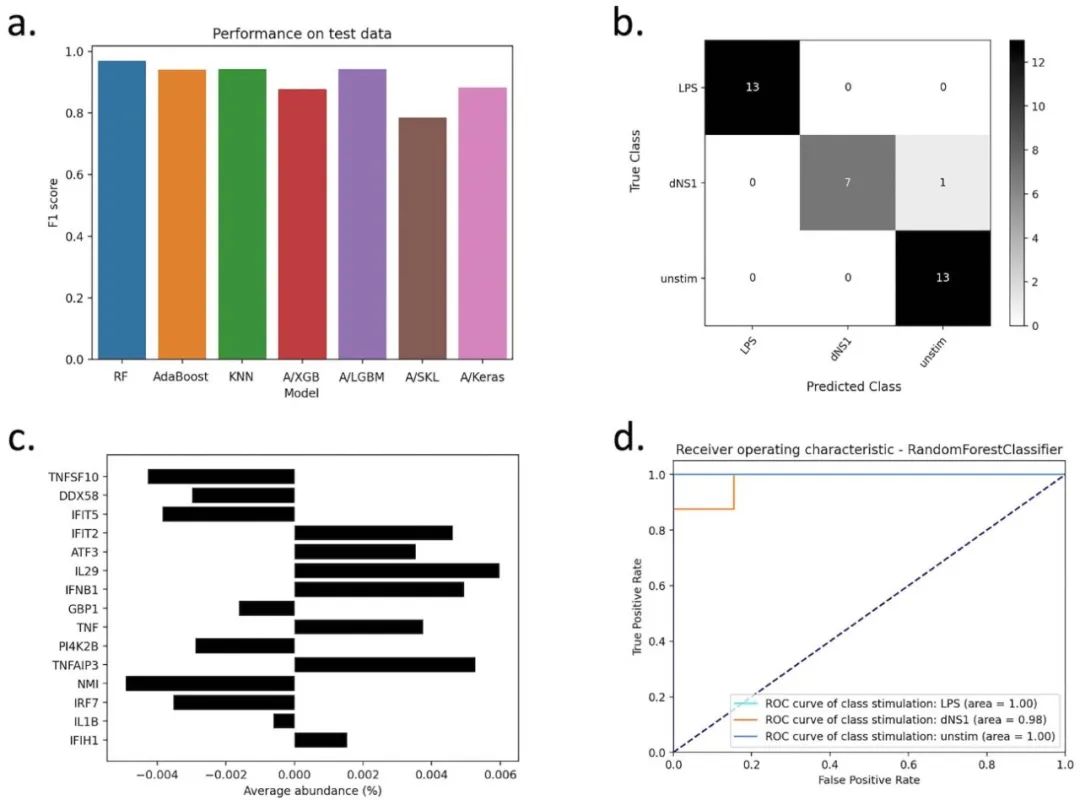
图4 AutoXAI4Omics多分类:人类RNA-seq数据的案例研究。(a) 条形图显示了在保留测试数据集上的F1分数,(b) 表现最佳的机器学习模型(随机森林)的混淆矩阵,(c) 条形图显示了特征重要性分析中的特征排名及其平均丰度,以及(d) 表现最佳的机器学习模型的ROC曲线。
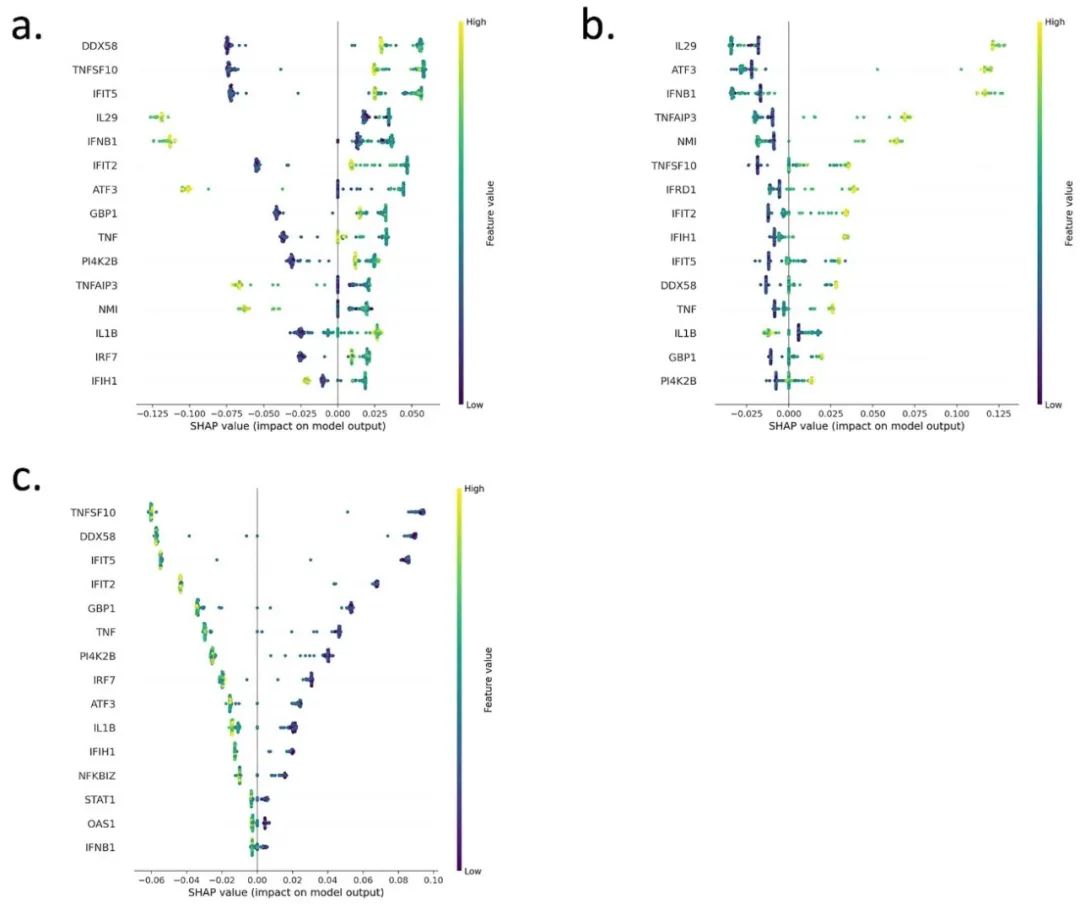
图5 AutoXAI4Omics多分类:人类RNA-seq数据的案例研究。与最佳机器学习模型(随机森林)相关的可解释人工智能(XAI)输出,用于预测3类:未刺激(unstim)、LPS和dNS1。(a) LPS,(b) dNS1,以及(c) 未刺激(unstim)类的全局解释。
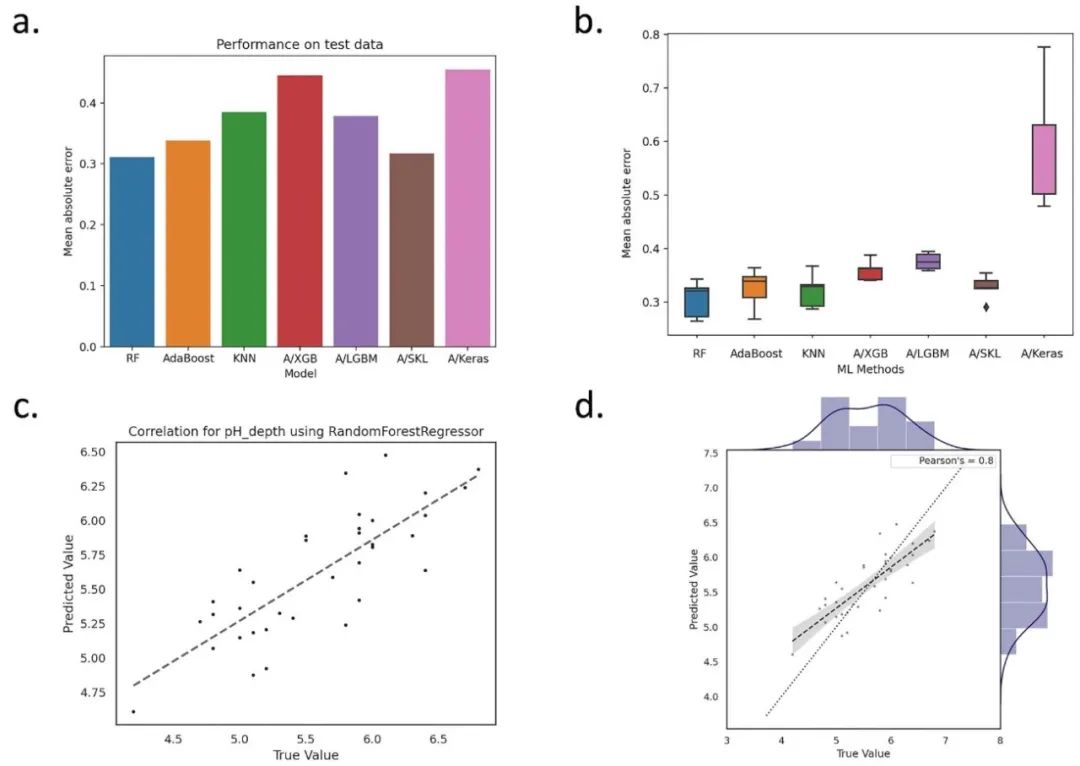
图6 AutoXAI4Omics回归:环境微生物组数据的案例研究。AutoXAI4Omics预测土壤pH值(基于土壤微生物组)的预测性能。(a) 条形图显示了在多种机器学习模型下保留测试数据集上的平均绝对误差(MAE),(b) 箱线图显示了在多种模型下交叉验证过程中的MAE,(c) 相关性,以及(d) 联合图,用于比较表现最佳的机器学习模型(随机森林)的预测值(y轴)与真实值(x轴)。
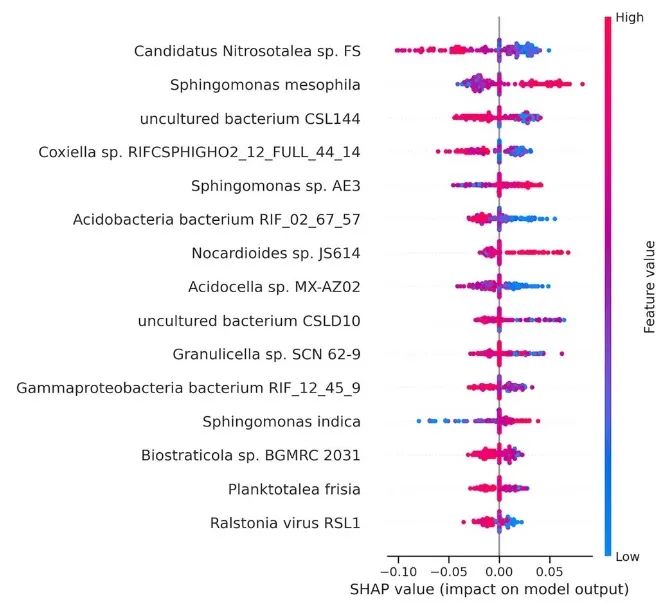
图7 AutoXAI4Omics回归:人类RNA-seq数据的案例研究。总结了与AutoXAI4Omics生成的最佳机器学习模型(随机森林)相关的SHAP可解释人工智能(XAI)输出,用于预测土壤pH值。
参考
[1] Brief Bioinform. 2024 Nov 22;26(1):bbae593. doi: 10.1093/bib/bbae593
注:AI辅助翻译,如有错误欢迎指出。请以复制粘贴,附上本公众号名片的方式转载此文。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)