天线阵列遗传算法赋形优化Python实现
本应用主要使用了两方面知识点,一方面是天线阵列合成公式;另一方面是遗传算法。通过遗传算法的数值优化,优化幅度/相位值,以达到优化方向图指标的作用。
关于原理的介绍
本应用主要使用了两方面知识点,一方面是天线阵列合成公式;另一方面是遗传算法。通过遗传算法的数值优化,优化幅度/相位值,以达到优化方向图指标的作用。
1. 天线阵列方向图合成
本公式被简化为了二维的合成,因为只要垂直面数据即可,适用于平面阵。
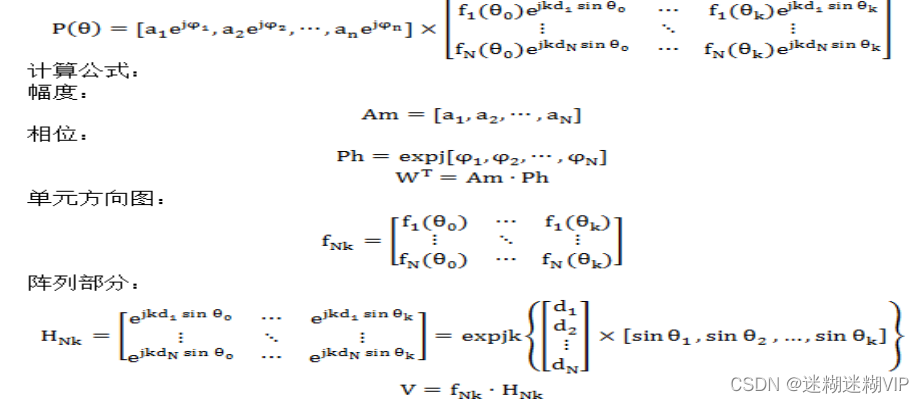
Am:幅度列表,Ph:x相位列表,f:为单元方向图组成的矩阵,H:阵列间距等位置信息
Ps:这个不理解的可以自己去相关书籍去查找,网上资料较少。
2. 遗传算法
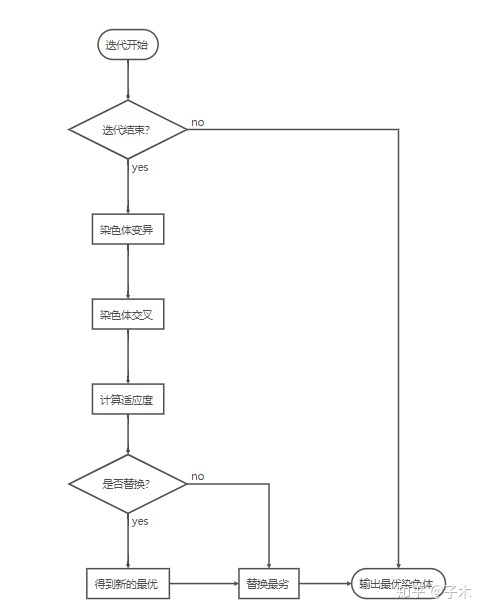
遗传算法的具体原理就不赘述了,具体的可以百度,网上很多相关资料,这个算法适合于多目标和多变量的优化求解。
本文这里主要涉及到的交叉知识点如下:
- 基因:这里的基因就相当于是变量,我们需要的幅度/相位就是算法里面的基因了。不同的基因组成一个变量组,也就是一个染色体。也就是多个变量的一个组合就是染色体。
- 交叉和变异:意思是给一个概率,相当于自然进化的一个选择,每两组染色体(变量值)进行较高概率的交叉,和较低概率的变异。这个概率值是可以调节的,以此来控制求解速度和范围。
- 适应度:这个概念是最重要的一个函数,相当于AI里面的评价函数,每个染色体(一组解)会产生一个基因表达,也就是一个结果,将多个目标值融合进去进行评价,评分高的保留,不高的就剔除。可以一直循环,一直查看适应度评分,如果达到要求,即求解完成(当然也可以继续优化更高得分,不过一般较难)。
这个算法的效果主要需要调整的设置值是迭代次数,变异概率,交叉设置,适应度评价函数调整。
Python代码开发
话不多说,上代码,主要分2部分,一部分是合成公式代码,另一部分是遗传函数的组合
预设变量
代码如下:
# coding=utf-8
import numpy as np
import matplotlib.pyplot as plt
import csv
Generations = 100 # 遗传代数
POP_size = 5000 # 群体规模
CROSS_rate = 0.8 # 交叉概率
MUTATION_rate = 0.1 # 变异概率
f_pre = 16 # 目标第一上旁瓣
f_pre_weight = 0.5 # 所占权重
db_pre = 14.5 # 目标最大增益
db_pre_weight = 0.5 # 所占权重
xiaqing = 2 # 中心下倾角
xiaqing_min = 2
xiaqing_max = 12
xiaqing_range = np.arange(xiaqing_min, xiaqing_max + 1, 1, dtype=np.int32) # 下倾角取值范围
Change = 1.5 # 下倾角精度
Ph_min = -180 # 相位取值范围
Ph_max = 180
Am = [0.6, 0.8, 1, 0.8, 0.6] # 幅度
Am_sum = sum(Am)
for i in range(len(Am)):
Am[i] = np.sqrt(Am[i] / Am_sum)
Freq = 1.71 * (10 ** 9) # 频点
d = 0.121
Disl = [0, d, 2 * d, 3 * d, 4 * d] # 间距列表
如图所示,备注里面每个都写明了改参数的解释。这里测试的是一个5个阵子组成的一个方向图。
天线合成公式
具体就是把上面的公式改写成代码,并且尽可能用计算较快的方式。代码如下:
def CalEleMat(k_list, Freq, Disl, f): # 这个函数是求解定值部分,不含幅度和相位,相当于求解公式中的V
# 并且去掉了for循环,改用了效率更高的矩阵运算
N = len(Disl) # N是单元数,Disl是关于d的列表
Disl = np.array(Disl)
Disl = Disl.reshape(Disl.shape[0], 1) # 矩阵行传列
SpeedOfLight = 2.0943951023931954923084289221863e-08 # 2π/c的值,即换算k=SpeedOfLight*Freq
AngleToRadian = 0.01745329251994329576923690768489 # π/180的值
ThetaVec = np.array(k_list) # 把角度列表变成一个一维数组
ThetaSinVec = np.sin(ThetaVec * AngleToRadian) # 1维的,sink值组成的数组
CellPatAm = np.power(10, f / 20) # db转成功率
EleMat = CellPatAm * (np.exp(-1j * SpeedOfLight * ThetaSinVec * Disl * Freq)) # DisL是阵子间距的d列表,需要做行列变换,变成n行单列的数组
return EleMat
def Cal(Am, Ph, EleMat): # Am:幅度,Ph:相位,k_list:Theta角列表,Freq:频点,Disl:间距d列表,f:原始方向图矩阵
Am = np.array(Am, dtype=np.float64)
Ph = np.array(Ph, dtype=np.float64)
AngleToRadian = 0.01745329251994329576923690768489 # π/180的值
W = Am * (np.exp(-1j * Ph * AngleToRadian))
PatData = np.dot(W, EleMat)
PatData = 20 * (np.log10(abs(PatData)))
return PatData
这个公式经过验证,是可以执行的,合成的方向图与HFSS仿真结果是吻合的。
遗传算法代码
分为适应度函数,自然选择的2个函数,染色体交叉的函数,以及变异的函数,具体的代码都有注释。
def F(k_list, values): # 分别求出最大增益和第一上旁瓣的数值
db_max = max(values)
k_list = list(k_list)
values = list(values)
x_max = k_list[values.index(db_max)]
# print('最大值坐标:('+str(x_max)+','+ str(db_max)+')') # 最大值的坐标
x2 = k_list.index(x_max)
x1_list = []
for i in range(x2):
if x_max - 31 < k_list[i] < x_max - 29:
x1_list.append(i)
x1 = min(x1_list)
F = []
for i in range(x1, x2):
if values[i] - values[i + 1] > 0 and values[i] - values[i - 1] > 0:
F.append(values[i])
else:
F.append(values[k_list.index(x_max - 30)])
f_ = db_max - max(F) # 第一上旁瓣
return db_max, f_, x_max
def get_fitness(db_max, f_, db_pre, f_pre, db_weight, f_pre_weight, x_max, xiaqing): # 根据增益和第一上旁瓣得出适应度函数值,单位一样,同样看待
fitness_db = np.fabs(db_max - db_pre)
fitness_f = np.fabs(f_ - f_pre)
fitness = db_weight * (1 / fitness_db) + f_pre_weight * (1 / fitness_f)
if np.fabs(x_max - xiaqing) >= Change or x_max < 0: # 下倾角不满足条件的适应度值就为0
return 0.0000001
elif db_max > db_pre or f_ - f_pre > 0:
return fitness ** 2 * 10000
else:
return fitness ** 2 * 1000
def select1(pop, fitness): # 根据适应度函数值选择个体,越高的几率越高
sum = np.sum(fitness)
idx = np.random.choice(np.arange(POP_size), size=POP_size, replace=True, p=fitness / sum)
return pop[idx]
def select2(pop): # 保留一半的原始群体,再随机生成另一半,合并成新的群体
pop_former = np.vsplit(pop, 2)[0]
pop_later = np.random.uniform(Ph_min, Ph_max, size=(int(POP_size / 2), DNA_size))
pop_new = np.vstack((pop_former, pop_later))
return pop_new
def crossover(parent, pop_copy): # 个体随机按概率在某个值上进行替换(两个个体之间进行)
if np.random.rand() < CROSS_rate:
i = np.random.randint(POP_size, size=1)
cross_point = np.random.randint(DNA_size, size=1)
# pop_copy=np.random.uniform(Ph_min, Ph_max, size=(POP_size, DNA_size))
parent[cross_point] = pop_copy[i, cross_point]
return parent
def mutation(child): # 个体随机按概率在某个值上进行变异,变异的范围(Ph_min, Ph_max),保证个体的可变性
for point in range(DNA_size):
if np.random.rand() < MUTATION_rate:
child[point] = np.random.uniform(Ph_min, Ph_max, size=1)
return child
主程序
先是读取方向图数据的矩阵,然后进行迭代技术,把符合要求的记录下来
#读取单元方向图数据
def read_csv():
datas = []
for i in range(1, 6):
name = 'h' + str(i) + '.csv'
data = []
with open(name) as csvfile:
csv_reader = csv.reader(csvfile)
header = next(csv_reader)
for row in csv_reader:
data.append(row)
data = [[float(x) for x in row] for row in data]
data = np.array(data, dtype=np.float64)
k_list = data[:, 0]
values = data[:, 1]
datas.append(values)
f = np.array(datas, dtype=np.float64) # 获取单元方向图矩阵
return k_list,f
if __name__ == '__main__':
k_list, f=read_csv()
N = len(Disl)
DNA_size = len(AntennaArrayTable(N))
pop = np.random.uniform(Ph_min, Ph_max, size=(POP_size, DNA_size)) # 就相当于相位个体,(-10,10相当于相位的取值范围)
x_list = []
y_list = []
ph_list = []
EleMat = CalEleMat(k_list, Freq, Disl, f)
for _ in range(Generations):
fitness = []
for i in range(POP_size):
Ph = pop[i]
Ph = Trans_Phase(Ph, N)
patdata = Cal(Am, Ph, EleMat)
db_max, f_, x_max = F(k_list, patdata)
fitness_eve = get_fitness(db_max, f_, db_pre, f_pre, db_pre_weight, f_pre_weight, x_max, xiaqing)
fitness.append(fitness_eve)
if db_max - db_pre > 0 and f_ - f_pre > 0 and np.fabs(x_max - xiaqing) < Change:
ph_list.append(pop[i].tolist())
print('一个合适的已经加入!!!')
pop = select1(pop, fitness)
pop = select2(pop)
x_list.append(_)
y_list.append(np.mean(fitness))
pop_copy = pop.copy()
for parent in pop:
child = crossover(parent, pop_copy)
child = mutation(child)
parent[:] = child
print('第%s代已经计算完成!!' % str(_ + 1))
plt.plot(x_list, y_list)
plt.title("迭代曲线",fontname="SimHei")
plt.xlabel("遗传代数",fontname="SimHei")
plt.ylabel("适应度值",fontname="SimHei")
plt.grid()
plt.show()
def qu(list): # 列表去重函数
list1 = []
for i in list:
if i not in list1:
list1.append(i)
return list1
ph_list = qu(ph_list)
print('共有%d组值满足条件!' % len(ph_list))
#随机取一个数值,看看效果如果
rand_ph=ph_list[1]
for i in range(len(ph_list)):
Ph = Trans_Phase(ph_list[i], N)
patdata = Cal(Am, Ph, EleMat)
db_max, f_, x_max = F(k_list, patdata)
if db_max>db_pre and f_>db_pre:
print(db_max,f_)
plt.plot(patdata)
plt.grid()
plt.show()至此,代码结束,我们看看运行的效果如何。
代码测试效果评估
条件1:增益>14.8,旁瓣>16,且中心下倾角在2附近。迭代100代。
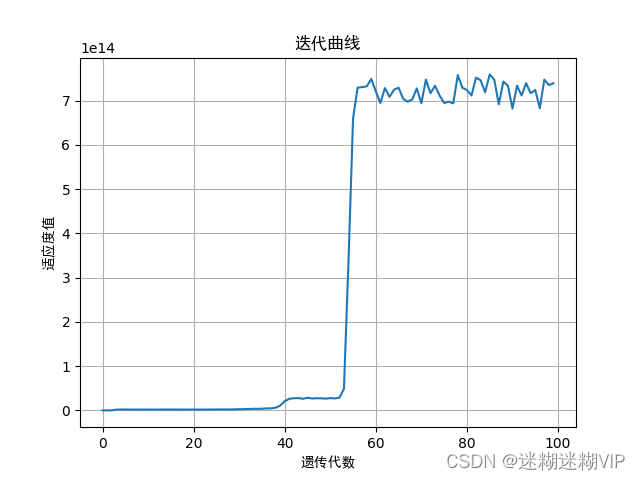
可以看到,遗传到50多代时,就出现了较好的值,一般来说,适应度值越高,效果也越好。统计出满足条件的相位值有1037组,大致的值和图形汇总如下:
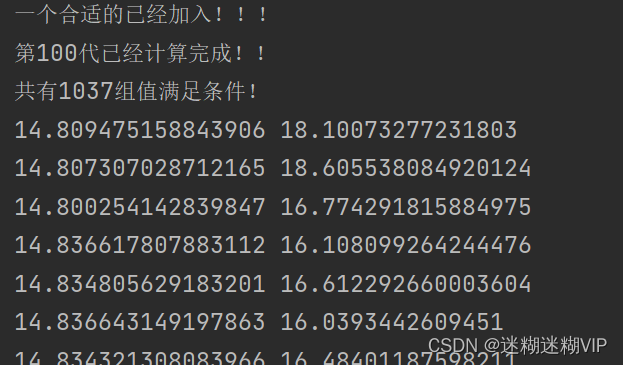
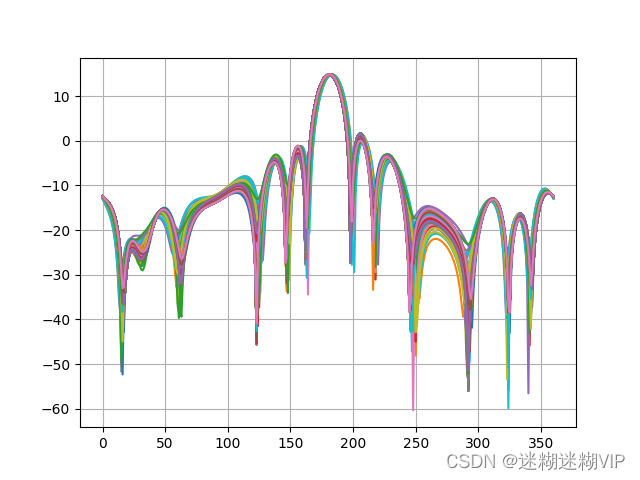
看吧,方向图还是很漂亮和一致的。
条件2:增益>14.83,旁瓣>17,且中心下倾角在2附近。迭代200代。
这时候的结果如下:
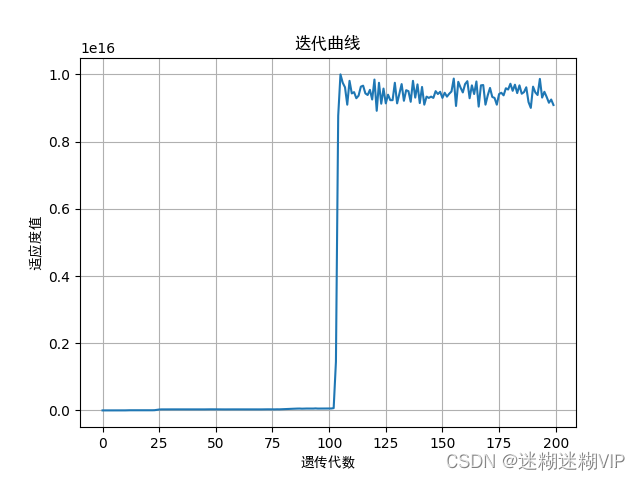
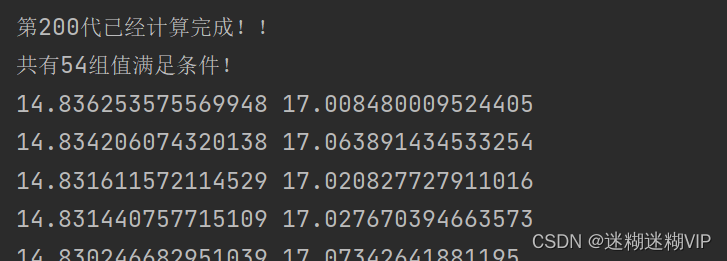
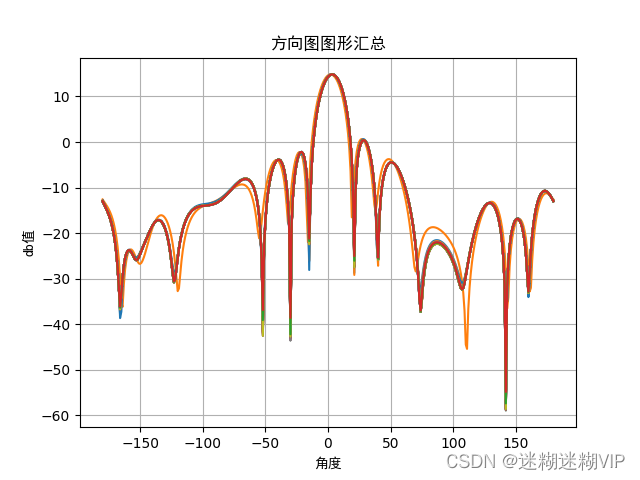
这时候我们发现,满足的组数明显减少,适应度值也下降了。不过依旧获取了有效的解。(这组值在验证时候,100次迭代有时候是无解的)
条件2:增益>14.84,旁瓣>17,且中心下倾角在2附近。迭代200代。
此条件下,我们跑了多次,都无解,那就说明了,我们给定的条件下,几乎无法优化出最优解。
总结:
通过此程序,我们验证了遗传算法在阵列赋形中的可行性,在此基础上,通过方向图图形逆推幅度相位,或者是优化幅度,间距等都可以尝试一下,也可以导入S2P文件,更真实模拟数值,开发更好的方案预估工具。
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容






所有评论(0)