R语言pheatmap包绘制热图全解析:从入门到高级定制(附完整代码)
本文详细介绍了如何使用R语言的pheatmap包绘制热图,涵盖从环境准备到进阶功能的全流程。首先,文章指导如何安装和加载pheatmap包,并生成模拟数据进行标准化处理。接着,讲解了基础热图的绘制方法,包括是否进行聚类、调整聚类方法与距离度量等。进阶部分则涉及颜色与单元格的调整、添加行列注释、热图分面与数值显示等技巧。最后,提供了一个完整的代码示例,帮助读者快速掌握热图绘制的各项功能。通过本文,即
·
热图(Heatmap)是数据分析和可视化中展示矩阵数据分布的核心工具,适用于基因表达分析、市场数据对比等场景。本文将基于 R语言的pheatmap包,详细讲解热图绘制全流程,涵盖 基础绘图、参数调优、注释添加、聚类分面 等技巧,并提供 逐行代码注释,新手也能快速掌握。
一、环境准备与数据生成
1.1 安装与加载包
# 安装pheatmap包(首次运行需执行)install.packages("pheatmap")# 加载包library(pheatmap)# 生成模拟数据(20行基因 × 10列样本)set.seed(123) # 固定随机数,确保结果可重复expr_matrix <- matrix(rnorm(200), nrow = 20, ncol = 10)rownames(expr_matrix) <- paste0("Gene", 1:20) # 行名为基因编号colnames(expr_matrix) <- paste0("Sample", 1:10) # 列名为样本编号# 查看数据前3行head(expr_matrix[, 1:3])
1.2 数据标准化
# 按行标准化(Z-score归一化,突出基因在不同样本中的表达差异)expr_scaled <- t(scale(t(expr_matrix)))# 按列标准化:scale(expr_matrix, center=TRUE, scale=TRUE)
二、基础热图绘制
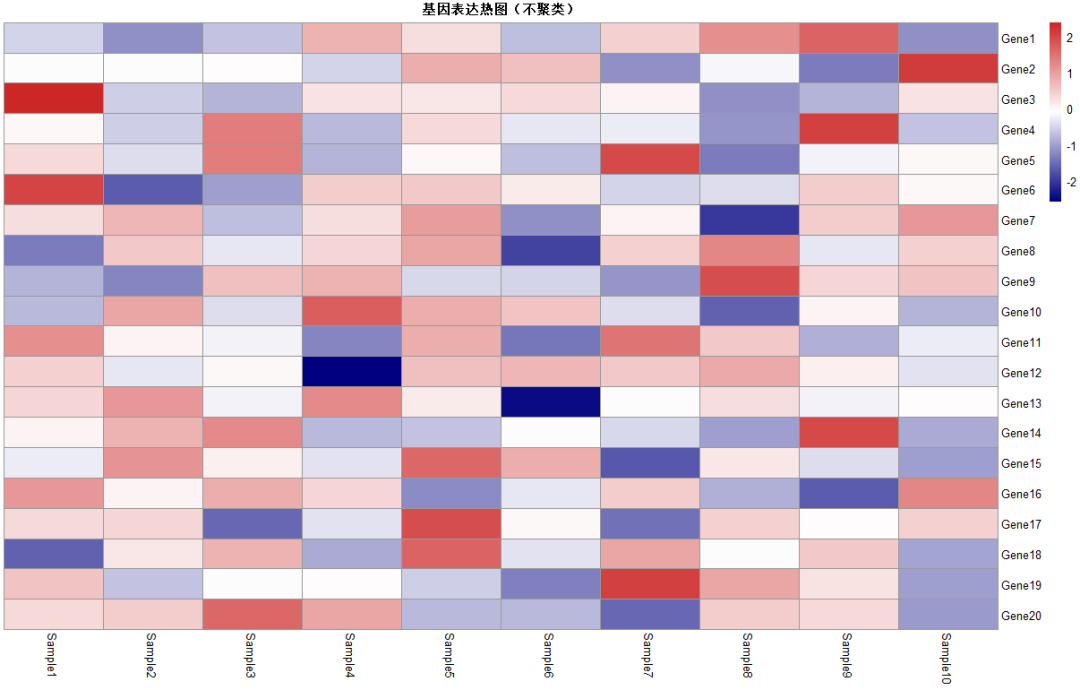
2.1 基础绘图(不聚类)
pheatmap(expr_scaled,main = "基因表达热图(默认参数)",cluster_rows = FALSE, #是否对行进行聚类,默认TRUEcluster_cols = FALSE, #是否对列进行聚类,默认TRUEcolor = colorRampPalette(c("navy", "white", "firebrick3"))(100))
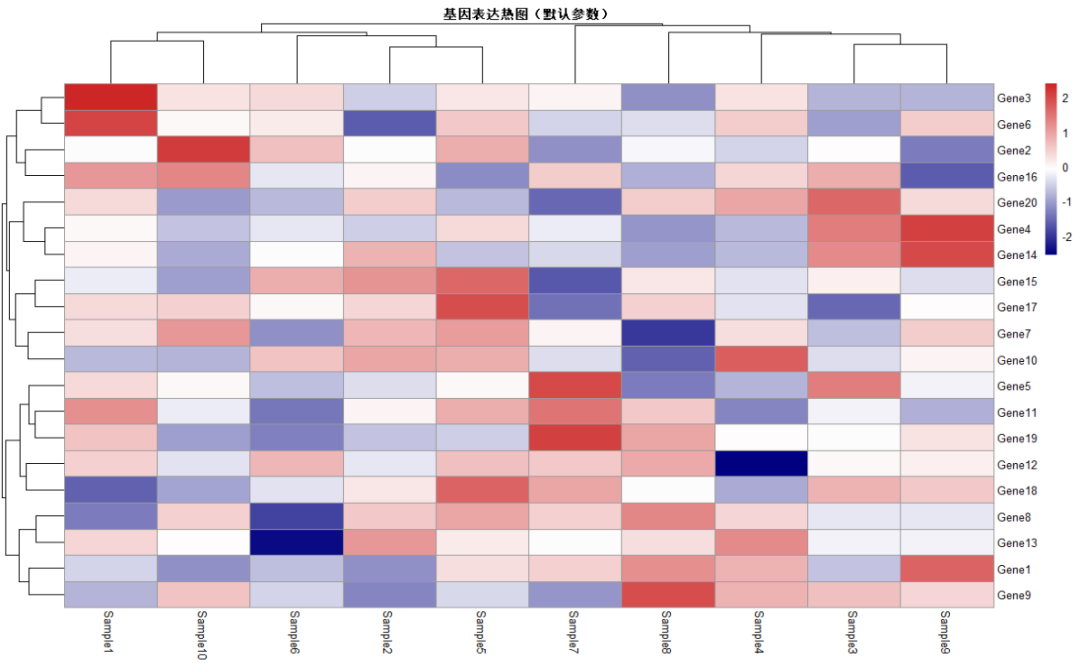
2.2基础绘图(默认聚类)
pheatmap(expr_scaled,main = "基因表达热图(默认参数)",color = colorRampPalette(c("navy", "white", "firebrick3"))(100))
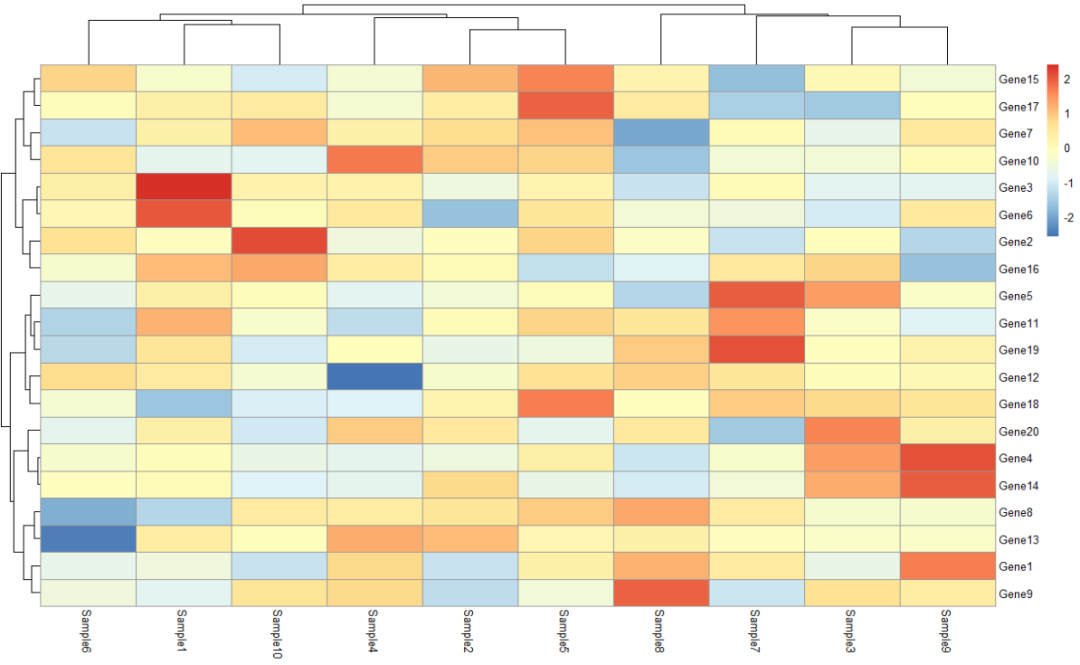
2.3 调整聚类方法与距离度量
pheatmap(expr_scaled,clustering_method = "ward.D2", # 聚类方法改为Ward法clustering_distance_rows = "correlation", # 行聚类用Pearson相关系数treeheight_row = 30, # 行聚类树高度treeheight_col = 50) # 列聚类树高度
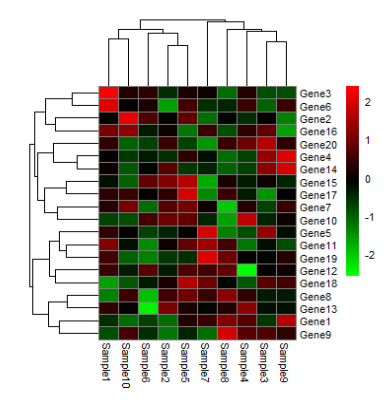
三、进阶功能与参数调优
3.1 颜色与单元格调整
pheatmap(expr_scaled,color = colorRampPalette(c("green", "black", "red"))(50), # 绿-黑-红渐变cellwidth = 15, # 单元格宽度cellheight = 10, # 单元格高度fontsize = 8, # 字体大小border_color = "grey50", # 单元格边框颜色na_col = "white") # 缺失值颜色
3.2 添加行列注释
# 创建列注释(样本分组:前5列为对照组,后5列为实验组)annotation_col <- data.frame(Group = factor(rep(c("Control", "Treatment"), each = 5)),row.names = colnames(expr_scaled))# 创建行注释(基因类型:随机生成)annotation_row <- data.frame(Type = sample(c("PathwayA", "PathwayB"), 20, replace = TRUE),row.names = rownames(expr_scaled))# 自定义注释颜色ann_colors <- list(Group = c(Control = "#1B9E77", Treatment = "#D95F02"), # 分组颜色Type = c(PathwayA = "#7570B3", PathwayB = "#E7298A") # 基因类型颜色)# 绘制带注释的热图pheatmap(expr_scaled,annotation_col = annotation_col,annotation_row = annotation_row,annotation_colors = ann_colors,angle_col = 45) # 列标签旋转45度
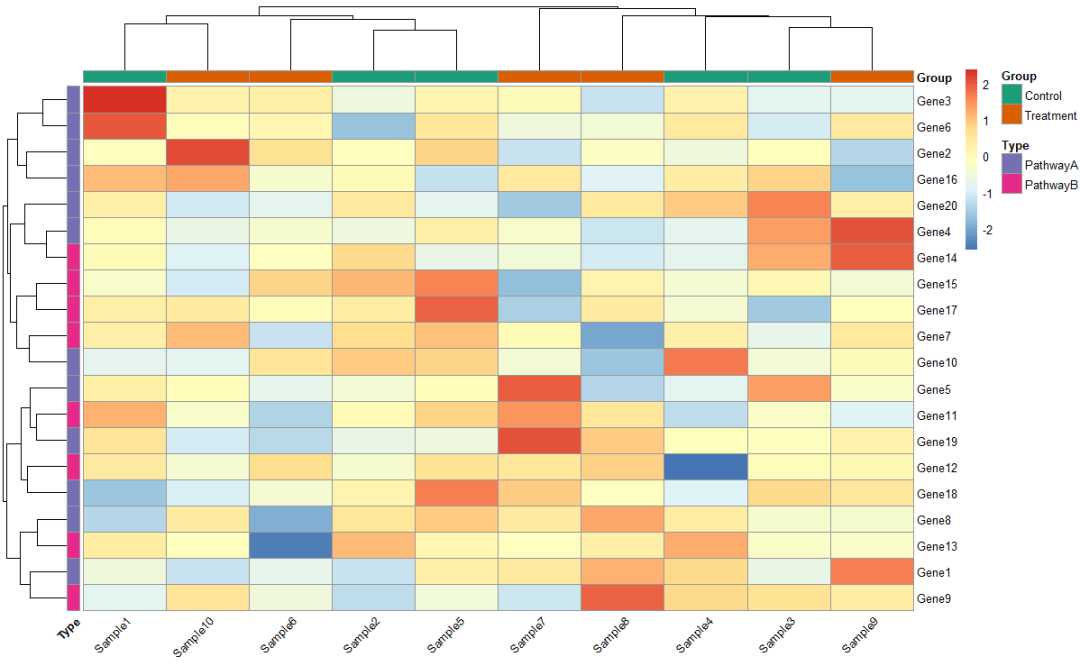
3.3 热图分面与数值显示
# 按行聚类结果分3组,按列分2组pheatmap(expr_scaled,cutree_rows = 3, # 行分3簇cutree_cols = 2, # 列分2簇gaps_row = c(5, 10), # 手动添加行分界线display_numbers = TRUE, # 显示单元格数值number_format = "%.2f", # 保留两位小数number_color = "black")
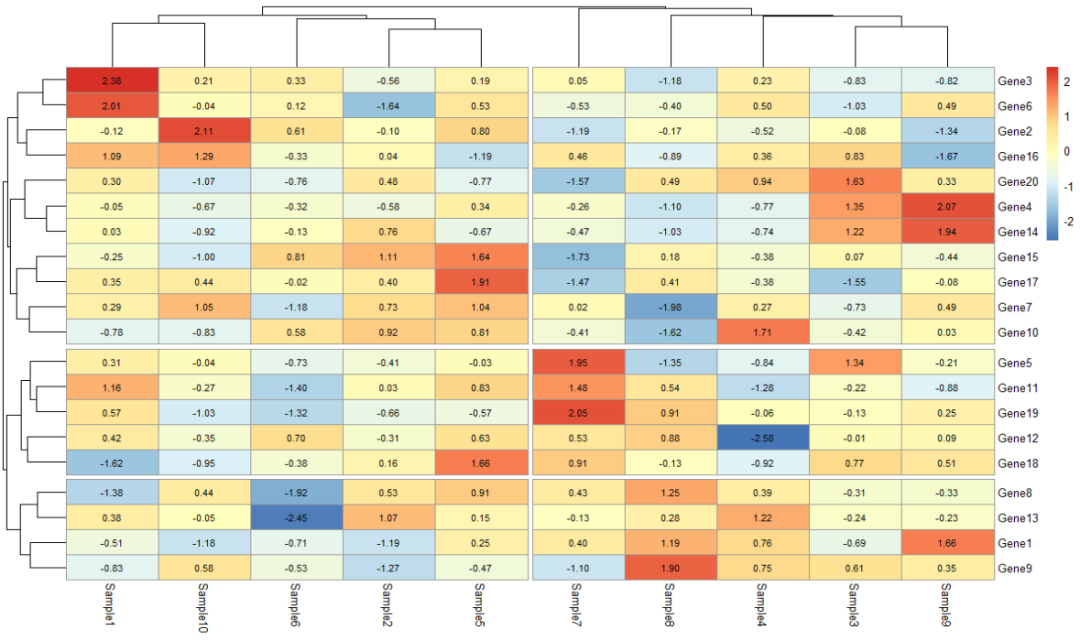
四、完整代码示例
# 加载包与数据生成library(pheatmap)set.seed(123)expr_matrix <- matrix(rnorm(200), 20, 10)rownames(expr_matrix) <- paste0("Gene", 1:20)colnames(expr_matrix) <- paste0("Sample", 1:10)expr_scaled <- t(scale(t(expr_matrix))) # 按行标准化# 创建注释与颜色annotation_col <- data.frame(Group = factor(rep(c("Control", "Treatment"), each = 5)),row.names = colnames(expr_scaled))ann_colors <- list(Group = c(Control = "#1B9E77", Treatment = "#D95F02"))# 绘制热图pheatmap(expr_scaled,color = colorRampPalette(c("navy", "white", "red"))(100),annotation_col = annotation_col,annotation_colors = ann_colors,cluster_rows = TRUE,cluster_cols = TRUE,main = "基因表达热图(完整示例)",fontsize_row = 8,cellwidth = 15,cellheight = 12)
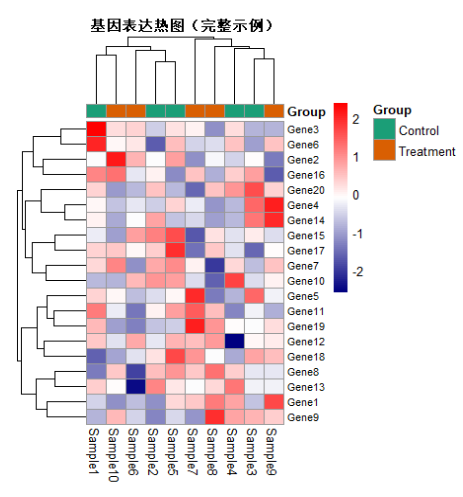
更多精彩内容,欢迎查看:
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容







所有评论(0)